Luecken MD, Theis FJ. Current best practices in single-cell RNA-seq analysis: a tutorial. Mol. Syst. Biol. 2019, 15: e8746.
摘要
single cell RNA-seq 提高了基因表达研究的分辨率,这项技术也带来越来越多的单细胞分析方法。这使得研究者难以驾驭这一多工具格局并从中搭建最新的工作流程来分析自己的数据。在这里,我们详细介绍了典型的单细胞 RNA-seq 数据分析步骤,包括预处理(质量控制、标准化、数据校正、特征选择和降维)以及细胞及基因水平的下游分析。我们根据独立比较研究为这些步骤制定了当前(2019年)最佳实践建议。我们已将这些最佳实践建议整合到工作流中,并将其应用于公共数据集,以进一步说明这些步骤在实践中如何工作。我们的案例研究可参见https://www.github.com/theislab/single-cell-tutorial。这篇综述将作为单细胞新手进入该领域的数据分析流程指南,并帮助现有的研究人员更新他们的分析流程。
关键词:分析流程开发;计算生物学;数据分析教程;单细胞 RNA-seq
概述
近年来,单细胞 RNA 测序 (scRNA-seq) 推进了我们对生物系统的认识。我们已经能够研究斑马鱼、青蛙和涡虫的细胞异质性 (Briggs et al,2018;Plass et al,2018;Wagner et al,2018),并发现之前被掩盖的细胞群 (Montoro et al,2018;Plasschaert et al,2018)。该技术的巨大潜力促使计算生物学家开发一系列分析工具 (Rostom et al,2017)。尽管该领域正在努力确保单个工具的可用性,但单细胞数据分析中,新手的一个进入障碍( a barrier of entry)是由于该领域相对不成熟而缺乏标准。在本文中,我们简述目前scRNA-seq 分析的最佳做法,为今后的分析标准化奠定基础。
标准化面临的挑战包括分析方法不断增加(截至 2019 年 3 月 7 日已达 385多种工具)和数据集规模爆炸性增长 (Angerer et al,2017;Zappia et al,2018)。我们正在不断寻找新的方法来使用我们所测得的数据。例如,最近的工具可预测分化中的细胞命运 (La Manno et al,2018)。分析工具的不断改进有利于产生新的科学洞察力,但这也使标准化更加复杂。
标准化的第二个挑战在于技术方面。scRNA-seq 数据的分析工具用各种编程语言,最突出的是 R 和 Python (Zappia et al,2018)。尽管跨环境的工具正在增长(预印:Scholz et al,2018),但编程语言的选择通常也是分析工具之间的一种选择。Seurat (Butler et al,2018)、Scater (McCarthy et al,2017) 或 Scanpy (Wolf et al,2018) 等热门平台提供了开发流程的集成环境,且包含大型分析工具。然而,这些平台仅限于使用各自编程语言开发的工具。通过扩展,语言限制也适用于目前可用的 scRNA-seq 分析教程,其中许多教程围绕上述平台(R 和 bioconductor 工具:https://github.com/drisso/bioc2016singlecell和https://hemberg-lab.github.io/scRNA.seq.Lun 等人,2016b;Seurat;scanpy.
考虑到上述挑战,我们并没有标准化分析流程,而是概述了当前的最佳实践和独立于编程语言的通用工具。我们指导读者完成 scRNA-seq 分析流程的各个步骤(图 1),介绍当前的最佳实践,并讨论分析陷阱提出开放性问题。由于工具的新颖性和缺乏比较,事实上无法确定最佳实践,因此我们列出了流行的可用工具。所概述的步骤从reads或计数矩阵开始,得出潜在分析终点,Lun et al (2016b) 涵盖了早期预处理步骤。整合现有最佳实践的详细案例研究可从我们的 github 获得,网址为:https://github.com/theislab/single-cell-tutorial/。在这里,我们在一个实际的示例工作流中应用了当前的最佳实践来分析公共数据集。分析工作流程用rpy2在 Jupyter-Ipython notebook中集成了 R 和 Python 工具。有了可用的文档,它很容易作为工作流模板进行二次修改。
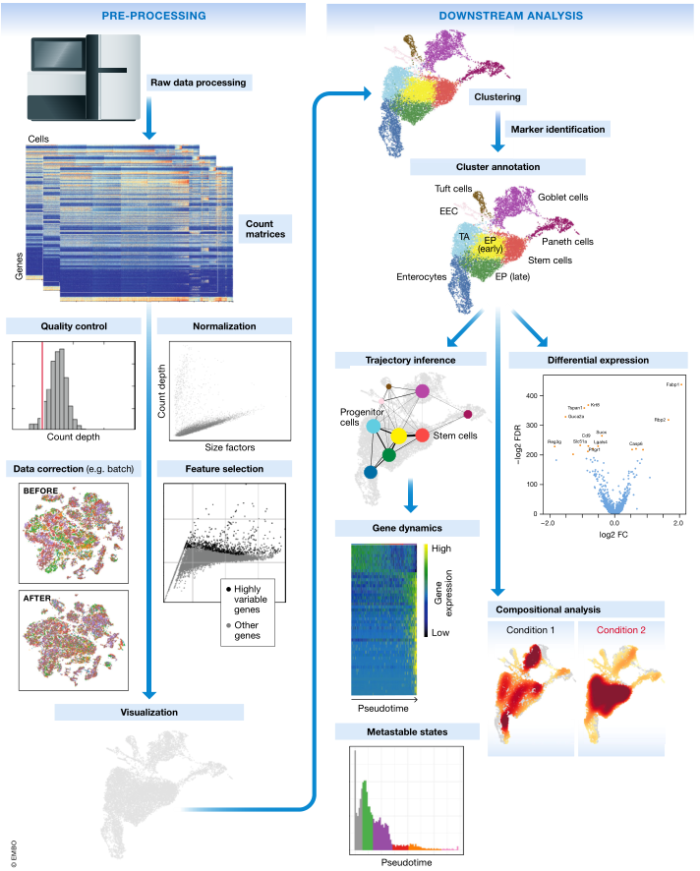
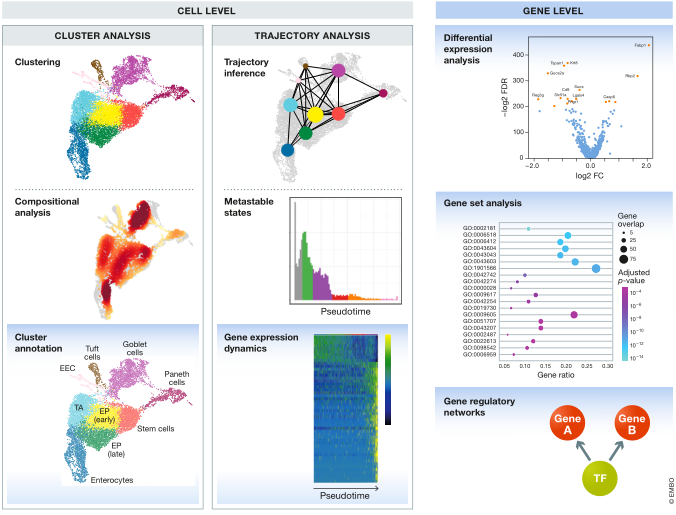
图 1. 典型的单细胞 RNA-seq 分析工作流程示意图。原始测序数据经过处理和比对,得到计数矩阵,代表工作流程的开始。计数矩阵经过预处理和下游分析。使用 Haber et al (2017) 肠上皮细胞数据的最佳实践工作流程生成子图。
框1:实验性scRNA-seq工作流的关键元素
从生物样本到可分析的单细胞数据需要经过多个步骤。典型的工作流程包括:单细胞解离、单细胞分离、文库构建和测序。对这些阶段的简要概述如下:单细胞实验的起始材料通常以生物组织样本的形式获得。
单细胞悬浮液的制备作为第一步,是在一个被称为单细胞解离的过程中产生的,其中组织被消化。为分析每个细胞中的 mRNA,必须分离细胞。单细胞分离根据实验方案的不同而不同。虽然基于平板的技术将细胞隔离到平板上的孔中,但基于液滴的方法依赖于在自己的微流体液滴中捕获每个细胞。在这两种情况下,都可能发生错误,导致多个细胞被捕获在一起(doublets or multiplets)、非活细胞被捕获或完全没有细胞被捕获(空液滴/孔)形成空滴的情况尤其常见,因为基于液滴的方法依靠低浓度的输入细胞流动来控制双联体率。每孔或液滴中都含有分解细胞膜和进行文库构建所必需的化学物质。胞内 mRNA 被捕获、反转录为 cDNA 分子并扩增的过程称为文库构建。当细胞隔离进行这一过程时,每个细胞的 mRNA 可以被一个孔或滴特定的细胞条形码标记。此外,许多实验方案也用唯一分子标识符 (UMI) 标记捕获的分子。测序前扩增细胞 cDNA,以增加其被测量的概率。UMIs 允许我们区分相同 mRNA 分子的扩增拷贝和从相同基因转录的不同 mRNA 分子的reads。
构建好文库后,使用细胞条形码进行标记,并根据协议进行UMIs标记。这些库汇集在一起(multiplexed)用于测序。序列产生reads数据,这些数据经过质量控制,再准备阶段根据指定的条形码(demultiplexing)和reads比对区分细胞。对于基于umi的协议,reads数据可以被进一步解复用以产生捕获的mRNA分子计数(count data)。
Pre-processing and visualization
对测序仪生成的原始数据进行处理,以获得分子计数(count 矩阵)或读数(reads矩阵)的矩阵,这取决于是否在单细胞文库构建方案中纳入了独特的分子标识符 ( unique molecular
identifiers ,UMI)(有关分析前的实验步骤概述,请参见框 1)。Cell Ranger (Zheng et al,2017)、indrops (Klein et al,2015)、SEQC (Azizi et al,2018) 或 zUMIs (Parekh et al,2018) 等原始数据处理流程负责reads质量控制 (QC),为其细胞barcode和 mRNA 来源分子(也称为解复用,demultiplexing)分配reads、基因组比对和定量。得到的reads或计数矩阵包含barcode x 转录本数量的高纬数据。此处使用术语barcode代替细胞,因为所有reads均为分配给相同的barcode可能与来自同一细胞的reads不一致。一个barcode可能错误地标记多个细胞(双联体)或可能不标记任何细胞(空滴/孔)。虽然reads和计数数据的测量噪声水平不同,但典型分析流程中的处理步骤相同。为了简单起见,我们将在本教程中将这些数据称为count矩阵。如果reads和count矩阵的结果不同,则专门指出reads矩阵。
Quality control
在分析单细胞基因表达数据之前,我们必须确保所有的细胞barcode数据都对应于活细胞。细胞 QC 通常基于三个 QC 变量进行:
- 每个barcode的计数数量(count depth )
- 每个barcode的基因数量
- 每个barcode的线粒体基因计数分数 (Ilicic et al,2016;Griffiths et al,2018)
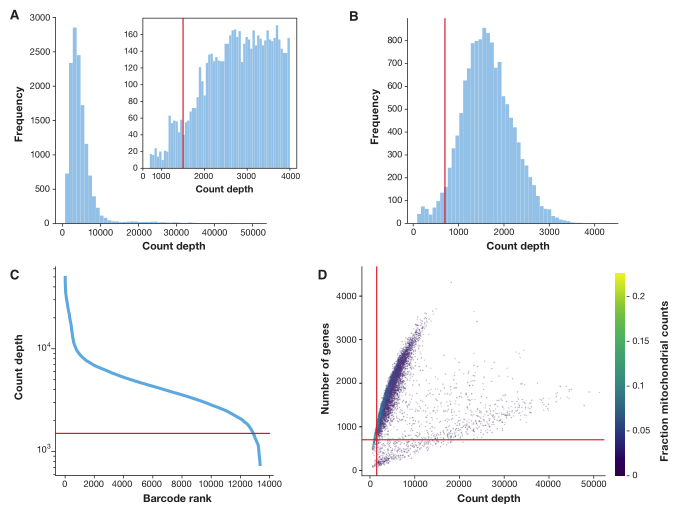
检查这些 QC 变量的分布,以确定是否存在通过阈值处理过滤掉的离群峰(图 2)。这些异常barcode可能对应于死细胞、膜破损的细胞或双联体。例如,低计数深度的barcode、很少检测到的基因以及线粒体计数的高分数都表明细胞的细胞质 mRNA 已经通过破损的膜漏出,只有位于线粒体中的 mRNA 仍然是保守的(图 2)。与之相反,非预期高计数和大量检测基因的细胞可能代表双联体。因此,高计数深度阈值常用于过滤掉潜在的双峰。最近的三种双联检测工具提供了更优雅和可能更好的解决方案 (DoubletDecon:preprint:DePasquale et al,2018;Scrublet:Wolock et al,2019;doublet Finder:McGinnis et al,2018)。
图 2. Haber et al (2017) 的小鼠肠上皮数据集过滤决策的质量控制指标图。(A) 每个cell的计数深度直方图。较小的直方图在计数深度低于 4,000 时放大。根据在约 1,200 个计数处检测到的峰值,此处应用的阈值为 1,500。(B) 每个细胞检测到的基因数的直方图。在大约 400 个基因处可见一个小的噪声峰。这些细胞使用描述的阈值(红线)700 个基因过滤掉。计数深度分布从高到低计数深度。该可视化与 Cell Ranger 输出中显示的 logClog 图相关,该输出用于过滤空液滴。它显示了一个肘部的计数深度开始迅速减少约 1500 计数。(D) 通过线粒体读数部分染色的基因数量与计数深度的关系。线粒体读取片段仅在检测基因很少的特别低计数细胞中高。这些细胞被我们的计数和基因数阈值过滤掉。联合可视化计数和基因阈值显示联合过滤效果,表明较低的基因阈值可能已经足够
单独考虑这三个细胞 QC 变量中的任何一个都可能导致对细胞信号的误解。例如,具有较高线粒体计数的细胞可能参与呼吸过程。同样,其他 QC 变量也有生物学解释。低count和(或)基因的细胞可对应静止细胞群,高count的细胞体积可能更大。事实上,细胞之间的分子计数可能存在强烈差异(参见项目 github 的案例研究)。因此,当单变量阈值决策时,应联合考虑细胞 QC 变量(图 2D),这些阈值应尽可能设置为允许的,以避免无意中过滤掉活细胞群。考虑到多变量细胞 QC 的依赖性,筛选模型可能提供更敏感的 QC 选项。
含有异质混合细胞类型的数据集可能显示多个 细胞QC 变量峰值。例如,图 2D 显示了具有不同 QC 分布的两个细胞群。如果之前没有进行过滤步骤(注意 Cell Ranger 也进行细胞 QC),那么只有每个barcode峰的最低计数深度和基因应该被认为是非活细胞。进一步的阈值指导原则是使用所选阈值过滤掉的细胞比例。对于高计数过滤,该比例不应超过预期的双联率。
除了检查细胞的完整性,细胞 QC 步骤也必须在转录本水平上进行。原始计数基质通常超过 20,000 个基因。通过过滤掉在少数细胞中不表达的基因,可以大幅减少这一数量。设置此阈值的一个准则是使用最小cell群,并留下一些dropout 效应(dropout effects. )的余地。例如,筛选出少于 20 个细胞中表达的基因可能会使检测少于 20 个细胞的细胞团变得困难。对于高脱落(dropout )率的数据集,这个阈值也可能使较大簇的检测复杂化。阈值的选择应根据数据集中的细胞数量和预期的下游分析进行调整。
可直接对计数数据进行进一步 QC。Ambient gene expression(环境基因表达)指不是来自barcode细胞,而是来自其他溶解细胞的count,这些细胞的 mRNA 在文库构建之前污染了细胞悬液。这些增加的环境计数会扭曲下游分析,如标记基因鉴定或其他差异表达检测,尤其是当样本之间的水平变化时。在基于液滴的 scRNA-seq 数据集中校正这些影响是可能的,由于大量的空液滴,可用于模拟环境RNA表达谱。最近开发的SoupX(预印本:Young &
使用这种方法直接纠正计数数据。在下游分析中忽视强环境基因的实用方法也被用来解决这个问题(Ange- lidis et al, 2019)。
进行质量控制以确保数据质量足以用于下游分析。由于无法先验确定足够的数据质量,因此根据下游分析性能(例如,聚类注释)进行判断。在分析数据时,可能需要多次重新审查质量控制参数。通常,从允许的质控阈值开始,在执行更严格的质控之前研究这些阈值的影响是有益的。这种方法对于包含异质性细胞群的数据集特别重要,其中细胞类型或状态可能被错误解释为低质量离群细胞。在低质量数据集中,严格的 QC 阈值可能是必要的。可通过试验 QC 指标确定数据集的质量(见附录补充文本 S2,卑微小王手头并没有补充文档,从略)。在这种迭代 QC 优化中,应该注意数据窥视(data peeking.)。不应调整 QC 阈值以改善统计检验的结果。相反,可根据数据集可视化和聚类中的 QC 变量分布来评价 QC效用。
问题和建议:
•通过基因数量、计数深度和线粒体reads分数的异常峰来执行细胞QC。考虑这些共同的影响而不是单独的考虑它们。
•尽可能地容忍QC阈值化,如果下游聚类无法解释,则重新QC。
•如果QC变量在样品之间的分布不同,则应针对每个样品分别QC,以解释样品质量差异,如Plasschaert等(2018)。
Normalization
计数矩阵中的每个计数代表细胞 mRNA 分子的成功捕获、逆转录和测序(框 1)。由于每个步骤固有的变异性,相同细胞的计数深度结果却可能不同。因此,当基于计数数据比较细胞间的基因表达时,任何差异可能仅由采样效应( sampling effects.)引起。通过例如缩放( sampling effects)计数数据以获得正确的细胞间相对基因表达丰度来解决这一问题。
bulk RNA数据已有许多标准化方法 (preprint:Pachter,2011;Dillies et al,2013)。虽然其中一些方法已应用于 scRNA-seq 分析,但单细胞数据特有的变异来源如技术脱落(technical dropouts )(取样导致的零计数,双零问题)促使开发出了针对 scRNA-seq 的标准化方法 (Lun et al,2016a;Vallejos et al,2017)。
最常用的规范化协议是 count depth scaling,也称为每百万计数或 CPM 规范化。该方案来自bulk 表达分析,并使用与每个细胞计数深度成比例的所谓大小因子对计数数据进行标准化。该方法的变体使用不同的因子或数据集中每个细胞的中位计数深度缩放。CPM 标准化假设数据集中的所有细胞最初包含相同数量的 mRNA 分子,计数深度差异仅由于取样产生。该假设与下采样(downsampling)方案相同,下采样方案是从数据中随机取样读取或计数,使所有细胞的计数预先规定的数量或更少。在下采样丢掉数据的同时,也增加了技术脱落率,而 CPM 和其他全局缩放标准化方法则没有。因此,下采样可以提供类似计数深度下细胞表达谱的更真实表示。
由于单细胞数据集通常由具有不同大小和分子计数的异质细胞群组成,因此更复杂的标准化方法通常是合适的。例如,Weinreb et al (2018) 使用了 CPM 的简单延伸,计算它们的大小因子时,排除了任何细胞中占总计数至少 5% 的基因。这种方法考虑到了少数高表达基因的分子计数变异性。基于 Scran 合并的尺寸因素估计(pooling-based size factor estimation)方法允许更多的细胞异质性 (Lun et al,2016a)。细胞合并后,根据基因的线性回归估算大小因子,以避免技术脱落效应。该方法将变异性限制在细胞间差异表达基因的 50% 以下,并且在独立比较中始终是性能最佳的标准化方法。经证明,Scran 在批次校正( batch correction) (Buttner et al,2019) 和差异表达分析 (preprint:vith et al,2019) 方面的性能优于其他检测的归一化方法。在与原作者的小规模比较中,该方法也显示出稳健的尺寸因子估计值 (Vallejos et al,2017)。
CPM、高计数过滤 CPM 和 scran 使用线性、全局缩放标准化计数数据。还存在非线性归一化方法,可解释更复杂的异质性(Cole et al,2019)。许多方法涉及到计数资料的参数化建模。例如,Mayer et al (2018) 使用技术变量(如测序深度和每个基因的计数数量)拟合负二项模型,以拟合模型参数。模型拟合的残差作为基因表达的标准化定量。这种方法可以将技术和生物数据校正(例如批次校正或细胞周期效应校正)与计数深度归一化相结合。已证明非线性归一化方法优于全局缩放方法,尤其是在具有强批次效应的情况下 (Cole et al,2019)。因此,非线性归一化方法对于基于平板的 scRNA-seq 数据尤其相关,这些数据往往在平板之间存在批次效应。此外,与基于液滴的数据相比,基于平板的数据可显示每个细胞计数深度的较大变化 (Svensson et al,2017)。虽然非线性归一化方法或替代方法(例如下采样)似乎更适合这些条件,但需要进行比较研究来确认该假设。在本教程中,我们倾向于将标准化和数据校正(批次校正、噪声校正等)步骤分开,以强调数据的不同处理阶段(参见预处理数据部分的阶段)。因此,我们着重研究全局尺度归一化方法(global scaling normalization)。
我们不能期望单一的标准化方法适合所有类型的 scRNA-seq 数据。例如,vith et al (2017) 表明reads和计数数据可通过不同模型进行最佳拟合。事实上,Cole et al (2019) 发现没有一种归一化方法对不同的数据集表现都是最佳的,并认为应使用其 scone 工具为特定数据集选择适当的归一化方法。此外,scRNA-seq 技术可分为全长和 30 种富集方法 (Svensson et al,2017;Ziegenhain et al,2017)。来自全长方案的数据可能受益于考虑到基因长度的标准化方法(例如 Patel et al,2014;Kowalczyk et al,2015;Soneson)
细胞计数数据可以归一化,使细胞间具有可比性,同样,基因计数也可以按比例调整,以改善基因间的比较。基因归一化构成基因计数的标度,使其均值和单位变异(z值)为零。这种比例的变化影响了所有的基因下游分析的权重。是否对基因进行归一化目前尚无共识。虽然流行的Seurat教程(Butler et al, 2018)通常应用基因缩放(scaling,),但Slingshot方法的作者在他们的教程中选择不缩放基因(Street et al, 2018)。这两种选择之间的偏好围绕着是否所有的基因都应该在下游分析中得到同等的权重,或者一个基因的表达量是否代表了该基因的重要性。为了尽可能多地保留数据中的生物信息,我们选择在本教程中避免对基因进行筛选。
归一化后,数据矩阵通常是对数 (+ 1) 转换的。这种转变有三个重要作用。
- 首先,对数转换的表达值之间的距离代表对数倍数变化,这是衡量表达变化的经典方式。
- 其次,对数转换减轻(但不消除)单细胞数据中的均值方差关系 (Brennecke et al,2013)。
- 最后,对数转换降低了数据的偏斜度,以适用于假设数据呈正态分布的下游分析工具。
虽然 scRNA-seq 数据实际上不是对数正态分布 (Vieth et al,2017),但这三种效应使对数转换成为一种粗糙但有用的工具。差异表达检测 (Finak et al,2015;Ritchie et al,2015) 或批次校正 (Johnson et al,2006;Buttner et al,2019) 的下游应用强调了这种有用性,这些应用将对数转换用于这些目的。但是,应该注意的是,归一化数据的对数转换可在数据中引入虚假差异表达效应(预印:Lun,2018)。当归一化大小因子分布在试验组之间存在强烈差异时,该效应尤其明显。
问题和建议:
•我们建议使用scran对非全长数据集进行标准化。
另一种方法是通过scone评估基于平台的数据集的标准化方法。全长scRNA-seq协议可以使用bulk 方法修正基因长度。
•对于将基因的均值和单位方差缩放到0没有共识。
我们宁愿不缩放基因表达(We prefer not to scale gene expression.)。
•规范化的数据应该是log(x+1)-转换后用于假设数据是正态分布的下游分析方法。
Data correction and integration
如上所述的标准化试图消除计数采样的影响。但是,归一化数据仍可能包含不希望的变异性。数据进一步的校正针对技术和生物学变量,如批次、脱落或细胞周期效应。这些变量并不总是进行校正。相反,决定考虑哪些变量将取决于预期的下游分析。我们建议分别考虑生物和技术变量的校正,因为这些变量用于不同的目的,并且存在独特的挑战。
Regressing out biological effects
校正技术变量(covariates)对于揭示潜在生物信号至关重要,校正生物变量对于挑选出关注的特定生物信号更加重要。最常见的生物数据校正是去除细胞周期对转录组的影响。该数据校正可通过 Scanpy 和 Seurat 平台 (Butler et al,2018;Wolf et al,2018) 或具有更复杂混合模型(如 scLVM (Buettner et al,2015) 或 fscLVM (Buettner et al,2017))的专门包装中实施的细胞周期评分的简单线性回归进行。用于计算细胞周期评分的标记基因列表来自文献 (Macosko et al,2015)。这些方法也可用于回归其他已知的生物学效应如线粒体基因表达,其被解释为细胞应激的指征。
在校正生物学效应数据之前,应考虑几个方面。
- 首先,校正生物学变量并不总是有助于解读 scRNA-seq 数据。虽然去除细胞周期效应可改善发育轨迹的推断 (Buettner et al,2015;Vento-Tormo et al,2018),但细胞周期信号也可提供生物学信息。例如,可根据细胞周期评分确定增殖细胞群(参见 github 项目的个案研究)。
- 生物信号必须在语境中理解。鉴于生物过程发生在同一生物体内,这些过程之间存在依赖性。因此,纠正一个过程可能无意中掩盖另一个过程的信号。
- 最后,有人认为,细胞大小的变化解释了通常归因于细胞周期的转录组效应 (McDavid et al,2016)。因此,通过标准化校正细胞大小,或专用工具如 cgCorrect (Blasi et al,2017),也部分校正了 scRNA-seq 数据中的细胞周期影响。
Regressing out technical effects
用于回归生物学变量的回归模型变量也可应用于技术变量。单细胞数据中最显著的技术变量是计数深度和批次。尽管标准化比例计数数据使细胞之间的基因计数相当,但计数深度效应通常保留在数据中。这种计数深度效应既可以是生物的,也可以是技术的。例如,细胞可能大小不同,因此 mRNA 分子计数也不同。然而,归一化后的技术计数效应可能仍然存在,因为没有缩放方法可以推断由于采样不佳而未检测到的基因的表达值。回归出计数深度效应可以提高轨迹推理算法的性能,它依赖于找到cell之间的转换(参见 project github 的案例研究)。当校正多个变量(例如,细胞周期和计数深度)时,应在一个步骤中对所有变量进行回归,以考虑变量之间的依赖性。
另一种基于回归的消除计数影响的策略是使用更严格的标准化过程,如下采样或非线性标准化方法(参见标准化部分)。这些方法可能特别适用于基于平板(plate-based )的 scRNA-seq 数据集,其中每个细胞计数深度的较大变化可以掩盖细胞之间的异质性。
Batch effects and data integration
当细胞以不同的分组处理时,可能发生批次效应。批次效应可以由不同芯片上的细胞、不同测序泳道中的细胞或不同时间收获的细胞组成。细胞经历的不同环境会对转录组的测量或转录组本身产生影响。产生的影响存在于多个层面:实验中的细胞组之间、在同一实验室进行的实验之间或来自不同实验室的数据集之间。
在这里,我们区分前两个和后两个场景。
- 在相同的实验中校正样品或细胞之间的批次效应是经典的来自 bulk RNA-seq 的批次校正(Batch effects)。
- 我们将这与多次实验的数据整合区分开来,我们称之为数据整合(data integration)。虽然批效应通常使用线性方法校正,但一般使用非线性方法进行数据整合。
最近对经典批次校正方法的比较显示,ComBat (Johnson et al,2006) 在低至中等复杂度的单细胞实验中也表现良好 (Buttner et al,2019)。ComBat 由基因表达的线性模型组成,其中在数据的平均值和方差中均考虑了批次贡献(图 3)。不考虑计算方法,**批量校正的最佳方法是通过巧妙的实验设计预先消除影响并完全避免影响 **(Hicks et al,2017)。通过合并实验条件和样品中的细胞,可避免批次效应。使用诸如细胞标记 (preprint:Gehring et al,2018) 或通过遗传变异 (Kang et al,2018) 等策略,可能分离实验中合并的细胞。
与批次校正相比,面临的另一个挑战是整合不同的数据集。估计批效应时,ComBat 使用一批中的所有细胞来拟合批次参数。这种方法将混淆批处理效应与细胞类型或数据集之间不相同状态之间的生物学差异。为克服该问题,开发了典型相关分析 (CCA;Butler et al,2018)、相互最近邻 (MNN;Haghverdi et al,2018)、Scanorama(预印:Hie et al,2018)、RISC(预印:Liu et al,2018)、scGen(预印:Lotfollahi et al,2018)、LIGER(预印:Welch et al,2018)、BBKNN(预打印:Park et al,2018)和 Harmony(预打印:Korsunsky et al,2018)等数据整合方法。数据整合方法虽然也可以应用于简单的批次校正问题,但考虑到非线性数据集成方法的自由度增加,我们建议警惕过度修正。例如,在更简单的批次校正设置中,MNN 的表现优于 ComBat (Buttner et al,2019)。需要对数据整合和批次校正方法进行进一步比较研究,以评估这些方法的应用范围。
Expression recovery
另一种类型的技术数据校正是表达恢复(expression recovery)(也就是去噪或插补)。单细胞转录组的测量包含各种噪声 (Gru net al,2014;Kharchenko et al,2014;Hicks et al,2017)。这种噪音的一个特别突出的方面是dropout。推断dropout事件,用合适的表达值替换这些零,减少数据集中的噪声,一直是几个最新工具(MAGIC:van Dijk et al,2018;DCA:Eraslan et al,2019;scVI:Lopez et al,2018;SAVER:Huang et al,2018;scImpute:Li& Li, 2018).已证明执行表达恢复可改善基因相关性估计 (van Dijk et al,2018;Eraslan et al,2019)。此外,该步骤可与归一化、批次校正和 scVI 工具中实施的其他下游分析整合 (Lopez et al,2018)。虽然大多数数据校正方法以归一化数据作为输入,但一些表达式恢复方法是基于预期负二项分布噪声,因此在原始计数数据上运行。应用表达恢复时,应考虑到没有一种方法是完美的。因此,任何方法均可能导致数据中的噪声过度校正或校正不足。事实上,表达恢复的结果报告了假相关信号(Andrews & Hemberg, 2018).考虑到在实际应用中评估成功的表达恢复的难度,这个场景对考虑是否消噪的用户来说是一个挑战。此外,对于当前可用的表达式恢复方法,大型数据集的可伸缩性仍然是一个问题。鉴于这些考虑,目前对于如何使用消噪数据尚未达成共识(见处理数据章节的阶段)。谨慎的方法是仅将表达恢复用于数据的直观显示,而不是在探索性数据分析过程中应用之。这里彻底的实验验证尤为重要。
问题和建议:
•回归出(Regress out )生物变量只是为了轨迹推断,其他生物过程被没有回归出的生物协变量所掩盖。
•同时考虑回归技术和生物变量,而不是针对某项回归。
•基于平台的数据预处理可能需要回归计数,通过非线性归一化方法进行归一化或向下采样。
•当cell类型和批次之间的状态成分一致时,我们建议通过ComBat进行批次校正
•数据整合和批次校正应通过不同方法进行。数据集成工具可能过度纠正简单的批处理效应。
•用户应谨慎对待仅在表达恢复后发现的信号。最好不使用该步骤进行探索性分析。
Feature selection, dimensionality reduction and visualization
一个人类的单细胞 RNA-seq 数据集可以包含多达 25,000 个基因的表达值。这些基因中的许多基因对于给定的 scRNA-seq 数据集不会提供有价值的信息,许多基因将大部分包含零计数。即使在 QC 步骤中过滤掉这些零计数基因后,单细胞数据集的特征空间也可以有超过 15000 个维度。为了减轻下游分析工具的计算负担,减少数据中的噪声,并使数据可视化,可以使用几种方法降低数据集的维数。
Feature selection
scRNA-seq 数据集降维的第一步通常是特征选择。在此步骤中,将筛选数据集仅保留可提供数据变异性信息的基因。因此,通常使用高度可变基因 (HVG) (Brennecke et al,2013)。根据任务和数据集的复杂性,通常选择 1,000 至 5,000 个 HVG 用于下游分析(见图 EV1 和数据集 EV1)。Klein et al (2015) 的初步结果表明,下游分析对 HVG 数量的准确选择具有稳健性。在 200 到 2400 之间变化 HVGs 数量时,作者报告了 PCA 空间中类似的低维表示。基于这一结果,我们宁愿选择更高数量的HVGs。
Dimensionality reduction
特征选择后,单细胞表达矩阵的维数可以通过专门的降维算法进一步降低。这些算法将表达式矩阵嵌入到一个低维空间中,目的是在尽可能少的维度中捕获数据中的底层结构。这种方法的工作原理是单细胞 RNA-seq 数据本身就是低维的 (Heimberg et al,2016)。换句话说,细胞表达谱所在的生物流形可以用比基因数量少得多的维度来充分描述。降维旨在找出这些维度。
降维方法主要有两个目标:可视化和提取主要变化。可视化是尝试以二维或三维的方式对数据集进行最佳描述。这些缩小的尺寸用作散点图上的坐标,以获得数据的直观表示。相反,对于描述数据中存在的变异性,较高的组分变得不太重要。摘要技术可以通过找到数据的固有维数,将数据简化为其基本组成部分,因此有助于下游分析。虽然 2 维可视化输出不应用于汇总数据集,但可以使用汇总方法,使用领先的缩减组件对数据进行可视化,专门的可视化技术通常可以更好地表示变异性。
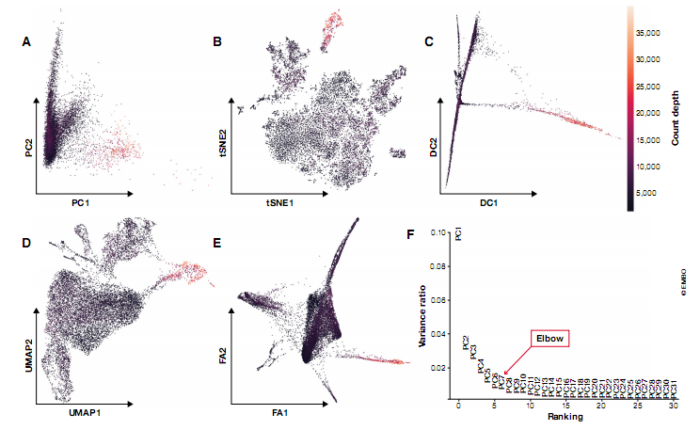
通过特征空间维度(基因表达载体)的线性或非线性组合减少维度。特别是在非线性的情况下,在这个过程中牺牲了降维的可解释性。一些常用的降维方法的应用示例如图 4 所示。随着越来越多的方法可供选择,详细回顾这些方法超出了本教程的范围。我们简要概述了可能帮助用户在常用降维方法之间进行选择的实际考虑。Moon et al (2018) 提供了单细胞分析降维的更详细综述。
图 4. scRNA-seq 数据的常见可视化方法。mhaber et al (2017) 提供的小鼠肠上皮区域数据显示了前两个组件:(A) PCA,(B) t-SNE,(C) 扩散图,(D) UMAP 和 (E) 通过 ForceAtlas 2 的力导向图布局。根据计数深度对细胞进行染色。(F) 前 31 个主成分 (PC) 解释的方差。该图用于选择相关 PC 分析数据集,位于 PC 5 和 7 之间。
两种流行的降维技术(主要是总结方法)是主成分分析 (PCA;Pearson,1901) 和扩散图(diffusion maps )(Coifman 等,2005),Haghverdi 等 (2015) 推广用于单细胞分析。主成分分析是一种线性方法,通过最大化每个进一步维度中捕获的残差来生成降维。尽管 PCA 并不像非线性方法那样能够捕获很少维度的数据结构,但它是目前许多可用的聚类或轨迹推断分析工具的基础。PCA 是一种常用的非线性降维预处理方法。通常,PCA 通过其前 N 个主成分汇总数据集,其中 N 可以通过肘部启发式(见图 4F)或基于置换测试的 jackstraw 方法(Chung Storey, 2015; Macosko et al, 2015).PCA 的简单线性的优点是在减少的维空间距离在这个空间的所有区域有一致的解释。因此,我们可以将感兴趣的数量与主成分相关联来评估它们的重要性。例如,主成分可以投影到技术干扰协变量上,以研究 QC 的性能、数据校正和标准化步骤 (Buttner et al,2019),或显示基因在数据集中的重要性 (Chung et al,2019)。由于扩散成分强调的是数据中的转换,它们主要用于连续过程(如差异)感兴趣的情况。通常,每个扩散组分(即扩散图维度)突出显示不同细胞群的异质性。
Visualization
出于可视化目的,使用非线性降维方法是标准实践(图 4)。scRNA-seq 可视化最常用的降维方法是 t 分布随机邻域嵌入(t-SNE;van derMaaten & Hinton, 2008)。t-SNE 降维以全局结构为代价来获取局部相似性。因此,这些可视化可能夸大细胞群体之间的差异,并忽略这些群体之间的潜在联系。另一个困难是选择其复杂度参数,因为 t-SNE 图可能显示不同数值的簇 (Wattenberg et al,2016)。t-SNE 常用的替代方法是UMAP(预本: McInnes & Healy, 2018) 或者基于图的工具,如 SPRING (Weinreb et al,2018)。UMAP 和弹簧力导向布局算法 ForceAtlas2 可以说是底层拓扑的最佳近似值(Wolf et al,2019,Supplemental Note 4)。该比较中 UMAP 的不同之处在于其扩展至大量细胞的速度和能力 (Becht et al,2018)。因此,在没有特殊生物学问题的情况下,我们将 UMAP 视为探索性数据可视化的最佳实践。而且,UMAP 还可以在两个以上维度汇总数据。虽然我们不知道 UMAP 在数据汇总中的任何应用,但它可能证明是 PCA 的合适替代方法。
细胞水平上经典可视化的替代方法是基于分区的图形抽象 (PAGA;Wolf et al,2019)。该工具已被证明可以充分近似数据的拓扑结构,同时使用集群粗粒化可视化。结合上述任何一种可视化方法,PAGA 都会产生粗粒度的可视化,这可以简化单细胞数据的解释,尤其是用大量细胞的时候。
问题和建议:
•我们建议根据数据集的复杂性选择1000到5000个高度可变的基因。
•当基因表达值被归一化为零均值和单位方差时,或当模型拟合的残差被归一化表达值时,不能使用使用基因表达均值和方差的特征选择方法。因此,在选择HVGs之前,必须考虑要进行什么预处理。
•应分别考虑降维方法进行总结和可视化。
•我们推荐使用 UMAP 进行探索性可视化;使用 PCA 进行一般性总结;使用扩散图替代 PCA 进行轨迹推断总结。
•具有 UMAP 的 PAGA 是可视化特别复杂数据集的合适替代方案。
Stages of pre-processed data
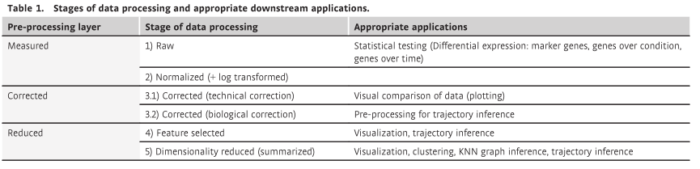
虽然我们已经将 scRNAseq 中常见的预处理步骤概述为上述工作流程,但下游分析通常倾向于采用不同水平的预处理数据,建议根据下游应用调整预处理。为了向新用户阐明这种情况,我们将预处理划分为 5 个数据处理阶段:
- (i) 原始数据,
- (ii) 标准化数据,
- (iii) 校正数据,
- (iv) 特征选择数据,
- (v) 降维数据。
这些数据处理阶段分为三个预处理层:
- 测量数据、
- 校正数据和
- 缩减数据(降维)。
应始终进行细胞和基因 QC,因此从此处省略。而这些处理层的顺序代表了 scRNA-seq 分析中的典型工作流程,也可以跳过某个处理层或者在处理阶段的顺序上有轻微的改变。例如,对于单批数据集,可能不需要进行数据校正。在表 1 中,我们总结了每一层预处理数据的适当下游应用。
表 1 预处理阶段分为实测数据、校正数据和缩减数据 3 组。我们将测量数据定义为原始数据和保留零结构的处理数据。通过使用cell特定因子缩放计数数据,全局缩放规范化方法即使在 log (+ 1)转换之后也保留 0 表达值。相反,纠正不需要的变异性数据替代零表达值。校正后的数据层代表数据的最干净版本,是基础生物信号的最接近近似值。我们称最后的预处理层为缩减数据。该数据层强调数据的主要方面,可以使用简化的功能集进行描述。
上述特征决定了预处理数据对于特定下游应用的适用性。作为最后的预处理阶段,缩减数据将是广泛适用的数据层的候选。然而,差异表达检测仅在基因空间中进行生物学解释,未(完全)用简化数据表示。约简数据的作用在于生物学的总结和噪声的减少,可能掩盖生物信号。因此,缩减数据用于需要数据总结(可视化、邻域图推理、聚类)的探索性方法和计算复杂的下游分析工具(轨迹推理)。的确,许多轨迹推理方法在工具本身中加入了降维过程。
单个基因的表达谱只能在基因空间中进行比较,在测量和校正数据中捕获。表达谱的比较可以通过可视化和统计学进行。我们认为应该对不同的数据层进行可视化和统计比较。基因表达的目测检查,校正数据最为合适。如果提供原始数据进行可视化比较,则要求用户固有地理解数据中的偏倚,以解释结果,校正数据有助于这种解释。然而,此处应单独考虑技术和生物学变量的校正数据。对生物变量的校正可能增加特定生物信号的强度,也将掩盖可能相关的其他信号。因此,生物校正数据主要适用于关注特定生物过程(如轨迹推理方法)的分析工具。
基因表达的统计学比较在测量数据层上最合适。没有完美的数据校正方法可用于消噪、批次校正或其他变异来源的校正。因此,数据校正方法不可避免地对数据进行了过高或过低的校正,因此以一种非预期的方式改变了至少一些基因表达谱的方差。基因表达的统计检验依赖于评估背景方差,作为数据中噪声的无效模型。由于数据校正倾向于减少背景变异(图 EV2),背景变异被数据校正方法过度校正的基因将更可能被评估为显著差异表达。此外,某些数据校正方法(例如 ComBat)将不符合实验设计的表达信号解释为噪声,随后从数据中删除。除了低估噪声外,这种实验设计信号的优化会导致高估效应量。鉴于这些考虑,使用测量数据作为输入,而不是使用校正数据,构成了对差异试验更保守的方法。使用测量数据,在差异检验模型中可以并且应该考虑技术变量。
上述观点得到了最近一次scRNA-seq差异分析方法比较的支持,该方法仅使用原始数据和规范化数据作为输入(Soneson & Robinson, 2018)。本研究使用的归一化数据仅围绕全局标度方法。然而,目前许多可用的非线性归一化方法模糊了归一化和数据校正之间的界限(参见“归一化”一节)。这种标准化的数据可能不再适合作为差异分析的输入。
问题和建议:
•使用测量数据(measured data)进行统计检验,根据发现的生物数据流形,对数据进行可视化比较时使用修正数据(corrected data ),对其他下游分析时使用简化数据( reduced data )。
Downstream analysis
预处理后,我们称之为下游分析的方法被用于提取生物学见解并描述潜在的生物学系统。这些描述是通过拟合数据的可解释模型获得的。这些模型的例子是
- 具有相似基因表达谱的细胞群代表细胞类型簇;
- 相似细胞之间基因表达的微小变化表示连续(分化)轨迹;
- 或具有相关表达谱的基因表明其共同调节作用。
下游分析可分为细胞水平和基因水平的方法,如图 5 所示。细胞水平的分析通常集中于两种结构的描述:簇和轨迹。这些结构又可以在细胞和基因水平上进行分析,从而形成聚类分析和轨迹分析方法。
图 5. 下游分析方法概述。方法分为细胞水平和基因水平分析。细胞水平分析方法再次细分为聚类分析和轨迹分析分支,这也包括基因水平分析方法。所有蓝色背景的方法都是基因水平的方法。
大体上,聚类分析方法试图根据细胞的聚类来解释数据的异质性。相比之下,在轨迹分析中,数据被视为动态过程的快照映射。在这里,我们描述了细胞和基因水平的群集和轨迹分析工具,在详细描述独立于这些细胞结构进行的基因水平分析之前。
Cluster analysis
将细胞聚类通常是任何单细胞分析的第一个中间结果,簇允许我们推断细胞类型。根据细胞基因表达谱的相似性对细胞进行分组,得到细胞簇。通过距离度量来确定表达谱相似性,通常将降维结果作为输入。相似性评分的一个常见示例是欧几里德距离,该距离在 PC 缩减的表达空间上计算。目前主要有两种方法聚类产生细胞簇:聚类算法和社区检测算法(community detection)。
聚类是一种经典的无监督机器学习方法,直接基于距离矩阵。通过最小化簇内距离或在减少的表达空间中找到致密区域,将细胞分配给 clusers。流行的 k-means 聚类算法通过确定簇中心并将细胞分配到最近的簇中心,迭代优化质心位置,将细胞分为 k 个簇(MacQueen,1967)。这种方法需要输入预期的簇数量,通常是未知的,必须进行启发式校准。k-means 应用于单细胞数据的距离指标各不相同。标准欧氏距离的替代方法包括余弦相似性 (Haghverdi et al,2018)、基于相关性的距离度量 (Kim et al,2018) 或 SIMLR 方法,该方法使用高斯核学习每个数据集的距离度量 (Wang et al,2017)。最近的一项比较表明,当使用 k-means或作为高斯核的基础时,基于相关的距离可能优于其他距离指标 (Kim et al,2018)。
社区检测方法是图聚类算法(graph-partitioning algorithms),依赖于单细胞数据的图表示。这个图的表示是使用 K 最近邻方法(KNN 图)。图中将细胞表示为节点,每个细胞与其 K 个最相似的细胞相连,这些细胞通常使用欧氏距离在 PC 缩减的表达空间上获得。根据数据集的大小,K 通常设置在 5 到 100 个最近的邻居之间。所得图表获取了表达数据的基础拓扑结构 (Wolf et al,2019)。表达空间的密集采样区域表示为图的密集连通区域。使用社区检测方法检测这些密集区。社区检测通常比一般的聚类更快,因为只有相邻的细胞对必须被认为属于同一个集群。这种方法大大减少了可能群的搜索空间。
在首创表型法(PhenoGraph method) (Levine et al,2015) 后,单细胞数据集的标准聚类方法已成为多分辨率模块优化(Newman & Girvan,2004;如Louvain算法(Blondel et al, 2008)实现在单细胞KNN图上。已经成为Scanpy和Seurat单细胞分析平台中默认聚类的方法。已有研究表明,它在单细胞RNA- seq数据聚类方面优于其他聚类方法(Duo ' et al, 2018;(Freytag et al, 2018)。
从概念上讲,Louvain 算法将社区检测为一组单元,它们之间的链接比从单元的总链接数预期的要多。优化的模块功能包含一个解析参数,允许用户确定集群分区的规模。通过子集 KNN 图,也可以只对特定的集群进行子集。这样的子聚类可以允许用户识别细胞类型聚类内的细胞状态 (Wagner et al,2016),但也可能导致仅来自数据噪声的模式。
问题和建议:
•我们建议在单细胞KNN图上通过Louvain社区检测进行聚类。
•聚类不需要在单个分辨率下执行。
特定的cell群是关注数据集中更详细的子结构的有效方法。
Cluster annotation
在基因水平上,通过寻找每个聚类的基因特征对聚类数据进行分析。这些所谓的标记基因(marker genes)表征了该簇,并被用来用一个有意义的生物学标签来注释它,该标签代表细胞簇内细胞的身份。由于任何聚类算法都会产生数据的分区,所以只有成功注释所代表的生物学才能确定所识别的聚类的有效性。
虽然可以假设在单细胞数据中检测到的簇代表细胞类型,但有几个变异轴决定了细胞同一性 (Wagner et al,2016;Clevers et al,2017)。首先,并不总是清楚什么是细胞类型。例如,虽然 T 细胞可能是某些细胞类型的满意标记,但其他细胞可能在数据集中寻找 T 细胞亚型并区分 CD4 和 CD8 T 细胞 (Wagner et al,2016;Clevers et al,2017)。此外,相同细胞类型的细胞在不同状态下可在单独的簇中检测到。由于上述原因,最好使用术语细胞身份(cell identities)而不是细胞类型(cell types)。在分群和注释群之前,用户必须决定哪一级别的注释细节,从而决定哪一级集群的分辨率。
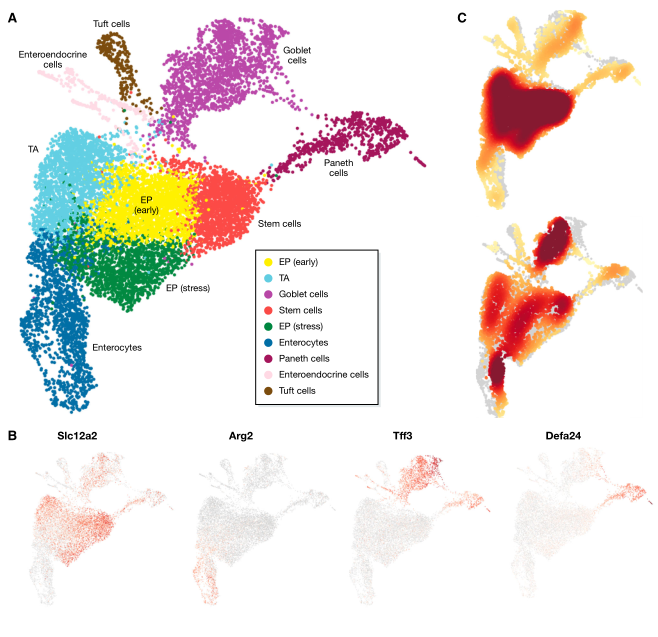
识别和注释簇依赖于使用描述单个细胞身份预期表达谱的外部信息来源。感谢最近和正在进行的努力,如小鼠大脑图谱 (Zeisel et al,2018) 或人类细胞图谱 (Regev et al, 2017),可用的参考数据库越来越多。这些数据库极大地方便了细胞身份注释。在没有相关参考数据库的情况下,可以通过比较数据来源的标记基因与来自文献的标记基因(见 project github 的案例研究)或直接可视化文献来源的标记基因的表达值(图 6B)来注释细胞身份。应该注意的是,后一种方法将用户限制在对来源于表达研究的细胞类型的经典理解,而不是细胞身份。此外,研究表明,常用的细胞表面标志物定义细胞特性的能力有限 (Tabula Muris Consortium et al,2018)。
图 6. Haber (2017) 小鼠肠上皮数据集的聚类分析结果。(A) 由Louvain聚类发现的带注释的细胞识别簇,在 UMAP 表示中可视化。(B) 细胞识别标记物表达,以鉴定干细胞 (Slc12a2)、肠细胞 (Arg2)、杯状细胞 (Tff3) 和潘氏细胞 (Defa24)。从低表达(灰色)到高表达(红色)可视化校正表达水平。如杯状细胞和潘氏细胞所示,标记基因也可能在其他细胞同一性群体中表达。近端(上)和远端(下)肠上皮区域的细胞-同一性组成热图。相对高细胞密度显示为暗红色。
使用参考数据库信息注释集群有两种方式:使用数据来源的标记基因或使用全基因表达谱。可通过应用两组之间的差异表达 (DE) 检验找到标记基因集:一组中的细胞和数据集中的所有其他细胞(参见差异表达检验)。典型的,我们集中在感兴趣的簇中上调的基因。由于标记基因预期具有较强的差异表达效应,因此通常使用简单的统计检验,如 Wilcoxon 秩和检验或 t 检验,通过基因在这两组之间的表达差异进行排序。将各检验统计量中排名靠前的基因视为标记基因。通过富集试验、Jaccard 指数或其他重叠统计,比较数据集中的标记基因和参考数据集中的标记基因,可对聚类结果进行注释。引用 web 工具,如www.mousebrain.org(Zeisel et al,2018) 或http://dropviz.org/(Saunders et al,2018) 允许用户可视化参考数据集中数据集标记基因的表达,以促进细胞识别注释。
检测标记基因时应注意两个方面。首先,标记基因获得的 P 值基于获得的细胞簇代表实际生物学过程。如果考虑到聚类的不确定性,在统计检验中必须考虑到聚类与标记基因检测的关系。由于聚类和标记基因通常是基于相同的基因表达数据确定的。DE 检验中隐含的无效假设是基因在两组之间具有相同的表达值分布。然而,由于这两组是由标记基因检测中的聚类方法的输出定义的,它们的基因表达谱在设计上存在差异。因此,即使对 splatter 生成的随机数据进行聚类,我们也发现了显著的标记基因 (Zappia et al,2017)(见附录补充文本 S3)。为了在聚类数据中获得一个合适的显著性度量,可以使用置换检验来解释聚类步骤。本试验在附录补充文本 S3 中详细说明。最近的一种差异表达工具也专门解决了这一问题(预印:Zhang et al,2018)。在当前设置下,P 值通常被夸大,这可能导致高估标记基因的数量。然而,基于 P 值的基因排序不受影响。假设聚类有生物学意义,排名靠前的标记基因仍将是最佳标记基因候选。首先,我们可以通过可视化检查预先验证标记基因。我们强调,通过无监督的聚类方法,当通过单个基因的表达确定细胞同一性群集时,可以解释所有其他基因的 P 值。这种单变量的聚类注释方法虽然常见,但不推荐在特殊情况下使用(例如 β 细胞中的胰岛素或红细胞中的血红蛋白)。其次,标记基因在数据集中区分一个簇与其他簇,因此不仅依赖于细胞簇,还依赖于数据集组成。如果数据集组成不能准确代表背景基因表达,检测到的标记基因将偏向于缺失的部分。特别是在计算细胞多样性较低的数据集的标记基因时,必须考虑这方面。
最近,自动集群注释已经可用。通过直接将注释参考簇的基因表达谱与单个细胞进行比较,scmap (Kiselev et al,2018b) 或 Gaett (preprint:Pliner et al,2019) 等工具可以在参考和数据集之间传输注释。因此,这些方法可以同时执行注释和聚类分群,而不需要数据驱动的聚类。由于细胞类型和状态组成在实验条件之间存在差异(Segerstolpe et al,2016;Tanay)基于参考数据的聚类不应取代数据驱动的方法。
聚类、聚类注释、重新或子聚类和重新注释的迭代可能是耗时的。自动注释方法极大地加速了这一过程。然而,自动化和手工方法有其优点和局限性,很难推荐一种方法而不是另一种。速度的提高与灵活性的降低是一致的。如上所述,参考图谱将不包含与研究数据集完全相同的细胞标识。因此,不应放弃标记基因计算进行手动注释。特别是对于包含许多集群的大型数据集,目前的最佳实践是两种方法的组合。为了提高处理速度,自动化的细胞识别注释可以用于粗略标记细胞和识别可能的子簇。随后,应对数据集簇计算标记基因,并与来自参考数据集或文献的已知标记基因集进行比较。对于较小的数据集和缺少参考图谱的数据集,手动注释即可。
问题和建议:
•不要使用标记基因p值来验证细胞身份群集,特别是当检测到的标记基因不能帮助注释群落时。p值可能被夸大。
•请注意,同一细胞识别簇的标记基因在不同数据集之间可能完全由于数据集细胞类型和状态组成而不同。
•如果存在相关的参考图集,我们建议使用自动化的集群注释,并结合基于数据的标记基因的手动注释来注释集群。
Compositional analysis
在细胞水平,我们可以根据其组成结构分析聚类数据。成分分析(Compositional analysis
)围绕着每个细胞同一簇的细胞比例,这些比例可因疾病反应而改变。例如,已证实沙门氏菌感染可增加小鼠肠上皮中肠细胞的比例 (Haber et al,2017)。研究单细胞数据中的组成变化需要足够的细胞数量来有力地评估细胞-同一簇的比例,以及足够的样本数量来评估细胞-同一簇组成中的预期背景变化。由于适当的数据集最近才出现,因此尚未开发专用工具。在上述小鼠研究中,使用泊松过程模拟细胞实体计数,包括条件作为协变量,检测到的细胞总数作为偏移。此处,可对回归系数进行统计检验,以评估特定细胞鉴别的频率是否发生显著变化。然而,相同数据集中其他细胞身份的检测并不相互独立。如果一个细胞同一性簇的比例发生变化,所有其他细胞同一性簇的比例也必须发生变化。因此,使用该模型无法评估总体组成是否发生显著变化。在没有专用工具的情况下,组成数据的可视化比较可以提供样品之间组成变化的信息(图 6C)。该领域的未来发展将可能借用流式细胞计数法(mass cytometry)(如 Tibshirani 等,2002;Arvaniti)或者微生物组文献 (Gloor et al,2017),其中成分数据分析受到了更多关注。
问题和建议:
•考虑样本间细胞同一簇比例变化的统计检验是相互依赖的。
Trajectory analysis
Trajectory inference
细胞多样性不能用诸如群集这样的离散类化系统来充分描述。驱动所观察到的异质性生物学过程是连续的过程(Tanay & Regev, 2017)。因此,为了捕捉细胞身份、分支分化过程或生物功能中渐进的、不同步的变化,我们需要基因表达的动态模型,这类方法称为轨迹推断(TI)。
轨迹推理方法将单细胞数据解释为连续过程的快照。通过寻找穿过细胞空间的路径,使相邻细胞之间的转录变化最小化,重建了这一过程(图 7A 和 B)。细胞的排列顺序径由伪时间(pseudotime)变量描述。虽然该变量与根细胞的转录距离有关,但其通常被解释为发育时间的代表 (Moignard et al,2015;Haghverdi et al,2016;Fischer et al,2018;Griffiths et al,2018)。
图 7. Haber (2017) 小鼠肠上皮数据的轨迹分析和图形提取(graph abstraction )。(A) Slingshot 推断的远端和近端肠细胞分化轨迹。远端谱系显示由红到蓝的伪时间颜色。数据集中的其他cell为灰色。PCA 空间中集群上的弹弓轨迹。细胞簇缩写如下:EPenterocyte progenitors;Imm,耳鼻喉科。未成熟肠上皮细胞;耳鼻喉科。成熟肠细胞;近端;距离。远端。(C) 图 7A 中肠细胞远端轨迹的密度。颜色代表每个假时间箱中的主导集群标签。投射到 UMAP 表示的数据集的抽象图形表示。集群显示为彩色节点。将出现在其他轨迹中的群集标记为比较。TA 表示转运扩增细胞。(E) 使用 GAM R 文库在一般肠细胞轨迹中假时间内的基因表达动态。
自从 Monocle(Trapnell 等,2014)和 Wanderlust(Bendall 等,2014)方法提出以来,可用方法的数量激增。目前可用的 TI 方法在建模路径的复杂性方面有所不同。模型的范围从简单的线性或分叉轨迹,到复杂的图形、树或多分支轨迹。在最近的 TI 方法综合比较中 (Saelens et al,2018),得出的结论是,对于所有类型的轨迹,没有一种方法的性能最佳。相反,TI 方法应根据预期轨迹的复杂性进行选择。比较显示,Slingshot (Street al,2018) 在线性模型、双叉模型和多分支模型的简单轨迹方面优于其他方法。如果预期有更复杂的轨迹,作者推荐 PAGA (Wolf et al,2019)。如果确切的轨迹模型已知,也可以使用更专业的方法来改善性能 (Saelens et al,2018)。一般而言,任何推断的轨迹均应使用替代方法进行确认,以避免方法偏倚。
在典型的工作流中,当有一个内建的降维步骤时,TI 方法被应用于约简数据或校正数据。由于多个生物过程通常在细胞内同时发生,因此逐步消除其他过程的生物效应可能有助于隔离预期轨迹。例如,T 细胞可能在成熟过程中进行细胞周期转换 (Buettner et al,2015)。此外,由于一些性能最好的 TI 方法依赖于聚集数据,TI 通常在聚集后进行。推断轨迹中的聚类可能代表稳定或亚稳态(参见亚稳态;图 7B 和 C)。随后,RNA 速度可以叠加到轨迹上,以增加方向性 (La Manno et al,2018)。
推断轨迹不一定代表生物过程。首先,这些仅表示转录相似性。少数 TI 方法包括对其模型中不确定性的评价 (Griffiths et al,2018)。因此,需要更多信息来确认是否确实采集了生物过程。这些信息可以以扰动实验、推断调控基因动力学和 RNA 速度支持的形式出现。
问题和建议:
•我们建议以 Saelens et 等人(2018)的综述为指南。
•推断的轨迹不一定代表一个生物过程。
应该收集进一步的证据来源来解释轨迹。
Gene expression dynamics
一种支持推断轨迹不是拟合转录噪声结果的方法是在基因水平分析轨迹。假时变化平滑的基因表征了轨迹,可用于识别潜在的生物学过程。再者,这组轨迹相关基因有望包含调控建模过程的基因。调节基因帮助我们理解如何以及为什么生物过程被触发,并代表潜在的药物靶标 (Gashaw et al,2012)。
虽然早期发现轨迹相关基因的方法涉及沿轨迹在细胞群之间进行 DE 测试 (Haghverdi et al,2016;Alpert et al,2018),但我们现在通过倒退假时基因表达检测沿轨迹变化的基因。为了使表达沿该协变量平滑变化,通过拟合样条或通过额外的局部回归步骤(例如 loess)平滑假时间。回归框架的噪声模型假设和用于描述假时间函数表达式的函数类别不同。通过对伪时间依赖基因进行模型选择,获得潜在的调控基因。伪时间上的 DE 测试被轨迹推理方法混淆,就像集群之间的 DE 测试被集群方法混淆一样(参见集群注释部分)。因此,在该设置中获得的 P 值不应视为显著性评价。
目前很少有专门的基因时间动力学工具存在。BEAM 是集成到 Monocle TI 流水线中的工具 (Qiu et al,2017a),允许检测分支特异性基因动态。在此管道之外,用户可以选择 LineagePulse(https://github.com/YosefLab/LineagePulse),它考虑了脱落噪声,但仍在开发中,或者使用 limma 包 (Ritchie et al,2015) 或标准 R 库编写自己的测试框架。可在在线弹弓教程 (Street et al,2018) 和图 7E 中找到这方面的示例。
由于可用的工具很少,研究基因时间动态的最佳实践还不能确定。基因动力学的探索性研究当然有可能使用上述所有的方法。高斯过程是研究基因时间动态的一个自然模型。此外,检测调控模块而不是单个基因可能会提高信噪比并促进生物学解释。
Metastable states
轨迹的细胞水平分析研究假时间内的细胞密度。假设细胞以无偏倚的方式被取样,沿着轨迹的密集区域表明首选转录状态。当把轨迹解释为一个时间过程时,这些致密区域可能代表亚稳态,例如,发展(Haghverdi 等,2016)。我们可以通过绘制假时间坐标直方图(图 7C)找到这些亚稳态。
Cell-level analysis unification
聚类和轨迹推断代表了单细胞数据的两种不同观点。这两个视图可以在粗粒度图表示中进行协调。通过将单细胞簇表示为节点,将簇之间的轨迹表示为边缘,可以表示数据的静态和动态性质。这种统一是由基于分割的图抽象工具提出的(PAGA;图 7D;Wolf et al,2019)。PAGA 使用一个细胞簇相互作用的统计模型,在细胞簇节点之间放置一个比预期更相似的边。在最近的综述中,PAGA 优于其他 TI 方法(Saelens 等,2018)。这是唯一审查的方法能够应付断开的拓扑和复杂的图表包含周期。这个特性使 PAGA 成为一个有用的工具,可以可视化整个数据集的拓扑结构,以便进行探索性分析。
Gene-level analysis
而我们到目前为止主要集中在表征细胞结构的基因水平分析方法,单细胞数据的基因水平分析具有更广泛的范围。差异表达检测、基因集分析和基因调控网络推断直接研究数据中的分子信号。这些方法不是描述细胞的异质性,而是使用这种异质性作为理解基因表达的背景。
Differential expression testing
关于表达数据的一个常见问题是,在两种实验条件下是否存在差异表达的基因。DE是一个有大量文献证明的问题,它起源于bulk rna 基因表达分析(Scholtens & von Heydebreck, 2005)。相对于bulk差异测试的一个优点是,我们可以通过在细胞识别簇中执行测试来解释单细胞环境中的细胞异质性。这种设置告诉我们,在特定的实验条件下,单个细胞的身份是如何进行转录反应的(Kang et al, 2018)。
尽管设计来回答相同的问题,但 bulk 和单细胞 DE 工具在方法上有所不同。虽然开发了bulk 方法以从少量样本中准确估计基因方差,但单细胞数据不存在此问题。另一方面,单细胞数据包含独特的技术噪声伪影,如脱落和高细胞间变异性 (Hicks et al,2017;Vallejos et al,2017)。专门为单细胞数据设计的方法考虑了这些人为因素 (Kharchenko et al,2014;Finak et al,2015)。然而,最近一项大规模的 DE 分析比较研究表明,bulk DE 测试包的性能与性能最好的单细胞工具(Soneson & Robinson, 2018)此外,当通过在测试中引入基因权重使散装工具适合模拟单细胞数据时,建议这些工具优于其单细胞对应物 (Van den Berge et al,2018)。根据该比较,性能最佳的 DE 分析工具为 DESeq2 (Love et al,2014) 和 EdgeR (Robinson et al,2010),结合 ZINB-wave (Risso et al,2018) 估计的权重。需要包括加权批量 DE 检测方法的独立比较研究来确认这些结果。
加权批量 DE 测试的改进性能是以牺牲计算效率为代价的。鉴于单细胞实验中细胞数量增加的趋势,算法运行时间正成为方法选择中越来越重要的考虑因素。因此,single-celltool MAST (Finak et al,2015) 代表了重量bulk DE 工具的有效替代品。MAST 使用栅栏模型来解释脱落,同时建立依赖于条件和技术协变量的基因表达变化模型。它是上述研究中表现最好的单细胞 DE 测试方法(Soneson & Robin-
son), 并在单个数据集的小规模比较中,表现优于bulk和单细胞方法 (Vieth et al,2017)。虽然 emast 的运行时间比加权批量方法快 10-100 倍 (Van den Berge et al,2018),但使用 limmaCvoom 可实现进一步 10 倍加速 (Law et al,2014)。尽管 limma 是一种bulk rna DE 试验方法,但 limmaCvoom 已被证明可实现与 MAST 相当的性能。
未校正的实测数据应用于 DE 检验,解释混杂因素对稳健估计差异表达基因至关重要。虽然 DE 测试工具通常允许用户灵活地加入混杂因素,但用户必须警惕哪些变量被添加到模型中。例如,在大多数单细胞实验设置中,样本和条件协变量被混淆,因为在多种条件下很少可能获得单个样本。如果我们将样本和条件协变量合并到模型中,与这些协变量相关的变异性就不能再明确分配。因此,当检验条件时,我们不能将样本协变量纳入给定形式的模型中。当校正多个分类批次协变量时,目测发现混杂的协变量组变得越来越困难。在这种情况下,检验模型设计矩阵是否是满秩的是有帮助的。即使设计矩阵不是完全秩,DE 测试工具也会经常调整矩阵并在没有输出警告的情况下运行。这不会产生预期结果。
在我们在此描述的情景中,条件协变量在实验设置中确定。因此,对该协变量(在同一聚类内)的 DE 检验独立于聚类程序。该设置区分了条件上的 DE 测试和分群上的 DE 测试。在条件下获得的 DE 检验 P 值代表预期的显著性指标,必须进行多重检验校正。为了减少多重检测负担,可能不相关的转录本可以从数据集中排除。而假基因或非编码 RNA 可以提供信息 (An et al,2017),它们在分析中往往被忽略。
问题和建议:
•DE测试不应该在校正数据(去噪、批次校正等)上进行,而应该在模型中包含技术协变量的测量数据上进行。
•用户不应该依赖DE测试工具来纠正带有混淆的协变量的模型。模型规范应该谨慎执行,以确保完整的设计矩阵。
•我们建议使用MAST或limma进行DE测试。
Gene set analysis
基因水平分析方法通常会产生长长的候选基因列表,难以解释。例如,数千个基因可能在处理细胞和对照细胞之间差异表达。我们可以根据共享特征将基因分组到集合中,并检测这些特征是否在候选基因列表中过度表达,从而促进这些结果的解释。
基因集信息可以在各种应用的精选标签数据库中找到。为了解释 DE 结果,我们通常根据共同的生物学过程对基因进行分组。生物过程标签存储在数据库中,如 MSigDB (Liberzon et al,2011)、Gene Ontology (Ashbuer et al,2000;the Gene Ontology Consortium,2017) 或 pathway 数据库 KEGG (Kanehisa et al,2017) 和 Reactome (Fabregat et al,2018)。Huang et al (2009) 和 Tarca et al (2013) 回顾并比较了大量工具,可以测试基因列表上注释的富集。
单细胞分析领域的一个最新进展是利用配对的基因标记进行配体受体分析。这里,细胞簇之间的相互作用是根据受体及其同源配体的表达推断的。配体受体对标记可以是从最近的 CellPhoneDB 中获得 (Vento-Tormo et al,2018),并使用统计模型解释跨集群的高表达基因 (Zepp et al,2017;Zhou et al,2017;Cohen et al,2018;Vento-Tormo et al,2018)。
Gene regulatory networks
基因并不独立发挥作用。相反,一个基因的表达水平是由与其他基因和小分子的调控相互作用的复杂的相互作用决定的。揭示这些调控相互作用是基因调控网络 (GRN) 推理方法的目标。
基因调控网络推断是基于相关、互信息等基因共表达的测量,或通过回归模型进行的(Chen & Mar, 2018)。如果两个基因表现出一种共表达信号,即使考虑到所有其他基因都是潜在的混杂因子,这些基因也被认为具有因果调控关系。推断基因调控关系与轨迹相关调控基因的检测有关。事实上,几种单细胞 GRN 推理方法使用轨迹与差异分析方程模型 (Ocone et al,2015;Matsumoto et al,2017)。
虽然有专门针对scRNA-seq数据开发的GRN推理方法(SCONE: Matsumoto et al, 2017;
PIDC: Chan等人,2017;最近的一项比较显示,bulk和单细胞方法在这些数据上的表现都很差(Chen & Mar, 2018)。GRN推断方法仍可能为识别生物过程的因果调节因子提供有价值的见解,但我们建议谨慎使用这些方法。
问题和建议:
•用户应警惕由此推断出生物关系中的不确定性。为调控关系而富集的基因模块比单个更可靠。
Analysis platforms
单细胞分析工作流是独立开发的工具。为了促进数据在这些工具之间的转移,围绕着一致的数据格式开发了单细胞平台,这些平台为分析管道的建设提供了依据。当前可用的平台存在于 R (McCarthy et al,2017;Butler et al,2018) 或 Python (Wolf et al,2018) 的命令行中,并作为本地应用程序 (Patel,2018;preprint:Scholz et al,2018) 或 Web 服务器 (Gardeux et al,2017;Zhu et al,2017) 使用图形用户界面 (GUI)。Zhu et al (2017) 和 Zappia et al (2018) 提供了平台概述。
在命令行平台中,Scater (McCarthy et al,2017) 和 Seurat (Butler et al,2018) 很容易与 R Bioconductor 项目提供的大量分析工具连接 (Huber et al,2015)。Scater 在 QC 和预处理方面具有特别的优势,而 Seurat 可以说是最流行和最全面的平台,它包括了大量的工具和教程。这个小组最近增加了 scanpy (Wolf et al,2018),这是一个不断增长的基于 python 的平台,它展示了对大量细胞。它充分利用了越来越多的用 Python 编写的工具,这些工具在机器学习应用程序中特别流行。
图形用户界面平台使非专业用户能够构建单细胞分析工作流程。用户通常通过规定的工作流程进行指导,以便于分析,但也限制了用户的灵活性。这些平台尤其适用于探索性分析。Granatum (Zhu et al,2017) 和 ASAP (Gardeux et al,2017) 等平台集成的工具不同,Granatum 包括的方法种类更多。作为网络服务器,这两个平台是现成的,但计算基础设施将限制其扩展到大型数据集的能力。例如,在仅有 92 个细胞的数据集上测试 ASAP。基于 Web 的 GUI 平台的替代方案是程序包,例如 FASTGenomics(预印:Scholz et al,2018)、iSEE (Rue-Albrecht et al,2018)、IS-CellR (Patel,2018) 和 Granatum(在本地服务器上运行)。这些都是平台和 GUI 包装器,可以与本地可用的计算能力进行缩放。未来,人类细胞图谱门户网站的持续发展(https://www.humancellatlas.org/data-sharing)将带来更强大的可视化数据探索工具,可扩展到大的cell数。
Conclusions and outlook
我们回顾了典型的 scRNA-seq 分析工作流程,并展示案例研究教程(httpsfwww.github.com/theislab/single-cell-tutorial)。本教程旨在遵循现有方法确定当前的最佳实践。虽然聚合单个最佳实践工具不能保证就是最佳的分析流程,但是我们的工作流程代表了单细胞分析领域最新技术水平的最新概览。因此,它为新来者提供了进入该领域的合适切入点,并借助人类细胞图谱的努力,以建立 scRNA-seq 分析的最佳实践 (preprint:Regev et al,2018)。应当注意的是,现有方法比较必然落后于最新方法开发。因此,我们提到了尚未在可能情况下独立评估的新发展。随着未来新的和更好的工具的发展,以及进一步的比较研究,这里提出的个别工具建议将需要更新,但关于数据处理阶段的一般考虑应保持不变。
两个特别感兴趣的开发途径是深度学习工作流和单细胞组学集成,因为它们有可能破坏现有分析流程。由于其向大数据扩展的灵活性,深度学习已经彻底改变了从计算机视觉到自然语言处理的领域,并开始在基因组学中产生强大的影响 (Webb,2018)。scRNA-seq 的首批应用开始从降维到去噪(例如 scVis:Ding et al,2018;scGen:preprint:Lotfollahi et al,2018;DCA:Eraslan et al,2019)。最近,深度学习已被用于产生一个嵌入式工作流,该工作流可拟合数据、消噪并在模型框架内进行聚类和差异表达等下游分析 (scVI:Lopez et al,2018)。在该设置中,可能将噪声和批效应估计值纳入下游统计检验中,同时保持数据方差的准确估计值。
随着单细胞 omic 技术的提高,对集成 omic 分析的需求将逐渐增长(Tanay & Regev, 2017)。未来的单细胞平台将必须能够处理不同的数据源,如 DNA 甲基化 (Smallwood et al,2014)、染色质可及性 (Buenrostro et al,2015) 或蛋白质丰度 (Stoeckius et al,2017),并包括整合这些模态的工具。对于这种设置,不再可能只使用单个读取或计数矩阵,我们将其用作本教程的起点。然而,平台已经在适应多模态数据结构,以整合 RNA velocity,这是根据未拼接和拼接读数数据计算的 (La Manno et al,2018)。单细胞多基因整合可以通过一致性聚类方法(SC3)、多基因因素分析 (Argelaguet et al,2018) 或多基因调控网络推断 (Colome-tatchen) 来实现。