为了确保单细胞转录组分析工具的可用性,许多开发人员已经做出了相当大的努力。到2021年5月28日,已经开发并提供了近1000种不同的生物信息学工具。
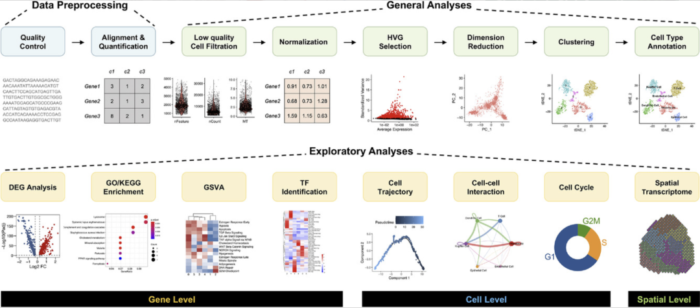
01 数据预处理
单细胞转录组原始测序数据的基本格式包括FASTQ和BCL格式,这些格式依赖于数据源和测序平台。由于只有FASTQ文件可以直接实现质控,一旦原始数据不是FASTQ格式,第一步就是用适当的工具将其转换为FASTQ格式。可以使用10x工具cellranger mkfastq从BCL文件生成FASTQ文件。重要的是,除了BCL文件的路径之外,还应该提供一个简单的CSV矩阵文件,至少包括三列(lane、sample和index)。然后,FastQC可以用于评估原始单细胞RNA测序数据的质量。
高质量的片段(reads)需要使用适当的比对软件(如STAR或Tophat)映射到特定的参考基因组。计数是Cell Ranger最重要的功能,其内部包含了比对、过滤、UMI计数和其他实际步骤。Cell Ranger使用了STAR作为比对软件,该比对器对基因组的片段进行剪接感知比对,然后使用转录注释一般转移格式(GTF)文件根据片段是否准确地与基因组比对,将这些片段分类为外显子、内含子和基因间区。
02 一般性分析
在单细胞悬液的制备过程中,由于不可避免的自然现象、实验操作和技术障碍,活细胞可能会死亡、细胞膜损伤或多细胞粘附。为了消除低质量细胞对基因表达的干扰,需要使用合适的工具进行第二轮质控,如Seurat、scran和scanpy。在引用方面,Seurat是最受欢迎的,它内置了处理低质量细胞过滤的功能。一个细胞是否需要保留,基本上可以通过以下质控指标来判断:基因的数量,UMI(转录本)的数量,线粒体基因的百分比,核糖体蛋白基因在每个细胞中的百分比。过滤阈值的设置没有绝对的标准,它通常取决于被分析的细胞和组织的类型。比如有研究过滤掉≤100或≥6000个表达基因、≤200个UMIs和≥10%线粒体基因的细胞;也有设置保留200<表达基因<2500、300<umis<15000、线粒体基因<10%的细胞。建议根据具体的疾病状态和组织类型的多样性,灵活调整上述qc阈值。需要注意的是,基于线粒体基因的细胞过滤应谨慎应用,因为某些细胞类型,如心肌细胞,在生物学上表达这些基因方面更丰富。
与分析传统的bulk RNA-seq数据类似,在分析单细胞RNA测序数据时,每个细胞都被视为一个独立的样本。原始表达矩阵不能直接用于下游分析,因为由于系统错误或技术噪音(如每个细胞的测序深度和转录组捕获率的差异),细胞之间的表达水平无法进行比较。标准化旨在抵消技术噪声或偏差,并确保每个细胞之间的可比性。在2020年,Lytal等人评估了七种归一化方法的有效性,包括BASiCS、GRM、Linnorm、SAMstrt、SCnorm、scran和Simple norm。值得注意的是,Linnorm和scran的速度优势来自于用C 编写,并在R中实现,适合于大数据集。相反,BASiCS和SCnorm需要更长的时间来生成更精确的结果。总的来说,这些方法之间存在很大的差异,不同的工具在不同的情况下表现最佳。
单细胞RNA测序数据集是高维的,一个样本中有数万个细胞,每个细胞中有数千个基因表达。每个细胞中的大部分基因都属于看家基因,因为它们的特点是细胞之间的表达水平没有显著变化,它们的存在往往会掩盖真正的生物信号。在数据集中表现出高度细胞间变异的特征子集也被称为高度可变基因(HVGs)。HVGs不仅突出了生物信号,而且由于计算量的显著减少,大大加快了对单细胞RNA测序数据下游分析的速度。一个高质量的HVGs应该包含能够区分不同细胞类型的基因,HVGs的质量对聚类的精度有显著影响。2018年,Yip等人评估了七种检测HVGs的方法,包括BASiCS、Brennecke、scLVM、scran、scVEGs和Seurat,发现不同方法的聚类结果以及运行时间存在很大差异。与其他方法相比,scran可以检测出稳定数量的HVGs,并且具有良好的运行时间。Brennecke在大范围数据集上具有稳定一致的性能。scran和Seurat在处理部分数据集时表现最佳。BASiCS和sclvm_logvar比其他的要慢得多。
不同的scRNA-seq数据可能产生于不同的时间、不同的测序平台,这些数据之间不可避免地存在着技术上或非生物学上的显著批次效应。scRNA-seq数据中的批次效应一直困扰着下游分析,因为它可以破坏基因表达模式,然后导致错误的结论。因此,批次效应校正对于分析scRNA-seq数据至关重要。虽然已经提出了一些针对scRNA-seq数据的批量效应校正算法,如Scanorama和Seurat V4,其只能一次合并两个数据集,并通过迭代整合多个数据集。它们中的大多数会消耗大量的计算内存和时间,并且随着scRNA-seq数据数量的增加,这一需求很可能会增加。最近,Zou等人提出了一种新的基于深度学习的方法,称为deepMNN,以纠正scRNA-seq数据中的批次效应。它比较了deepMNN和最先进的批次校正方法的性能,包括广泛使用的Harmony、Scanorama和Seurat V4方法,以及最近开发的基于深度学习的MMD-ResNet和scGen方法。结果表明,deepMNN的精度优于现有的常用方法,特别是在大数据集情况下。而deepMNN算法的时间复杂度和空间复杂度几乎是优良的。对大数据集完成批次效应校正耗时17 min,而Harmony和Scanorama分别耗时35和77 min。此外,它具有比Seurat V4和scGen更大的存储空间。同时,deepMNN可以在一步中整合多批数据集,无需多次迭代。deepMNN的这些特性使其有可能成为大规模单细胞基因表达数据分析的新选择。
除特征选择外,降维也是处理这类高维数据的主要策略之一。对于单细胞RNA测序数据,通常需要进行两轮降维,首先进行主成分分析(PCA)降维,然后进行t-分布式随机近邻嵌入(t-SNE)或统一流形近似与投影(UMAP)降维可视化。PCA是一种数学线性维度算法,它利用正交变换将一系列可能线性相关的变量转换为新的线性不相关的变量,从而利用新的变量在低维上显示数据的特征。PCA已广泛应用于scRNA-seq研究,以克服任何单一特征中广泛的技术噪音。Wu等在2019年对这两种非线性降维方法进行了系统的比较。他们指出UMAP在高维细胞学和单细胞RNA测序中的使用,特别强调与t-SNE相比UMAP表现出运行时间更快和一致性,以及更有意义的细胞群结构和连续体的维持。此外,UMAP在细胞子集的连续性方面比t-SNE有明显的优势,因为它保留了更多的全局结构,尽管t-SNE仍然应用于许多单细胞研究,似乎是由于更好的视觉偏好。
单细胞RNA测序数据的复杂性促进了广泛的聚类方法的发展。基于恢复已知亚群的能力、稳定性、运行时间和可伸缩性,最近的一篇论文在总共12个不同的数据集上评估了14种聚类方法。值得注意的是,SC3和Seurat在这些方法中综合来看表现更好,Seurat的速度要快几个数量级。在cluster数量相同的情况下,Seurat通常与真实区分的一致性最好,而FlowSOM在cluster数量大于真实数量时其与真实区分的一致性更好。
聚类后,为每个聚类分配生物学注释是后续分析的基础。通常,在scRNA-seq数据中注释细胞的工作流程包括三个主要步骤:自动注释、人工注释和湿实验验证。首先,主要的自动化注释工具利用一组预定义的标记基因,这些标记基因在已知的细胞类型中特异表达,通过将它们的基因表达模式与已知的细胞类型匹配来标记cluster。自动细胞标注方法的优点是快速、可重复性好,对常见细胞类型的标注结果更可靠。然而,由于参考标记基因集的限制,它无法定义罕见的和新的细胞类型。2020年,Huang等人对Seurat、scmap、SingleR、CHETAH、SingleCellNet、scID、Garnett、SCINA、CP和RPC等10种细胞类型注释方法进行了系统的比较和评估。他们发现,在Seurat、SingleR、CP、RPC和SingleCellNet这五种最常用的方法中,Seurat是注释主要细胞类型的最佳方法。然而,Seurat在预测罕见细胞类型和区分高度相似的细胞类型方面表现相对较差。其次,人工注释是标注细胞的金标准,虽然它需要搜索相关文献和挖掘已有的scRNA-seq数据,既主观又费力。最后,通常需要湿实验来进一步验证scRNA-seq的发现。传统的验证方法包括免疫荧光和免疫组化,这两种方法都是基于抗体与抗原(标记基因编码的表面蛋白)特异性结合的原理来证明数据分析得到的细胞类型的真实存在。此外,新兴的空间转录组测序技术也可以考虑提高注释的可靠性,它可以结合细胞成像和scRNA-seq在一个实验中检测空间转录模式和细胞形态。
03探索性分析
为了准确地揭示特定细胞群体的功能偏倚和生物学意义,有必要对目标差异表达基因集进行功能富集分析。功能富集的通用分析策略也适用于单细胞数据,如GO和KEGG通路。大量成熟的功能富集分析工具已经开发出来。Huang等人在2009年权衡了68种富集分析工具的优缺点后,综合比较了它们。此外,GSVA还以通路为中心的方式广泛应用于功能富集分析等标准分析中。GSVA可以计算每个样本中不同信号通路的富集分数,以评估表型差异的原因,可以作为KEGG通路的补充,使结果更具生物学解释性。
为了从scRNA-seq数据中识别每个细胞簇中富集的转录因子,Aibar等人于2017年开发了SCENIC可以实现转录因子(TF)的推断,首先通过搜索靶基因的假定的调控区域来富集转录因子基序。然后转录因子基序富集可以实现候选TF调控因子与候选靶基因的连接。虽然SCENIC可以用R和Python实现,但是强烈推荐使用pySCENIC来运行大数据集,因为它可以更快地实现SCENIC流程。值得注意的是SCENIC的最新版本支持智人、家鼠和黑腹果蝇,并有可能手动创建其他物种的定制数据库。虽然由于其出色的可扩展性和对各种数据库的稳健性而被广泛使用,但它忽略了不同细胞类型中基因调控机制的动态变化。2020年,Ma等人从scRNA-seq中开发了IRIS3,一个整合的细胞类型特异性调控推断服务器。在实际应用中,IRIS3更适合没有大量编程技能的研究人员使用其用户友好的web服务器。然而,IRIS3需要在准确性和效率上不断改进。
拟时分析可以在单细胞水平上推断细胞的轨迹,有望发现罕见的细胞类型和隐蔽的状态。在拟时分析方面,已经开发了不同类型的分析工具。2019年,Saelens等人对45种拟时分析工具进行了综合比较,发现现有工具具有很强的互补性。Monocle是应用最广泛的拟时分析工具之一,它借鉴显式主图来描述数据,并通过嵌入反向图来重建单细胞轨迹,以提高预测轨迹的稳健性和准确性。着重指出,建立单细胞基因表达动力学的整个过程在很大程度上是数据驱动的。
生物体受到刺激后会自我调节以维持体内稳态,这需要多种细胞的共同参与和协调。随着细胞-细胞通讯研究的快速发展,分析细胞-细胞通讯的工具不再有限,包括CellChat、CellPhoneDB、NicheNet、SingleCellSignalR和iTalk等。虽然每一种工具都依赖于细胞表面配体和受体相互作用的强度,但每一种工具都有其优缺点。具体来说,如果要考虑配体和受体的结构组成,CellPhoneDB是首选。如果需要考虑辅助因子(如启动子和拮抗剂)的调控,可以选择CellChat来提高性能。还建议灵活地结合多种细胞-细胞通信分析工具,以避免系统偏差。
单细胞悬液中的每个细胞都处于细胞周期的特定阶段:DNA合成前期(G1期)、DNA合成期(S期)、DNA合成后期(G2期)或有丝分裂期(M期)。每群细胞都混合着不同细胞周期的细胞。Seurat的CellCycleScoring功能根据其内置包内的G2/M和S期标记基因的表达给每个细胞打分。近年来,基于机器学习的方法已经被开发出来,从单细胞RNA测序数据预测细胞周期阶段。2015年,Scialdone等人比较了五种已建立的监督机器学习方法,以及基于转录组将细胞分配到其细胞周期阶段的定制预测器。他们特别指出,只有基于PCA的方法和定制的预测器性能最好,可以稳健地捕获细胞周期信号。
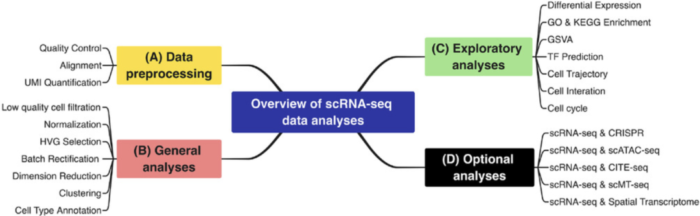
04可选择性分析
虽然解释了单细胞测序分析过程的主要步骤,但还有很多其他重要的方面值得更多的关注和探索,如scRNA-seq和CRISPR筛选的联合应用,scRNA-seq和多组学的综合分析,包括scATAC-sEquation(单细胞染色质可及性和转录组测序)、scMT-sEquation(单细胞甲基化组和转录组测序)、CITE-sEquation(通过测序对转录组和表位进行细胞索引)和空间转录组。这些技术的结合可以更好、更深入地了解关键的生物过程和机制,是未来单细胞技术发展的一个重要方向。在单细胞RNA转录组研究领域,分析算法和工具在改善数据探索和更好地理解细胞功能方面仍有很大的潜力。
参考文献:
Jovic, Dragomirka, Xue Liang, Hua Zeng, Lin Lin, Fengping Xu, and Yonglun Luo. "Single‐cell RNA sequencing technologies and applications: A brief overview."Clinical and Translational Medicine12, no. 3 (2022): e694.


