单细胞测序是目前研究的热点,那么我重新回来探究一下,首先问自己几个问题:
都是转录组测序,有什么不一样?
看到转录组我们都不陌生,有参无参也都做了许多了,但是对一个细胞测序,为什么呢?10X给出了一个说明
https://www.10xgenomics.com/solutions/single-cell/
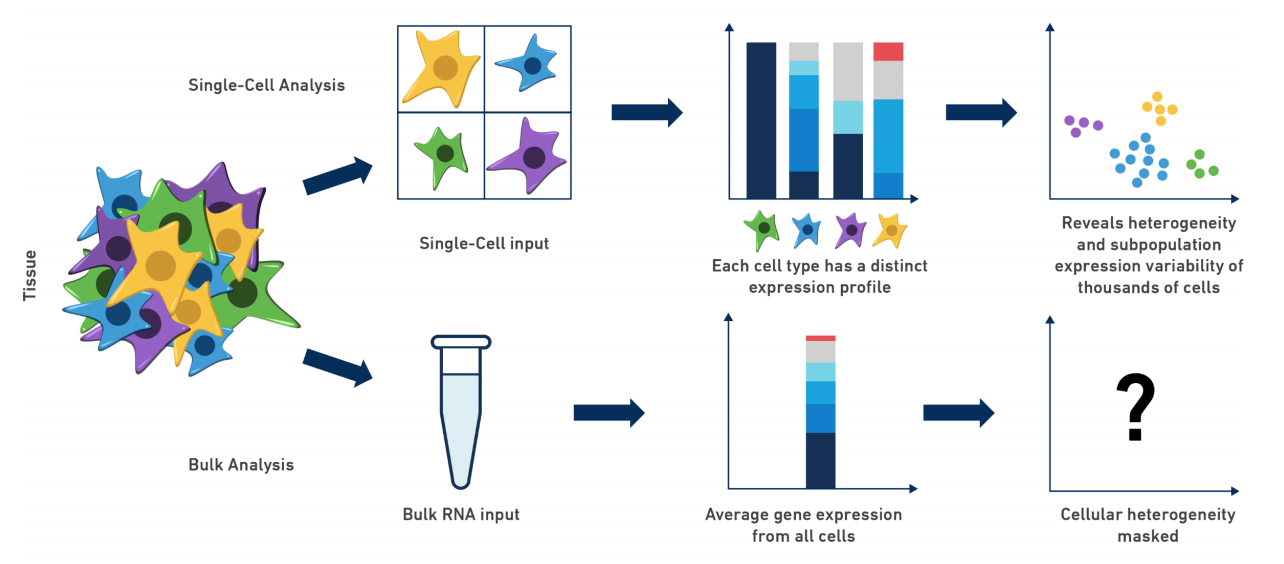
传统的是测一群细胞(Bulk RNAseq),然后我们定量是基于所有细胞内的基因平均表达量进行统计,这样得到的是组织之间的差异性,也就是说只能看到大体的趋势,但是对于继续深入研究还是存在比较大的背景噪音。对于癌症或者系统发育相关的研究,往往是想看到某些特殊的细胞发挥的作用,也是相当于一步步筛选。
单细胞更加精准,最终得到的是每个细胞型的表达量,从而可以帮助理解组织异质性,鉴定罕见的细胞类型,检测细胞组分的变化,例如在癌症异质性、胚胎发育、细胞对药物的反应、神经细胞分型、差异表达通路等方面都发挥重要作用
怎么去做单细胞研究?
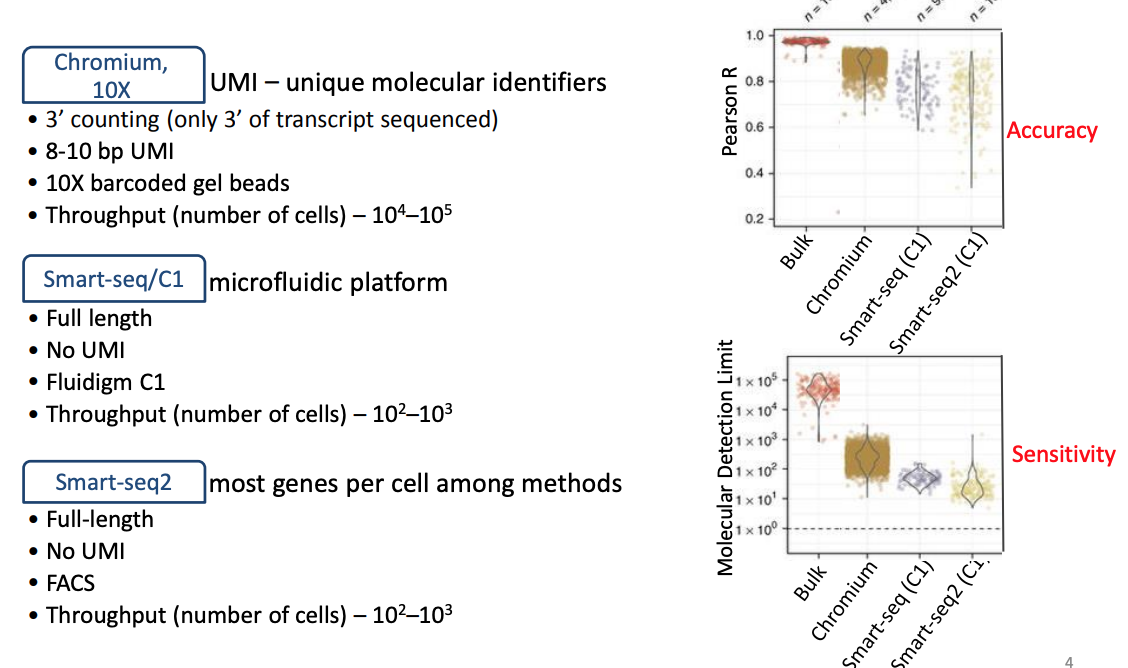
我们常规的转录组测序,需要提取样本RNA、建库测序,但是对于细胞来讲,材料提取是个大问题,但是目前主要有两个获取细胞的方法:Smart-seq和Drop-seq
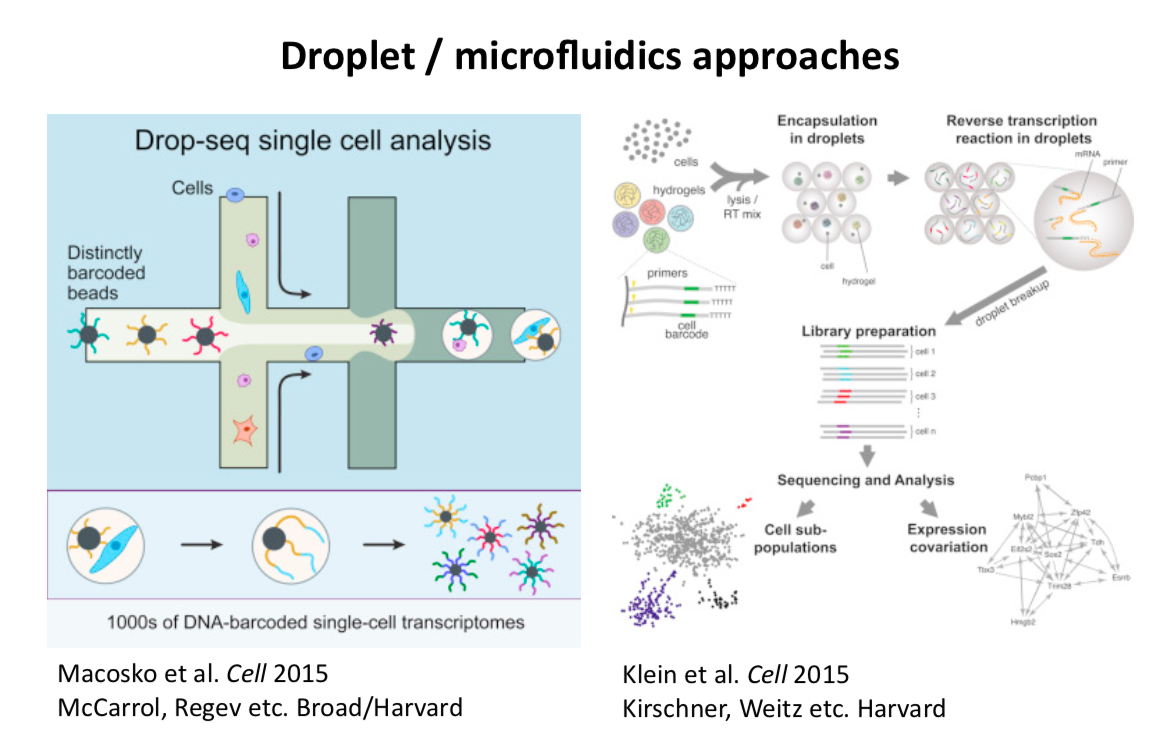
Smart-seq的特点就是测序细胞量少(因为一次通过一个细胞),但是测序的reads数多;Drop-seq(10X 公司的方法)一次可以测许多细胞,但是测得reads数都不是很多
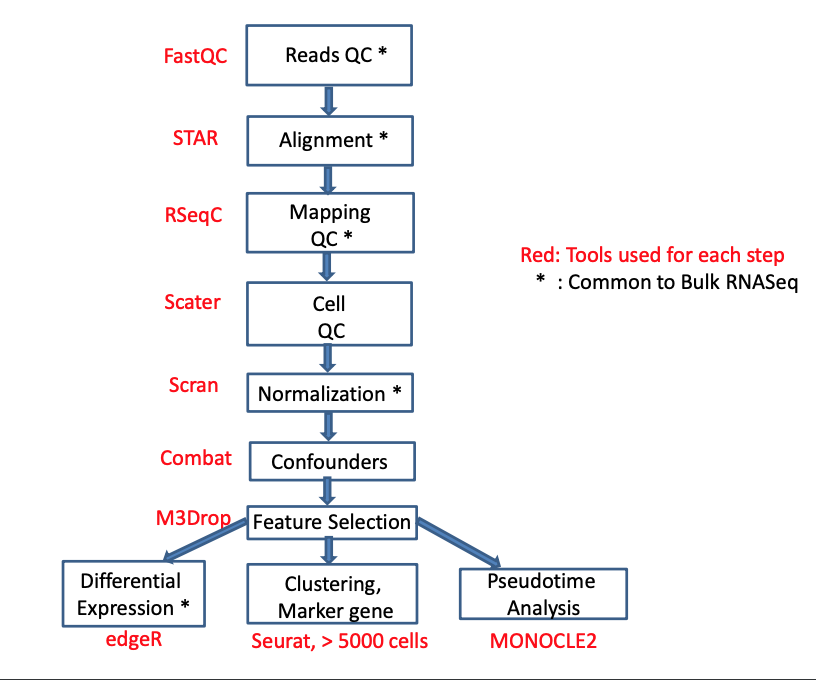
获得了样本,一般公司会直接给你表达矩阵,也就是一般的转录组测序利用featureCount获得的结果,自己需要先进行归一化(例如CPM)
当然,这仅仅是许多资料中的一个流程
- For differential expression:
- – SCDE: http://pklab.med.harvard.edu/scde/index.html
- – SCA: https://github.com/RGLab/SingleCellAssay
- – MAST: https://github.com/RGLab/MAST
- – SAMseq: http://cran.r-project.org/web/packages/samr
- For clustering etc.:
- – Monocle: https://github.com/cole-trapnell-lab/monocle-release
- – Rtsne: http://cran.r-project.org/web/packages/Rtsne
- – Sincell: http://master.bioconductor.org/packages/devel/bioc/html/sincell.html
- – scLVM: https://github.com/PMBio/scLVM
- – BASiCS: https://github.com/catavallejos/BASiCS
- – Pagoda: http://pklab.med.harvard.edu/scde
- – Seurat toolkit: http://www.satijalab.org/seurat.html
- – Sincera pipeline: https://research.cchmc.org/pbge/sincera.html