Abstract: Epigenetics is the study of meiotically and mitotically heritable changes in gene expression that are not coded for in the underlying DNA sequence. The term epigenetics is derived from epi - (meaning upon) and genetics . Epigenetic regulation of mammalian gene expression has profound effects in controlling cell growth, differentiation and cancer development. The important epigenetic mechanisms include DNA cytosine methylation, histone modifications and the more recently discovered non-coding RNAs. Non-coding RNAs are functional RNA molecules that are not translated into proteins. The main classes of regulatory non-coding RNAs include siRNA, miRNA, piRNA and long non-coding RNAs. There is growing evidence that regulatory non-coding RNAs play essential roles in the regulation of gene expression and are important in mammalian development and disease processes. Here, we review current research efforts aimed at understanding non-coding RNA s and their mechanisms of function in mammalian cells.
Keywords: epigenetics; non-coding RNAs; DNA methylation; histone modifications; mammalian cells
目前表观遗传学 (Epigenetics) 通常被定义为 基因表达通过有丝分裂或减数分裂发生了可遗传的改变 , 而 DNA 序列不发生改变。
表观遗传学的机制主要包括 DNA 甲基化、组蛋白修饰及非编码 RNA。
DNA 甲基化 (DNA methylation) 是指在 DNA甲基转移酶 (DNA-methyltransferases, DNMTs) 的催化下 , CpG 二核苷酸中的胞嘧啶被选择性地添加甲基 , 形成 5- 甲基胞嘧啶。 DNA 甲基转移酶有两种 , 其中 DNMT1 主要起维持甲基化的作用 , 能使半甲基化的 DNA 双链分子上与甲基胞嘧啶相对应的胞嘧啶甲基化 , 可参与 DNA 复制双链中新合成链的甲基化; 而 DNMT3a和 DNMT3b 主要起形成甲基化的作用 , 能在未发生甲基化的 DNA 双链上进行甲基化。 DNA 甲基化一般与基因的沉默相关 , DNA 去甲基化则与基因的活化相关。组蛋白修饰 (Histone modifications) 是指组蛋白的基础氨基末端尾部突出于核小体 , 常在转录后发生变化 , 包括甲基化、乙酰化、 磷酸化和泛素化等翻译后的修饰, 这些修饰构成了丰富的 “ 组蛋白 密码 ”(Histone code), 能影响染色质的压缩松紧程度,因此在基因表达中起重要的调节作用。其中甲基化是组蛋白 重要的修饰方式 , 多发生于组蛋白H3 、 H4 的赖氨酸 (K) 和精氨酸 (A) 残基上 , 组蛋白赖氨酸甲基化既可以导致激活 , 也可以导致抑制 , 通常取决于它所位于的残基情况。 如 H3K9 、 H3K27和 H4K20 甲基化一般与异染色质形成有关, 是学者们熟知的重要的 “ 失活 ” 标记物 (Hallmark), 而 “ 活性 ” 标记物则包括 H3K4 及 H3K36 的甲基化。乙酰化也是组蛋白重要的修饰方式 , 多发生于 N- 末端保守的赖氨酸残基上 , 如组蛋白 H3 上的 9 号和 14 号赖氨酸残基 , H4 上的 5 号、 8 号、 12 号和 16 号赖氨酸残基 , 组蛋白 H3 和 H4 上赖氨酸的乙酰化与活化或开放的染色质有关。 与此相反 , 赖氨酸残基经脱乙酰作用导致染色质压缩和基因的失活。不同的组蛋白修饰之间可以相互影响 , 并与 DNA 甲基化相互作用。非编码 RNA( Non-coding RNAs) 是指不能翻译为蛋 白的 功能性 RNA 分子 , 分为看家 非编码RNA(Housekeeping non-coding RNA) 和调控非编码RNA(Regulatory non-coding RNA), 其中具有调控作用的非编码 RNA 按其大小主要分为两类: 短链非编码 RNA( 包括 siRNA 、 miRNA 、 piRNA) 和长链非编码 RNA(Long non-coding RNA, lncRNA)( 表 1) 。 尽管近年来大量研究表明非编码 RNA 在表观遗传学修饰中扮演了重要的角色 , 能在基因组水平及染色体水平对基因表达进行调控 , 决定细 胞分化的命运, 但相对于其他生物 ( 酵母、 果蝇、 线虫及植物等 ), 哺乳动物细胞中表观遗传学的研究相对滞后。
图 1 非编码 RNA 在表观遗传学中的作用
1 siRNA
siRNA 来源于长的双链 RNA 分子 ( 包括 RNA 病毒复制子、转座子或转基因靶点等 ), 经 Dicer 酶剪切为 21~25 nt 的双链 RNA 片段 , 装载至 AGO 蛋白而发挥作用,近年研究表明 , siRNA 能在哺乳动物细胞中介导 DNA 甲基化和组蛋白修饰 , 从而导致转录基因沉默 (Transcriptional gene silencing, TGS) 。 Kawasak等首先合成了靶向 CpG 岛 E -cadherin 基因启动子的 siRNA, 通过亚硫酸氢盐修饰结 合测序法 (Bisulphite sequencing) 证实 , 同源 siRNA 转染的细胞 ( 人乳腺癌细胞 MCF-7 和人正常乳腺细胞 ), 其靶标 DNA 发生了特异性的甲基化及组蛋白 H3K9 的甲基化 , 而且该沉默依DNMT1/DNMT3b, 提示基因沉默发生于转录水平 , 是由 DNA 甲基化引起的。 Morris 研究结果表明 靶向 延长因子 1 α (Elongation factor 1alpha, EF1A) 启动子的 siRNA能够导致 TGS, 其机制与靶标的 DNA 甲基化密切相关, 进一步证实了哺乳动物细胞内siRNA 介导的TGS 的保守性。
表1 表观遗传学中起主要调控作用的非编码RNA
| 种类 | 长度(nt) | 来源 | 主要功能 |
| siRNA | ~21~25 | 长双链RNA | 转录基因沉默 |
| miRNA | ~21~25 | 含发卡结构的pri-miRNA | 转录基因沉默 |
| piRNA | ~24~31 | 长单链前体或起始转录产物等多途径 | 生殖细胞内转座子的沉默 |
| lncRNA | >200 | 多种途径 | 基因组印记和 X 染色体失活 |
但有实验报道: TGS 过程并未发生DNA 甲基化, 提示DNA 甲基化并非参与所有小RNA 介导的TGS, 或者是DNA 甲基化在暴露于小RNA 的过程中发生了变化。最近Morris 实验室的研究结果表明: 以siRNA 持续作用于人泛素C(Uboquitin C,UbC)基因启动子至少3 d, 可导致长期的基因沉默,靶标首先发生组蛋白甲基化, 随后是DNA 的甲基化,即靶标DNA 甲基化的检出明显迟于组蛋白修饰, 这在一定程度上可以解释并非所有的siRNA 介导的TGS 中都能检测到DNA 的甲基化。同时提示, 相对于组蛋白修饰而言, 启动子的DNA 甲基化是一个更容易遗传、更长久的沉默, 即DNA 甲基化主要在维持长期的基因沉默方面起作用。为探讨哺乳动物细胞内siRNA 介导的TGS 机制, Morris 等进一步研究了靶标启动子的组蛋白甲基化、转录的依赖性以及siRNA 的双链是否均参与了TGS, 实验结果表明siRNA 能引起靶标启动子EF1A 区域H3K9 和H3K27 的甲基化, 而且靶向启动子的siRNA 中, 只有其21 bp 的反义RNA 链—— 引导链(Guide strain) 对于TGS 是必需的, 且其主动转录需要RNA 多聚酶II(RNA polymerase II, RNAPII)的参与。
目前研究表明: Argonautes 蛋白家族 (AGO1 及AGO2), DNMT3a, 组蛋白去乙酰化酶(Histonedeacetylase-1, HDAC-1)和/或Polycomb 蛋白家族(Polycomb group, PcG)的EZH2 (Enhancer of zestehomolog 2)参与了siRNA 诱导的TGS [30, 37~39]。哺乳动物细胞内有4 种AGO, 其中主要是AGO1及AGO2 参与TGS, AGO1 虽然与AGO2 的同源性为80%, 但它缺乏一个关键的催化酶, 不能有效地裂解RNA, 因此对于AGO1 及AGO2 的选择可能依赖于小RNA 与靶标之间的互补性[38]。通常完全互补的dsRNA 前体诱导的染色质重塑是由AGO2 引发,而发卡状的dsRNA 前体诱导的染色质重塑是由AGO1 引发[40]。有意义的是, AGO 在TGS 中的作用早于靶标启动子的组蛋白甲基化, 当靶标沉默态组蛋白修饰( H3K9 甲基化)增加时, AGO-1 明显减少,证明AGO1 及 RNAPII 对H3K9 的双甲基化是必需的[37]。
图 2 siRNA、miRNA 及piRNA 的生物合成[26, 27, 58]
a: (人类)siRNA 来源于长的双链RNA 分子, 经Dicer 酶剪切为21~25 nt 的双链RNA 片段, Dicer 酶和dsRNA 结合蛋白将siRNA 二聚体装载至Argonaute 蛋白(AGO2)而发挥作用; b: (人类)miRNA 由内源性的生物体基因产生含有发卡结构的65~70 nt 长的pri-miRNA, 该发卡结构在细胞核内经Drosha-DGCR8 复合物加工产生pre-miRNA。在细胞浆内, pre-miRNA 进一步经Dicer 酶剪切为miRNA-miRNA*二聚体 (其中miRNA为引导链, miRNA* 为信息链), 装载至Argonaute 蛋白1(AGO1) 而发挥作用。C: (鼠类) piRNA 的生物合成尚不清楚。piRNA 来源于单链RNA前体, 而且不依赖于Dicer 酶。产生的初级正义piRNA 倾向于与 MILI 结合, 在出生前的睾丸、MILI 和MIWI2 均参与复制周期, 在次级反义piRNA 中MIWI2 比MILI 更为丰富, 次级反义piRNA 可能直接裂解转座子mRNA。
EZH2 作为组蛋白甲基转移酶, 在一定程度上能够引起H3K27 的甲基化, 而甲基化的H3K27 作为锚点可募集多余的PcG, 从而参与沉默态染色质的形成, 导致TGS[39]。染色体免疫沉淀结果表明:DNMTs 与EZH2 抑制的基因之间的结合依赖于EZH2 的存在; 亚硫酸氢盐修饰结合直接测序结果也证明EZH2 对于其靶向的启动子甲基化是必需的,提示EZH2 作为募集DNA 甲基转移酶的平台, 参与了DNA 的甲基化。研究报道, 在某些基因的靶标启动子内DNMT3a 能与小RNA发生免疫共沉淀, 表明DNMT3a 也参与了靶标启动子的TGS[36]。
总之, TGS 的建立与维持需要多种不同的蛋白:通常AGO1、DNMT3a 及HDAC-1 对于起始的沉默是必需的, 而DNMT1 对于维持沉默是必需的[34]。
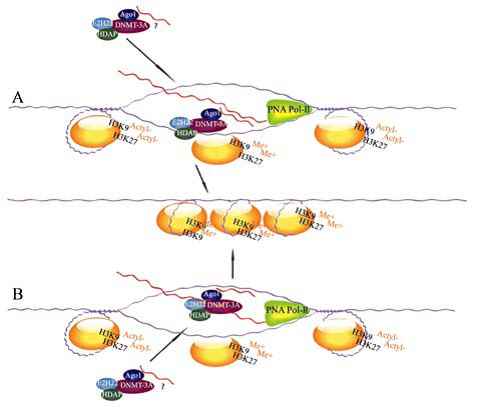
已知 RNAPII 参与了小RNA 介导的TGS, 研究报道RNAPII 与AGO1免疫共沉淀于启动子区域, 但尚不清楚RNAPII 是如何发挥作用的。目前研究主要集中为两种模型。第一种为RNA-RNA 模型(图3A): 其主要特征是RNA 引导链与RNAPII 合成的低拷贝转录子结合, 从而导致沉默。近来更多的研究支持这一模型, 其中一个关键的实验是应用对siRNA 敏感的INK4/ARF 位点的调节域(Regulatorydomain, RD)作为实验平台, 研究siRNA 介导的RD染色质重塑, 结果显示其重叠转录是靶向转录链而非模板链, 而且完全互补的双链RNA 前体及不完全互补的双链RNA 前体均可介导异染色质的形成[41]。另有研究结果表明经RNAPII 合成的低拷贝转录子,可被siRNA 的引导链识别, 并作为识别域引导沉默复合物至启动子区域导致TGS[29]。以互补的硫代磷酸酯寡核苷酸 (Phosphorothioate oligonucleotides,PS-ODNs) 封闭这些低拷贝转录子, 可终止siRNA靶向的启动子沉默。第二种为RNA-DNA 模型(图3B): 其主要特征是在转录过程中, RNA 引导链与靶标的一条DNA 形成二聚体, 从而导致沉默。支持该模型的直接证据包括: RNAPII 能与TATAA 转录起始点上游松解的核小体结合, 从而参与TGS[42], 以及DNMT3b 直接参与了siRNA 介导的TGS[30]。事实上, 这两种模型并非相互排斥, 可能是siRNA 参与了不同的功能过程, 当siRNA 直接靶向TATAA或RNPII 结合位点时, 以RNA-DNA 方式介导TGS,而大多数情况下siRNA 是以RNA-RNA 方式发挥作用。
图 3 RNA 基因沉默的作用模式(人细胞)[35]
A: RNA/RNA 模型: RNA 引导链与RISC(AGO1、Ezh2、DNMT3a 或HDAC-1)结合, 随后与RNAPII 合成的低拷贝转录子结合, 从而介导TGS。B: RNA/DNA 模型: RNA 引导链与RISC(AGO1、Ezh2、DNMT3a或HDAC-1)定位于基因组DNA, 随后与靶标的一条DNA 形成二聚体, 从而介导TGS。
2 miRNA
miRNA是长约21∼25 nt 的单链RNA, 其中50%定位于易发生结构改变的染色体区域[43]。最初认为miRNA 和siRNA 的区别主要有两点: (1) miRNA 是内源性的, 是生物体基因的表达产物; siRNA 是外源性的, 来源于病毒感染、转座子或转基因靶点。(2)miRNA 是由不完整的发卡状双链RNA, 经Drosha和Dicer 酶加工而成; siRNA 是由完全互补的长双链RNA, 经Dicer 酶剪切而成[44](图2b)。尽管如此区别,由于miRNA与siRNA密切相关, 如两者片段大小相近, 均经Dicer 加工后装至AGO 蛋白而发挥作用,因此推测miRNA也能介导TGS, 而且两者的作用机制有所重叠[22, 27, 40]。
目前研究表明: 哺乳动物细胞内miRNA的调控机制反映了miRNA 的特异装载蛋白AGO 以及miRNA 与mRNA 之间的互补程度[45]。通常只有极少数的miRNA 与其mRNA 靶标几乎完全互补, 从而可以经内切酶直接裂解mRNA, 而绝大多数miRNA 与其mRNA 靶标只有部分互补, 一般为6~7个碱基, 位于mRNA 的5′端的第2~7 位核苷酸, 称为“种子区” (Seed region), 在与靶标的结合中起重要作用, 因此种子区是筛选靶标的最基本的特异性的决定因素, 同时不少研究提示一个miRNA 可调控许多甚至上百个不同的基因[45~48]。Lewis 等[48]通过对13 000 多个人类基因的研究进一步推测: 组蛋白甲基化酶、甲基化CpG 结合蛋白、染色质域蛋白及组蛋白去乙酰化酶等均是miRNA 潜在的作用靶标。
已有研究表明miRNA 可通过调控组蛋白修饰引起染色质重塑。有学者报道组蛋白去乙酰化酶4(HDAC4)是鼠胚胎软骨组织特异的miR-140 的靶标, 提示miRNA 可通过调控组蛋白的修饰而参与TGS[49]。Kim 等[50]研究发现: 哺乳动物细胞基因组保守的miRNA 之一—— miR-320(由细胞周期基因POLR3D 启动子上游编码)能引导AGO1 至POLR3D启动子, 同时还有EZH2 及H3K27 三甲基化的参与,以顺式方式诱导TGS, 这进一步证实miRNA可在哺乳动物细胞内引起TGS。
近来的研究进一步证明miRNA 可通过调控DNA 甲基化酶的表达而影响DNA 甲基化。Fabbri等研究发现在非小细胞癌内, miR-29 家族(包括miR-29a、miR-29b 和miR-29c)的表达是下调的, 而DNMT3a、DNMT3b 处于高表达状态, miR-29 家族与DNMT3a、DNMT3b 的3′UTR 端的互补性提示DNMT 的mRNAs 是miR-29 家族的作用靶点。Benetti 等及Sinkkonen 等的研究均表明Dicer缺失鼠胚胎干细胞内, DNMT3a、DNMT3b 表达的下调是依赖于miR-29 家族, 其主要机制是: Dicer 酶的缺失使miR-29 家族表达下调, 而miR-29 家族可抑制视网膜母细胞瘤样蛋白2(Retinoblastoma-likeprotein 2, Rbl2) 的表达, 后者通过抑制DNMT3a、DNMT3b 的活性, 使DNA 甲基化缺失, 同时提示miR-29 通过增强抑癌基因的表达而抑制肿瘤的发生。Gonzalez 等[41]研究结果显示: 哺乳动物细胞的肿瘤miRNA—miR17-5p 及miR20a 能在具有重叠转录功能并与miRNA 种子区互补的启动子区域诱导异染色质的形成, 进一步揭示了miRNA 调控的染色质重塑和基因转录的新机制。上述研究将有助于我们更加全面真实地阐明肿瘤的发生机制, 为寻找肿瘤治疗的新靶点提供一个全新的视野。
3 piRNA
piRNA 是近年来在哺乳动物细胞内发现的长度约为24∼31 nt 的RNA 分子, 因在生理状态下能与Piwi 蛋白偶联, 故命名为piRNA(Piwi interactingRNA, piRNA)。由于Piwi 为一表观遗传学调控因子, 能与PcG 蛋白共同结合于基因组PcG 应答元件上, 协助PcG 沉默同源异型基因,因此推测与Piwi相关的piRNA 也应具有表观遗传学的调控作用。
研究表明哺乳动物细胞piRNA 分为两个亚簇[7~9]: 一是粗线期piRNA簇: 主要出现于减数分裂的粗线期, 持续表达至单倍体精子细胞阶段,一般很少有重复片段; 二是粗线前期piRNA 簇: 主要出现于减数分裂前的生殖细胞, 虽然具有粗线期piRNA 簇的分子特征, 但来源于一个完全不同的簇,与果蝇及斑马鱼的piRNA 相似, 具有重复片段。大多数piRNA 的5′端有单磷酸化基团而且偏爱尿嘧啶碱基(> 84%), 3′端有2′O –甲基修饰, 其生物学意义仍不清楚, 推测可能对piRNA 的稳定性及功能至关重要。
目前 piRNA 的生物合成和作用机制尚不清楚。鉴于piRNA 的作用不依赖于Dicer 酶, 哺乳动物细胞粗线期piRNA 簇具有明显的链不对称性, 提示piRNA 来源于单链前体, 而非双链分子。因此产生两种假说: 一是piRNA 由长单链分子产生; 二是piRNA 可能为初级转录产物。Aravin 等通过鼠细胞内的实验证据提出哺乳动物细胞piRNA 的自我扩增模型, 与果蝇扩增模型不同的是: 基因簇并非初级piRNA 的主要来源, 而是由转座子mRNA 产生初级正义piRNA 参与扩增循环(图2c)。
已有研究表明DNA 甲基酶家族(DNMT3a、DNMT3b 及DNMT3L)在转座子甲基化的形成中起主要作用, 其中DNMT3a 和DNMT3b 的催化活性在生殖细胞及体细胞内均非常重要, DNMT3L 则是生殖细胞内甲基化形成的一个核心调控子[6]。实验发现鼠的MILI 和MIWI2 蛋白对于其睾丸中LINE-1 和IAP 转座子成分的沉默是必需的, MILI 或MIWI2 的缺失可以使转座子甲基化标识丢失, 而且突变鼠的表型与DNMT3L 缺失鼠的表型一致; 在甲基化形成的关键时期, MIWI2 始终定位于细胞核内;小RNA 序列分析显示MILI 和MIWI2 均作用于DNMT3L 的上游, 随后作用于DNMT3a 和DNMT3b的上游。上述实验结果证实Piwi/piRNA 复合体能介导转座子甲基化的形成, 且Piwi 途径位于DNA 甲基化调节因子的上游, piRNA 是生殖细胞内DNA 甲基化的特异性决定子。目前在果蝇中的研究已表明Piwi 能通过直接募集染色质结构域蛋白HP1a而参与异染色质的形成(H3K9 甲基化), 但在哺乳动物细胞内尚无类似研究报道。
4 lncRNA
lncRNA一般是指大于200nt 的RNA, 不参与或很少参与蛋白编码功能, 位于细胞核内或胞浆内。通常分为5 类: 即正义(Sense)、反义(Antisense)、双向(Bidirectional)、基因内(Intronic)及基因间(Intergenic)lncRNA[18]。
lncRNA 有多种不同的来源, 目前认为可能是[18](1)编码蛋白的基因结构中断而转变为lncRNA;(2) 染色质重组的结果, 即两个未转录的基因与另一个独立的基因并列而产生含多个外显子的lncRNA;(3) 由非编码基因复制过程中的反移位产生; (4)由局部的串联复制子产生邻近的非编码RNA; (5)基因中插入一个转座成分而产生有功能的非编码RNA。尽管众多的lncRNA 没有共同的起源, 但研究表明它们在基因表达的调控方面起着相似的作用。
哺乳动物细胞内lncRNA 调控的表观遗传学研究, 最早源于基因组印记(Genomicimprinting) 和X染色体失活(X chromosome inactive), 分别与H19 和Xist RNA 密切相关。H19 作为基因组印记的、母源表达的lncRNA, 经过剪接及多聚腺苷酸化后输送至胞浆内, 并可累计达到较高浓度。虽然H19 是第一个发现的与基因组印记密切相关的基因, 但H19的功能至今尚不明确。近来有学者在人和鼠细胞中发现H19 RNA 是 miR-675 的前体, 提示H19 RNA
可能通过miRNA 发挥基因调控作用。基因组印记除了与H19 基因簇有关, 还有Kcnq1ot1、Air 及 Nespas基因的参与, 它们通常是父源表达的, 通过卵母细胞中启动子的DNA 甲基化抑制母系等位基因的表达。Kcnq1ot1 和 Air 启动子的缺失实验结果表明, lncRNA 或其转录产物分别对于Kcnq1ot1 和lgf2r/Air 印记基因簇的沉默是必需的, 而且对长达800bp 的印记基因簇抑制性组蛋白标记(H3K27 三甲基化和 H3K9 双甲基化)以及某些基因的DNA甲基化也是必需的。
Xist RNA(17 kb 长的非编码RNA)对于哺乳动物细胞内X 染色体失活非常重要, 但Xist 并不输送至胞浆, 而是与即将被它失活的X 染色体的某个RNA结构域或成分相连, 在失活染色体表面形成“外套”(Coating)并以顺式方式介导基因沉默。目前认为Xist RNA 在没有Pol II 的情况下, 建立了一个特别的核区, 大多数即将被失活的染色体在此定位并失活, 其基因沉默特征是失去激活性的组蛋白修饰(如乙酰化)而获得抑制性的组蛋白修饰(如H3K27的三甲基化), 最后, 失活染色体上的许多CpG 岛启动子甲基化。近来Zhao 等研究表明: RepA(XistRNA 上的一个1.6 kb 片段), 能通过募集Polycomb 抑制复合物2( Polycomb repressive complex 2, PRC2)并通过EZH2 靶向将被失活的染色体, 使染色体H3K27 发生三甲基化, RepA 缺失实验表明RepA 对完整的Xist RNA 及H3K27 的甲基化是非常重要的。
有意义的是, 即使在细胞分裂中期, 该“外套”始终以顺式方式与失活的染色体相连, 经多次细胞分裂后, X 染色体失活始终存在, 表明RNA“外套”与组蛋白修饰及DNA 甲基化共同构成表观遗传学的记忆, 从而使失活的染色体进行有丝分裂。但在体细胞, Xist 基因的缺失并不影响基因沉默, 提示一旦建立了记忆, lncRNA 诱导的沉默结构域是非必需的。
近来研究表明X 染色体失活和基因组印记可能共享一些基因簇, 其基因沉默的机制不仅包括顺式作用, 还有反式作用。例如HOX 转录反义RNA (Hoxtranscript antisense RNA, HOTAIR), 它来源于HOXC位点, 能以反式方式沉默HOXD 位点长达40 kb 的基因转录, 主要机制是通过HOTAIR 募集PRC2 至HOXD 位点, 使X 染色体和Kcnq1 域分别发生H3K9三甲基化, 从而引发异染色质的形成及TGS。另有研究报道: X 染色体失活尚存在另一机制, 即Xist 和Tsix 复性形成RNA 二聚体, 经Dicer酶剪切成为小干扰RNA, 其对于失活的X 染色体上异染色质的修饰是必需的。这两个不同的途径或许可以协调 lncRNA 和小RNA 在染色质重塑方面的作用, 同时提示RNA 调控存在着一个更为复杂的、交互的网络。
参考文献(References):
[1] Egger G, Liang G, Aparicio A, Jones PA. Epigenetics inhuman disease and prospects for epigenetic therapy. Nature,2004, 429(6990): 457–463.
[2] Bird A. Perceptions of epigenetics. Nature, 2007,447(7143): 396–398.
[3] Bird A. DNA methylation patterns and epigenetic memory.Genes Dev, 2002, 16(1): 6–21.
[4] Goll MG, Bestor TH. Eukaryotic cytosine methyltransferases.Annu Rev Biochem, 2005, 74: 481–514.
[5] Weber M, Hellmann I, Stadler MB, Ramos L, Pääbo S,Rebhan M, Schübeler D. Distribution, silencing potential and evolutionary impact of promoter DNA methylation in the human genome. Nat Genet, 2007, 39(4): 457–466.
[6] Okano M, Bell DW, Haber DA, Li E. DNA methyltransferases Dnmt3a and Dnmt3b are essential for de novo methylation and mammalian development. Cell, 1999, 99(3):247–257.
[7] Goldberg AD, Allis CD, Bernstein E. Epigenetics: a landscape takes shape. Cell, 2007, 128(4): 635–638.
[8] Bernstein BE, Meissner A, Lander ES. The mammalian epigenome. Cell, 2007, 128(4): 669–681.
[9] Shelley L. Berger. The complex language of chromatin regulation during transcription. Nature, 2007, 447(7143):407–412.
[10] Jenuwein T, Allis CD. Translating the histone code. Science,2001, 293(5532): 1074–1080.
[11] Kouzarides T. Chromatin modifications and their function.Cell, 2007, 128(4): 693–705.
[12] Bühler M, Moazed D. Transcription and RNAi in heterochromatic gene silencing. Nat Struct Mol Biol, 2007,14(11): 1041–1048.
[13] Cao R, Zhang Y. The functions of E(Z)/EZH2-mediatedmethylation of lysine 27 in histone H3. Curr Opin GenetDev, 2004, 14(2): 155–164.
[14] Grewal SI, Elgin SC. Transcription and RNA interference in the formation of heterochromatin. Nature, 2007,447(7143): 399–406.
[15] Mellor J, Dudek P, Clynes D. A glimpse into the epigenetic landscape of gene regulation. Curr Opin Genet Dev,2008, 18(2): 116–122.
[16] Tchurikov NA. Molecular mechanisms of epigenetics. Biochemistry (Mosc), 2005, 70(4): 406–423.
[17] Reik W. Stability and flexibility of epigenetic gene regulation in mammalian development. Nature, 2007, 447(7143):425–432.
[18] Ponting CP, Oliver PL, Reik W. Evolution and functions of long noncoding RNAs. Cell, 2009, 136(4): 629–641.
[19] Zaratiegui M, Irvine DV, Martienssen RA. Noncoding RNAs and gene silencing. Cell, 2007, 128(4): 763–776.
[20] Mattick JS, Makunin IV. Non-coding RNA. Hum Mol Genet, 2006, 15(1): R17–29.
[21] Amaral PP, Mattick JS. Noncoding RNA in development. Mamm Genome, 2008, 19(7−8): 454–492.
[22] Ghildiyal M, Zamore PD. Small silencing RNAs: an expanding universe. Nat Rev Genet, 2009, 10(2): 94–108.
[23] Bernstein E, Allis CD. RNA meets chromatin. Genes Dev, 2005, 19(14): 1635–1655.
[24] Amaral PP, Dinger ME, Mercer TR, Mattick JS. The eukaryotic genome as an RNA machine. Science, 2008, 319(5871): 1787–1789.
[25] Costa FF. Non-coding RNAs, epigenetics and complexity. Gene, 2008, 410(1): 9–17.
[26] Jinek M, Doudna JA. A three-dimensional view of the molecular machinery of RNA interference. Nature, 2009,457(7228): 405–412.
[27] Moazed D. Small RNAs in transcriptional gene silencing and genome defense Nature, 2009, 457(7228): 413–420.
[28] Bayne EH, Allshire RC. RNA-directed transcriptional gene silencing in mammals. Trends Genet, 2005, 21(7):370–373.
[29] Han J, Kim D, Morris KV. Promoter-associated RNA is required for RNA-directed transcriptional gene silencingin human cells. Proc Natl Acad Sci USA, 2007, 104(30):12422–12427.
[30] Kawasaki H, Taira K. Induction of DNA methylation and gene silencing by short interfering RNAs in human cells.Nature, 2004, 431(7005): 211–217.
[31] Morris KV, Chan SW, Jacobsen SE, Looney DJ. Small interferingRNA-induced transcriptional gene silencing inhuman cells. Science, 2004, 305(5688): 1289–1292.
[32] Kawasaki H, Taira K, Morris KV. siRNA induced transcriptional gene silencing in mammalian cells. Cell Cycle,2005, 4(3):442–448.
[33] Ting AH, Schuebel KE, Herman JG, Baylin SB. Short double-stranded RNA induces transcriptional genesilencing in human cancer cells in the absence of DNA methylation. Nature Genet, 2005, 37(8): 906–910.
[34] Hawkins PG, Santoso S, Adams C, Anest V, Morris KV. Promoter targeted small RNAs induce long-term transcriptional gene silencing in human cells. Nucleic Acids Res, 2009, 37(9): 2984–2995.
[35] Hawkins PG, Morris KV. RNA and transcriptional modulation of gene expression. Cell Cycle, 2008, 7(5): 602–607.
[36] Weinberg MS, Villeneuve LM, Ehsani A, Amarzguioui M, Aagaard L, Chen ZX, Riggs AD, Rossi JJ, Morris KV. The antisense strand of small interfering RNAs directs histone methylation and transcriptional gene silencing in human cells. RNA, 2006, 12(2): 256–262.
[37] Kim DH, Villeneuve LM, Morris KV, Rossi JJ. Argonaute-1 directs siRNA-mediated transcriptional gene silencing in human cells. Nat Struct Mol Biol, 2006, 13(9): 793–797.
[38] Janowski BA, Huffman KE, Schwartz JC, Ram R, Nordsell R,Shames DS, Minna JD, Corey DR. Involvement ofAGO1 and AGO2 in mammalian transcriptional silencing. Nat Struct Mol Biol, 2006, 13(9): 787–792.
[39] Viré E, Brenner C, Deplus R, Blanchon L, Fraga M, Didelot C, Morey L, Van Eynde A, Bernard D, Vanderwinden JM, Bollen M, Esteller M, Di Croce L, de Launoit Y, Fuks F. The Polycomb group protein EZH2 directly controls DNA methylation. Nature, 2006, 439(7078): 871–874.
[40] Siomi H, Siomi MC. On the road to reading the RNA- interference code. Nature, 2009, 457(7228): 396–404.
[41] Gonzalez S, Pisano DG, Serrano M. Mechanistic principlesof chromatin remodeling guided by siRNAs andmiRNAs. Cell Cycle, 2008, 7(16): 2601–2608;
[42] Boeger H, Bushnell DA, Davis R, Griesenbeck J, Lorch Y, Strattan JS, Westover KD, Kornberg RD. Structural basisof eukaryotic gene transcription. FEBS Lett, 2005, 579(4):899–903.
[43] Ruvkun G. Molecular biology. Glimpses of a tiny RNA world. Science, 2001, 294(5543): 797–799.
[44] Carthew RW, Sontheimer EJ. Origins and mechanisms of miRNAs and siRNAs. Cell, 2009, 136(4): 642–655.
[45] Bartel DP. MicroRNAs: Target recognition and regulatory functions. Cell, 2009, 136(4): 215–233.
[46] Rajewsky N. MicroRNA target predictions in animals. Nat Genet, 2006, 38: S8–13.
[47] Lim LP, Lau NC, Garrett-Engele P, Grimson A, Schelter JM, Castle J, Bartel DP, Linsley PS, Johnson JM. Microarray analysis shows that some microRNAs downregulate large numbers of target mRNAs. Nature, 2005, 433 (7027): 769–773.
[48] Lewis BP, Burge CB, Bartel DP. Conserved seed pairing, flanked by adenosines, indicates that thousands of human genes are microRNA targets. Cell, 2005, 120(1): 15–20.
[49] Tuddenham L, Wheeler G, Ntounia-Fousara S, Waters J, Hajihosseini MK, Clark I, Dalmay T. The cartilage specificmicroRNA-140 targets histone deacetylase 4 in mouse cells. FEBS Lett, 2006, 580(17): 4214–4217.
[50] Kim DH, Saetrom P, Snøve O Jr, Rossi JJ. MicroRNAdirected transcriptional gene silencing in mammalian cells.Proc Natl Acad Sci USA, 2008, 105(42): 16230–16235.