small RNA是生物体内一类重要的功能分子,包括microRNA. siRNA和piRNA。它的主要功能是诱导基因沉默,调控细胞生长、发育、基因转录和翻译等生物学过程。基于新一代高通量测序技术的small RNA测序,可一次性获得数百万条small RNA序列,能够快速地鉴定某种组织在特定状态下的所有已知small RNA并发现新的small RNA,为small RNA功能研究提供有力工具。
一、研究内容
1.测序和基本数据处理
对总RNA(或small RNA)样品进行检测,检测合格后进行small RNA样品制备,经过下A克隆检测合格之后上机测序。
基本数据处理包括:图象识别、碱基识别、过滤接头序列和污染序列、去除低质量序列。
2.高级生物信息分析内容
- small RNA长度的分布统计;
- 将small RNA定位到基因组,探索其在全基因组的分布特征;
- 两个样本的公共序列和特有序列分析;
- 与microRNA数据库(miRBase 14.0)进行比对,鉴定已知microRNA;
- 通过与Rfam 9.1数据库以及GenBank数据库比对,鉴定rRNA、tRNA、snRNA等降解片段;
- 鉴定与重复序列相关的small RNA;
- 鉴定mRNA降解片段;
- small RNA分类注释;
- 预测新的microRNA;
- 单个样本的microRNA表达模式分析;
- 两个样本的microRNA差异表达分析;
- microRNA的表达模式聚类分析;
- MicroRNA靶基因预测。
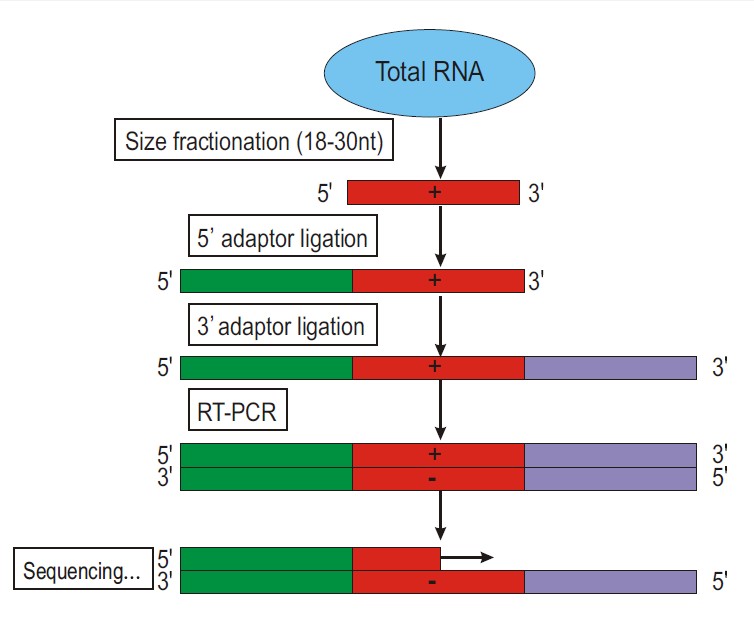
二、技术路线
图1:small RNA测序分析的实验流程
三、技术特点
- 高通量:一次测序得到300万条以上的序列;
- 不依赖已知信息:既能鉴定已知microRNA,又能发现新的microRNA;
- 高分辨率:可以检测microRNA单个碱基的差异;
- 高精确度:精确计数表达量为几个到数十万个拷贝的small RN氏
- 重复性好:深度测序保证了抽样随机性,可靠性高,不需重复实验。
四、研究案例
取某动物组织,提取其20 ug总RNA,进行样品制备和新一代高通量测序。
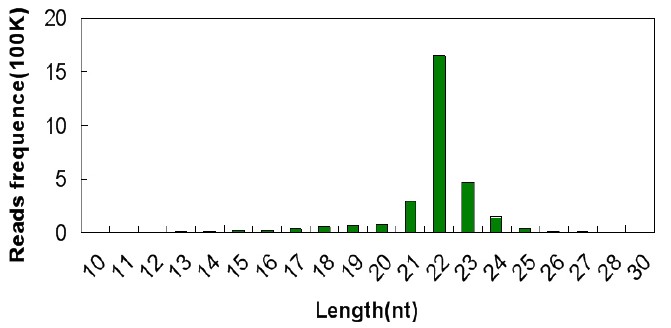
1. small RNA长度分布统计
对获得序列的分子长度和数量进行统计分析,结果显示出该动物small RNA的特征分布,大量分子长度集中在22 nt附近(图2)。长度分布统计可用于验证实验的可靠性。
图2:small RNA序列长度分布统计
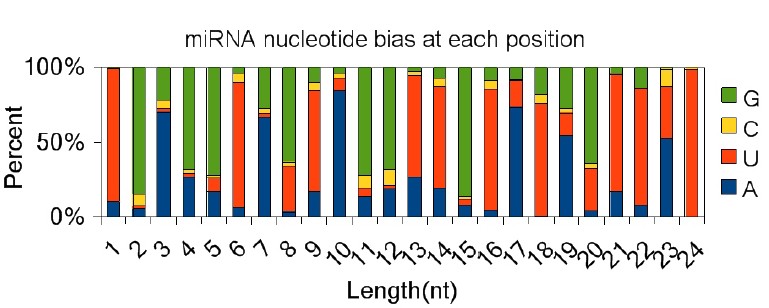
2. microRNA碱基偏好性鉴定
microRNA的5’端第一个碱基对U有很强的偏好性,对G则有抗性;第二到第四个碱基缺乏比其他位置碱基通常缺乏C(第四个碱基有时例外)。统计microRNA碱基使用偏好是检验数据质量的依据(图3)。
图3:已知microRNA的碱基使用偏好
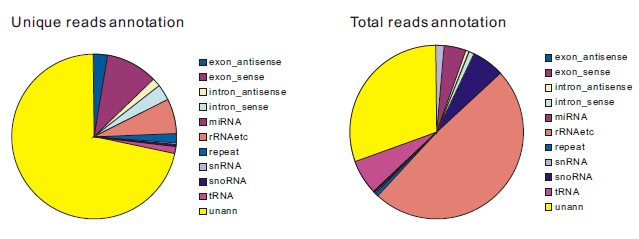
3.与数据库进行比对注释small RNA
将得到的序列分别与miRBase数据库、mRNA/EST数据库、rRNAetc等数据库进行序列比对,鉴定出已知的microRNA(图4)。
图4:microRNA及其它不同种类的RNA分子所占比例统计
4.鉴定与重复序列相关的small RNA
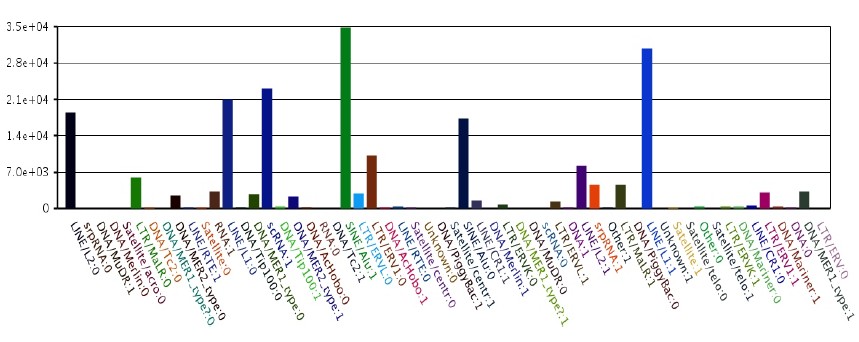
研究发现,部分small RNA来源于高重复区域或转座子区,它们会与不同的Argonaute蛋白结合并且参与一些重要的生物学过程,例如DNA甲基化和转座子的调控。这些small RNA被称作重复序列相关的small RNA (repeat-associated small RNAs)。根据结合的Argonaute蛋白不同,它们可以被细分为不同的类型 (图5)。
图5:样品中重复序列相关的small RNA的分布情况
5.预测新的microRNA
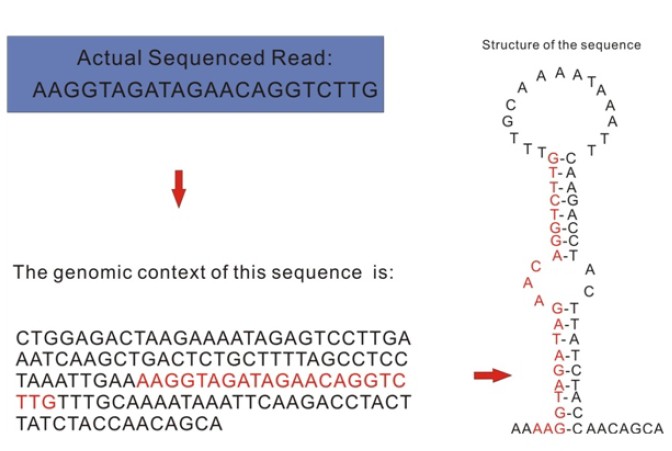
MicroRNA前体的标志性发夹结构能够用来预测新的microRNA。将测序得到的序列比对到该物种的基因组后,截取附近区域的一段序列,通过折叠模型分析,如发现该序列位于茎环结构上,则初步判定该序列为一个候选的新microRNA(图6)。
图6:新microRNA的预测
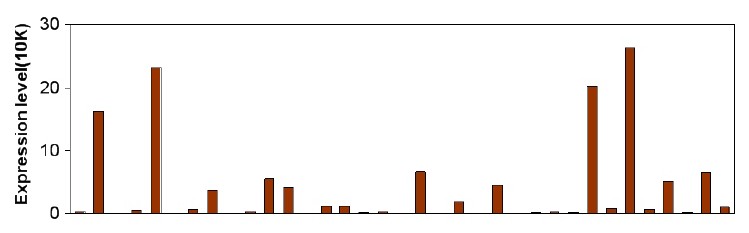
6. MicroRNA表达模式分析
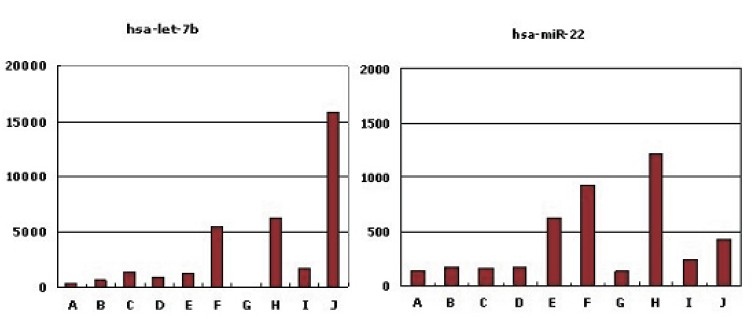
根据研究需要,选择目标microRNA分子进行表达模式分析。同一组织中的microRNA表达谱见图7;不同组织中microRNA表达模式变化见图5-8,图5-8反映了microRNA表达的组织特异性。
图7:部分microRNA在同一组织中的表达谱
图8:两种microRNA在不同组织中的表达谱 hsa-let-7b和hsa-miR-22为两种microRNA, A-J是10种组织
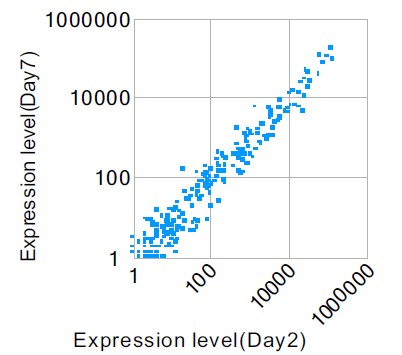
7.两个样本的microRNA差异表达分析
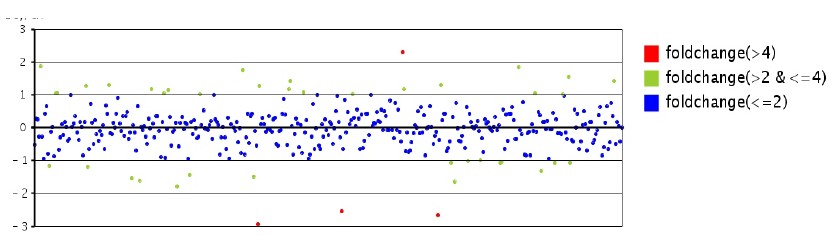
我们利用数学统计的方法对不同样品间的microRNA进行差异显著性分析,图5-9中游离的散点、图5-10中绿点和红点是有显著表达差异的microRNA。
图9:两种样品间microRNA的差异表达分析
图10两个样品间microRNA差异的强度分析。差异强度小于2倍的为蓝色点,2-4倍的为绿色点,大于4倍的为红色点
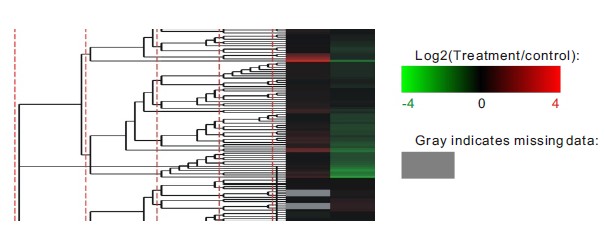
8. MicroRNA的表达模式聚类分析
表达模式相似的microRNA通常提示功能的相关性。表达模式聚类分析把表达模式相似的microRNA聚到一起,便于筛选出感兴趣的类别(图11)。
图11 microRNA聚类分析。红色表示上调,绿色表示下调,灰色表示在某个样品中未表达的microRNA
五、常见问题
1、small RNA测序对样品的要求
答:请提供浓度>750 ng/uI、质量>20ug的总RNA样品,提取总RNA时请避免使用过柱法,以防止small RNA丢失。我们会使用Agilent 2100检测仪和电泳法等方法对总RNA样品的各项指标进行检测,我们建议您在送样品前检测OD值及用电泳方法等对样品进行初步检测。
2、实验中,为什么需要做TA克隆
答:下A克隆是在建库实验后一个用于质控的抽样检测步骤。我们会随机抽取80条或者更多的片段用Sanger法进行测序,初步检测小分子的片段长度和种类分布情况,并和数据库进行比对鉴定物种来源并确定样品是否污染,下A克隆能够直观的反映样品和建库的质量。
3、实验中,为什么会有讲解的mRNA
答:由于总RNA常发生轻微的降解,并且生物体内也有自然的降解过程,因此数据中就会含有小部分mRNA降解片段。通常这个比例很低,并且取决于样品总RNA的质量。
4、为什么说原始数据中含有3' 接头序列和5' 接头污染是正常的?
答:新一代高通量测序产生的序列长度为35 nt,而small RNA序列长度是18~30 nt,所以测序得到的序列上有一段3’接头序列。污染指的是5’接头分子,由于加接头时接头分子都是过量加入的,因此会有空载现象,结果造成数据中含有少量的5’接头序列污染。通常污染的比例会低于5%,属于正常现象。
5、请问small RNA测序除了样品以外还需要提交哪些相关的信息?
答:需要提供相应物种的基因组和相关的exon. intron. repeat信息。如果没有本物种的基因组,需要提供近缘物种的相关信息。
6、我没有生物信息学基础,怎样能够读懂庞大的数据分析结果?庞大的结果文件我的电脑能否打开?
答:我们信息分析的所有数据和统计结果都以Excel表格的形式提供,并附有相应的图片,以及对每项分析内容的分析方法的详细描述。我们推荐使用UntraEdi十或EditPlus打开结果文件。
六、参考文献
1 .Chen X, Li QB, Wang J, et al. Identification and characterization of novel amphioxus microRNAs by Solexa sequencing. Genome Biology. 2009. 10(7): R78 (1-13).
2. Chen X, Ba Y, Ma LJ, et al. Characterization of microRNAs in serum: a novel class of biomarkers for diagnosis of cancer and other diseases. Cell Research. 2008. 18: 997-1006.
3. Mi S, Cai T, HuY, ChenY et al. Sorting of Small RNAs into ArabidopsisArgonaute Complexes Is Directed by the 5' Terminal Nucleotide. Cell. 2008. 133(1):116-127.
4. Montgomery TA, Howell MD, et al. Specificity ofARGONAUTE7-miR390 Interaction and Dual Functionality in TAS3Trans-Acting siRNA Formation. Cell. 2008. 133(1):128-141.
5. Morin RD, O'Connor MD, et al. Application of massively parallel sequencing to microRNAprofiling and discovery in human embryonic stem cells. Genome Res. 2008. 18: 610-621.
来源:华大基因ncRNA服务的宣传手册