在第二代测序技术的协助下,个人基因组图谱正在如火如荼地绘制中。但第二代测序技术很快就遇上了强劲的对手——第三代测序技术,也被称为“下、下一代的测序(next-next-generation sequencing)”。第三代测序技术是基于纳米孔(nanopore)的单分子读取技术,有着更快的数据读取速度,应用潜能也势必超越测序。
2月5日,基因组科学家们齐聚美国佛罗里达州的基因组生物和技术进展会议,来了解哪家公司的第三代测序技术能实现人类基因组的3分钟测序或以5000美元的价格出售。尽管科学家们对公布的数据表示谨慎乐观,但他们对于此类测序仪的优越之处仍心存疑虑。
Complete Genomics
在2008年10月,美国加利福尼亚州的Complete Genomics公司曾宣称他们将在2009年以5000美元的价格售卖人类基因组,但当时没有公布支持数据。在这次会议上,该公司公布了一个人类基因组,据称是用9台仪器在8天内完成的。
该公司的CEO,Clifford Reid表示,他们将254GB的数据拼接成草图,覆盖某个匿名男性基因组的92%,每个碱基平均读取了91次。与目前应用中的高速测序,即第二代测序类似,Complete Genomics也产生短的DNA读长。通过对每个碱基的多次测序,它的目标是排除悄悄混入的可能错误。Reid认为这项技术非常准确,碱基错误的概率低于0.33%。这与目前的测序仪相当。
Complete Genomics并不出售测序仪,但用自己的测序仪来完成所有的内部工作。这让某些科学家质疑,但另一些却深受鼓舞。
速度和费用成为Complete Genomics的最大卖点。该公司并没有透露基因组测序的确切费用,但据称每个基因组的原材料费用低至1000美元。它的目标是在上个月推出市场,今年对1000个基因组进行测序,明年测序数量达到20000个。
Pacific Biosciences
在Complete Genomics做报告前的一小时,Pacific Biosciences的首席技术官Stephen Turner展示了大肠杆菌的完整基因组,并称每个碱基的平均读取了38次,准确率大于99.9999%。
Pacific Biosciences利用了单分子技术和DNA聚合酶,在反应的同时读取测序产物。尽管目前仪器的读取速度仅为3 碱基/秒,但它的目标是在2013年前实现三分钟读完人类基因组。它还有望实现更长的读长。Tuner表示大肠杆菌基因组的平均读长是586 bp,有些能达到2805 bp。某些科学家期望长读长能排除错误,让他们了解到难以读取的部分。
Pacific Biosciences打算在明年正式推向市场。同时,目前的测序技术,如Illumina、Applied Biosystems和Roche也正以惊人的速度制造数据,在单次几天的运行中产生相当于多个人类基因组的数据。速率不断增长的同时,费用也在下降。例如,Illumina在会议中表示它在今年年底能实现10000美元的人类基因组测序。
要实现单分子实时测序,有三个关键的技术。
第一个是荧光标记的脱氧核苷酸。显微镜现在再厉害,也不可能真的实时看到“单分子”。但是它可以实时记录荧光的强度变化。当荧光标记的脱氧核苷酸被掺入DNA链的时候,它的荧光就同时能在DNA链上探测到。当它与DNA链形成化学键的时候,它的荧光基团就被DNA聚合酶切除,荧光消失。这种荧光标记的脱氧核苷酸不会影响DNA聚合酶的活性,并且在荧光被切除之后,合成的DNA链和天然的DNA链完全一样。
第二个是纳米微孔。因为在显微镜实时记录DNA链上的荧光的时候,DNA链周围的众多的荧光标记的脱氧核苷酸形成了非常强大的荧光背景。这种强大的荧光背景使单分子的荧光探测成为不可能。Pacific Biosciences公司发明了一种直径只有几十纳米的纳米孔[zero-mode waveguides (ZMWs)],单分子的DNA聚合酶被固定在这个孔内。在这么小的孔内,DNA链周围的荧光标记的脱氧核苷酸有限,而且由于A,T,C,G这四种荧光标记的脱氧核苷酸非常快速地从外面进入到孔内又出去,它们形成了非常稳定的背景荧光信号。而当某一种荧光标记的脱氧核苷酸被掺入到DNA链时,这种特定颜色的荧光会持续一小段时间,直到新的化学键形成,荧光基团被DNA聚合酶切除为止(见图)。
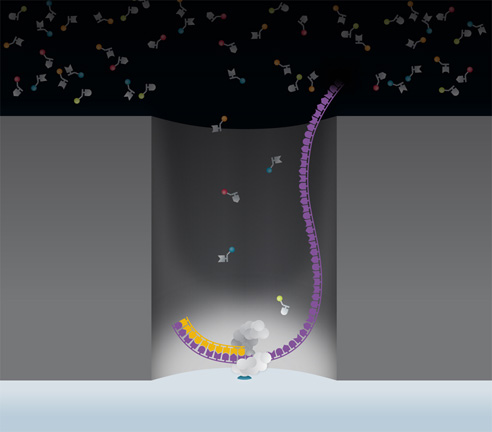
第三个是共聚焦显微镜实时地快速地对集成在板上的无数的纳米小孔同时进行记录。由于我对显微原理的物理知识匮乏,而Pacific Biosciences公司又没有非常强调在这方面的发明,不做进一步探讨。
他们还对这一技术进行进一步的优化。
第一个是把双链DNA环化反复测序。人们可以在双链DNA的两头连上发夹结构的DNA adaptor,从而使DNA环化。而DNA聚合酶就能够以环化的DNA作为模板滚环复制,反复测一段DNA序列。这种反复测序,纠正了偶尔出现的复制错误,从而使测序精度非常高。
第二个是激发光中断测序法。DNA聚合酶虽然很稳定,但是在强大的激发光作用下酶也是有一定寿命的。如果把激发光中断一段时间,在这段时间内DNA聚合酶继续复制DNA,当激发光重新开启以后,人们就可以测到长DNA链后面的序列。
第三代测序技术非常可怕。1、它实现了DNA聚合酶内在自身的反应速度,一秒可以测10个碱基,测序速度是化学法测序的2万倍。2、它实现了DNA聚合酶内在自身的processivity(延续性,也就是DNA聚合酶一次可以合成很长的片段),一个反应就可以测非常长的序列。 二代测序现在可以测到上百个碱基,但是三代测序现在就可以测几千个碱基。这为基因组的重复序列的拼接提供了非常好的条件。3、它的精度非常高,达到99.9999%。
此外,它还有两个应用是二代测序所不具备的。
第一个是直接测RNA的序列。既然DNA聚合酶能够实时观测,那么以RNA为模板复制DNA的逆转录酶也同样可以。RNA的直接测序,将大大降低体外逆转录产生的系统误差。
第二个是直接测甲基化的DNA序列。实际上DNA聚合酶复制A、T、C、G的速度是不一样的。正常的C或者甲基化的C为模板,DNA聚合酶停顿的时间不同。根据这个不同的时间,可以判断模板的C是否甲基化。
Pacific Biosciences公司预计2010年或者2011年就会推出商业化的测序仪器。在不远的将来,如果他们能和二代测序一样集成100万个纳米微孔,那么一台仪器15分钟就能够准确地测出一个人的基因组。以后每个人的基因组测序成本将变成100美元,人人都可以消费得起。想想人类基因组计划耗资30亿美元,费时十几年,无数科学家参与其中,技术的革新意义是多么重大啊!
Helicos Biosciences
并不是每家测序公司都这么幸运。Helicos Biosciences制造的第三代测序仪就被测序错误所困扰。就在会议前几天,Helicos透露它的第一名顾客已经将测序仪退还。在这次会议上,该公司表示它已经拼接了线虫的基因组。但是它的历史问题和高昂的仪器费用,即使降低至99.9999万美元,与其他测序仪的50万美元左右的价格相比,仍然让许多科学家望而却步。
在会议前的研讨会上,来自多伦多安大略癌症研究所的John McPherson说出了大家的心声:“Helicos是单分子测序的先锋,但我认为他们还没有达到预定的目标。”但Helicos的首席技术官William Efcavitch却不认同这种说法,“关于我们不行的传闻太夸张了”。
许多科学家希望他是对的,他们期待着Helicos与其他公司继续竞争,以更低的价格获得更多的数据。一位科学家表示:“这种竞争是良性的。无论这些公司干得多好,我们都期望更多。”
基于纳米孔的单分子读取技术,英国牛津纳米孔公司成功研发出第三代基因测序技术。该测序技术读取数据更快、有望大大降低测序成本,改变个人医疗的前景。
当前,基因测序工作费时且昂贵,测序时,分子必须进行多次复制(这一步被称为扩增),同时进行荧光示踪标记,这一过程会带来错误,因此,一个基因要被测序多次才能得到值得信赖的结果。此外,购买和操作测序仪器的费用也不菲,目前,测定一个完整的基因组需要上万美元。
在纳米孔测序技术中,DNA分子依靠被称为核酸外切酶的蛋白质以一次一个碱基的速度通过小孔。这个酶能清楚地区分出4个DNA碱基编码:A、C、G、T,也可以检测出该碱基是否被甲基化,一个单孔能在大约70天左右测定一个完整的基因序列。
纳米孔技术不需要荧光标记物并且很可能不需要进行扩增,能直接并快速“读”出DNA,同时足够廉价,使进行大量重复实验成为可能。
纳米孔公司已经研发出包含几百个纳米孔的芯片,该芯片可以用在一台机器上,快速且廉价地给大量DNA进行排序。
基因测序于上世纪70年代由弗雷德·桑格尔发明,他因此获得了诺贝尔奖,第一份人类基因草图于2001年绘制成功,花费了40亿美元。
纳米孔公司总裁戈登·桑赫纳说,该技术预示了基因测序领域的一个跳跃变化,花费不到1000美元就可以完成一个基因测序。借助该技术,在未来5年内,测序费用将有可能降至500美元。到那时,基因测序可以成为英国国民健康保险制度的一部分,民营保险公司也支付得起。该技术也可以让医生使用DNA来预测并且预防诸如心脏病、糖尿病等疾病,更加有效地开药。
世界著名基因测序公司Illumina的总裁杰伊·弗拉特利称,10年后,每一个新生婴儿都会被“配备”完整的基因排序,费用不超过5000美元。
最早的Sanger测序在人类基因组计划中立下赫赫战功,但也给基因组测序贴上了数亿美元的价格标签,让人生畏。这两年发展迅猛的第二代测序仪——Illumina的Genome Analyzer、Roche 454的GS系列以及ABI的SOLiD系统——让人类基因组重测序的费用蹭地降低到10万美元以下。现在,能对单个DNA分子进行测序的第三代测序仪也加入到这场比赛中,让竞争更加激烈。
目前,第三代测序主要有三种技术平台。两种通过掺入并检测荧光标记的核苷酸,来实现单分子测序。Helicos的遗传分析系统已上市,而Pacific Biosciences准备在明年推出单分子实时(SMRT)技术。第三种Oxford Nanopore的纳米孔(nanopore)测序还尚未有推出的时间表,但有可能是这三种当中最便宜的。纳米孔测序的优势在于它不需要对DNA进行标记,也就省去了昂贵的荧光试剂和CCD照相机。
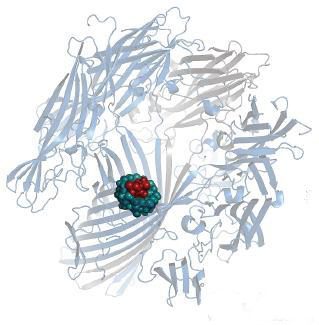
最近,Oxford Nanopore Technologies的Hagan Bayley及他的研究小组正致力于改善纳米孔。根据他们之前的工作,他们以a-溶血素来设计纳米孔,并将环式糊精共价结合在孔的内侧(下图)。当核酸外切酶消化单链DNA后,单个碱基落入孔中,它们瞬间与环式糊精相互作用,并阻碍了穿过孔中的电流。每个碱基ATGC以及甲基胞嘧啶都有自己特有的电流振幅,因此很容易转化成DNA序列。每个碱基也有特有的平均停留时间,它的解离速率常数是电压依赖的,+180 mV的电位能确保碱基从孔的另一侧离开。
a-溶血素纳米孔(剖面图)以及共价结合的环式糊精(浅蓝色)瞬间结合落入孔中的碱基(红色)。
以往对甲基胞嘧啶进行测序,都要先进行重亚硫酸盐转化,而纳米孔技术能直接读出这第五种碱基。这对表观基因组测序的研究人员来说可谓是个好消息。
纳米孔测序预计能满足大部分测序用户的需求:99.8%的准确性相当高,且错误很容易通过计算来纠正。均聚物延伸也没有问题,因为纳米孔记录每一个碱基,而不管其前后的碱基。读长也会很长。Bayley认为:“它有可能读取数千个碱基,序列质量也不会下降。即使中途有一些小差错,它也可以重新开始。”
但是,Oxford Nanopore的测序仪仍面临两个重要的技术问题。一是如何将核酸外切酶更好地附着在孔上,让它每次只掉入一个碱基,这是一个大挑战。另一个是并行化。这个问题可能简单一些。他们可以开发出一个芯片,上面有数万个孔,来确保整个测序过程更快速。
在纳米孔测序技术的推动下,实现千元基因组的目标指日可待了。
参考文献:
Clarke, J. et al. Continuous base identification for single-molecule nanopore DNA sequencing. Nat. Nanotechnol. advance online publication 22 February, 2009.
本文来自:http://blog.renren.com/share/222909213/1804282689



来自外部的引用