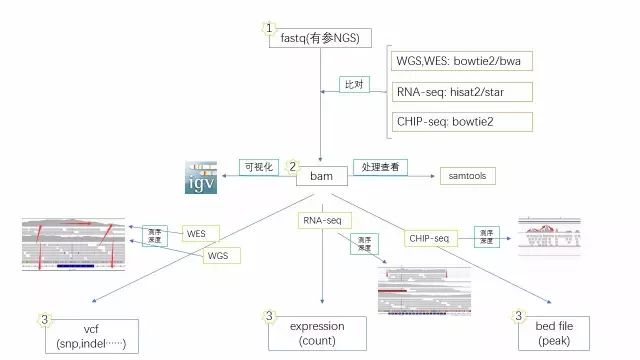
全外显子(Whole-exome sequencing)测序是啥?转录组(RNA-seq)测序是啥?ChIP-seq又是啥?它们之间有什么差别么?傻傻分不清,不用怕,多学习下就会了,下面让我们一起来从平均测序深度和区域覆盖度的角度来区分它们吧!
1 基础概念
平均测序深度:
指定区域内得到的所有碱基数目与该区域的长度的比值,如果是全基因组,就是整个测序的碱基数目除以基因组的大小。比如人类的基因组大小是3G(30亿个碱基),我的全基因组测序共8.9亿条150bp的reads,那么全基因组范围的平均测序深度就是8.9亿*150/30亿~45X,这个概念很重要!
覆盖度:
指测序获得的序列占整个基因组(或者指定区域)的比例。由于基因组中的高GC、重复序列等复杂结构的存在,测序最终拼接组装获得的序列往往无法覆盖有所的区域,这部分没有获得的区域就称为Gap。例如一个细菌基因组测序,覆盖度是98 %,那么还有2%的序列区域是没有通过测序获得的。
(你想实际测到的占你想测的区域比例。)
由于我们研究目的不一样,通常我们不需要覆盖到全基因组,所以就有了各种针对性的组学技术,也就是我们需要明白的!
2 理概念
理解了上面的测序深度和覆盖度的概念,我们就可以根据它们来区分WGS,WES,RNA-seq组与ChIP-seq,简单地说就是搞清楚这些组学要测什么,而且测多深即可。
全外显子(Whole-exome sequencing):
首先外显子组(Exome)是指真核生物基因组中全部外显子区域的总和,包含了蛋白质合成最直接的信息。外显子 组测序(Exome-seq)是利用设计好的探针试剂盒将坐标已知的全基因组外显子区域的DNA捕捉并富集后,进行高通量测序的基因组分析方法。 对于人类基因组来说,外显子区域大概占到基因组的1%,大概在30M左右。
一般全外显子测序的测序深度为50X~200X,具体深度依研究目的而定,其个体之间的变异小(在VCF文件上记录着少许差异,一点点)。
转录组测序(RNA-seq):
首先转录组是指在相同环境(或生理条件)下的在一个细胞、或一群细胞中所能转录出的所有RNA的总和,包括信使RNA(mRNA)、核糖体RNA(rRNA)、转运RNA(tRNA)及非编码RNA。转录组测序(RNA-seq)是将提取所要研究的特定类型的RNA,将其反转录成cDNA,利用高通量测序技术获得某一物种特定组织或器官在某一状态下的几乎所有转录本序列信息。对于已知参考基因组的物种,所获得大部分序列是已知的,同时会有一些新的转录本会被检测到,几乎可以忽略;甚至处于不同状态的人,其转录组数据有所不同。因此其主要的研究点——研究随着时空的变化、组织的变化、样本的变化,转录本发生改变。
染色质免疫共沉淀测序(ChIP-seq):
主要用于蛋白质与DNA相互作用研究,采用特异抗体对目的蛋白进行免疫沉淀,分离与目的蛋白结合的基因组DNA片段,对其进行纯化和文库构建,再通过高通量测序的方法,在全基因组范围内寻找目的蛋白的DNA结合位点,从而获得全基因组范围内与组蛋白、转录因子等互作的DNA片段信息。(与外显子测序不一样,不是通过设计好的探针来捕获序列的,而是通过特异的RNApoly酶、组蛋白、转录因子来捕获序列的,蛋白结合在哪里就捕获哪里。每做一次实验,换一个蛋白,所捕获的序列是不一样的。)因此其主要研究点——研究用不同组蛋白、转录因子等不同蛋白来做不同的实验,找出互作的DNA序列的不同。
3 明差异
测序范围的区别:
全外显子测序测的是所有的能被探针捕获到的外显子区域,在IGV上面能看到reads都是覆盖到外显子及其侧翼区域。所以分析要点就是哪些已知的外显子覆盖度不够好,是探针捕获失败还是样本本身变异呢?外显子的哪些区域跟参考基因组序列不一样呢?
转录组测序测的是能被转录的区域,不需要是已知的外显子,而且reads是可以跨越外显子比对的!所以分析要点是哪些外显子被连接起来了?每个外显子都被覆盖了吗?
ChIP-seq测的是目标蛋白结合的DNA序列,取决于目标蛋白的结合能力,所以它的分析要点就是这些DNA序列在基因组的位置。
测序深度的区别:
全外显子测序的测序深度在大部分区域都是均匀的(反应捕获效果,或者拷贝数变异);
转录组测序一定是不均匀的,以外显子为单位的不均匀(反应表达量差异);
染色质免疫共沉淀测序的测序深度也是不均匀的,以每个碱基为单位的不均匀(反应蛋白结合位点);
4 看图说话
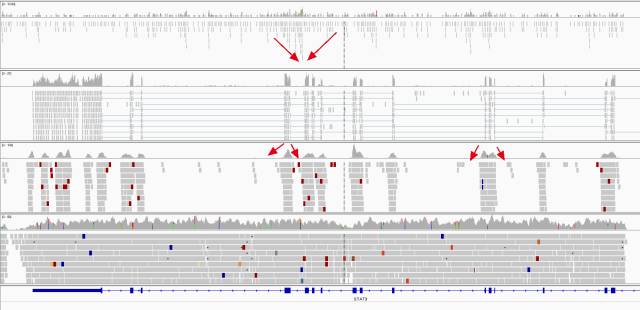
首先从全局的角度来看看这4者的差异
如下图所示:
我只显示了STAT3基因区域的reads覆盖情况。
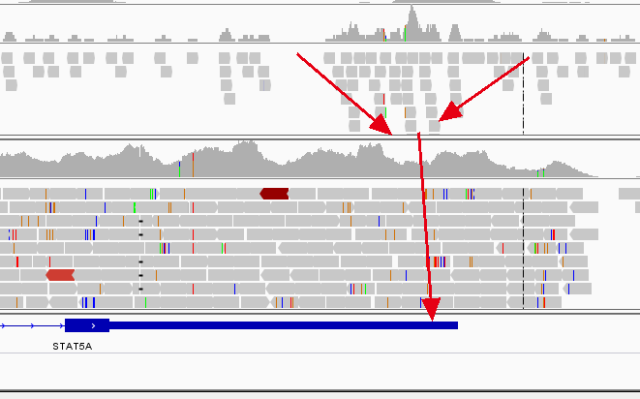
最上面的是ChIP-seq数据,首先,测序深度都不高,而且测序深度极度的不稳定,深浅不一;其次,整个STAT3基因区域似乎都有覆盖到。
第二层是RNA-seq数据,可以看到只有exon对应的区域是有reads覆盖的,非常exon和intron的间隔非常明显,因为是PE测序,还可以看到不同的exon被同一个read跨越了intron连接起来了。至于测序深度,STAT3基因的大部分exon都是等深度的,但是STAT3基因与其它基因的测序深度就不一样了,这个图没办法显示那么多。
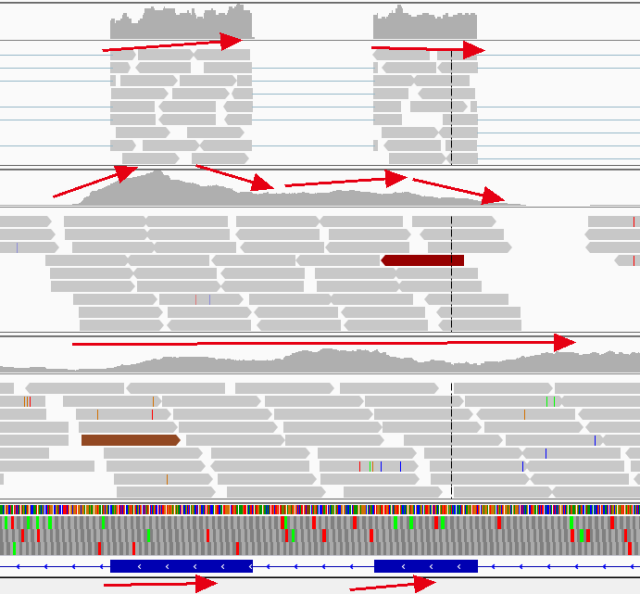
第三层是WES测序,可以看到也是主要覆盖exon区域,但是跟intron区别是没那么整齐的分割线了,因为exon是探针捕获富集的,那么会把exon的侧翼区域也部分捕获,测序深度会由exon往intron递减。(下图是放大比较WES和RNA-seq)
最后是WGS测序,理论是它应该是全基因组覆盖,而且测序深度非常稳定,但是实际上,测序深度还部分由GC含量,扩增随机性影响。
但是它肯定不能像ChIP-seq数据那些很明显形成一些峰,下面可以看比较差异。
写在最后
上面的WGS,WES,RNA-seq组与ChIP-seq之间的异同我曾经在生信菜鸟团QQ群里面讲解过,以QQ群视频的形式,而且还录制了视频,大家可以在公众号后台回复"组学"就可以获取啦!
其实还有关于这几个组学数据分析的异同点, 暂时还没有时间来整理:
(1)使用范围有所不同。外显子组测序只能对已知基因组序列信息的物种进行测序,而转录组测序没有这样的限制。转录组可以对Non-coding RNA等进行测序,而外显子组测序仅限于外显子区域;
(2)转录组可以反映特定时刻、特定组织该物种的基因表达情况,而外显子组测序不具备此能力。但是,因为部分基因低表达或组织特异表达,转录组难以获得物种全部外显子的信息,而外显子组测序不受表达情况影响,可均一地获得外显子区域序列信息;
(3)从转录组获得的遗传信息可能受到转录后加工的影响而导致与基因组不符,而外显子测序无此影响。 染色质免疫共沉淀的测序范围是不确定的、未知的,研究不同蛋白质,其所捕获DNA序列区域是不同的;