今天一个同学给我推荐一篇Nature Protocol 上文章,关于转录组差异表达分析。尚在正式通读之前习惯性浏览一遍图表,说实在这篇文章着实让我觉得有点“另类”。这是一篇活生生的利用Bowtie、tophat和Cufflinks做转录组差异表达分析的protocol。里面详细讲解每一步需要分析什么,用哪些些软件,已经相关命令和参数。
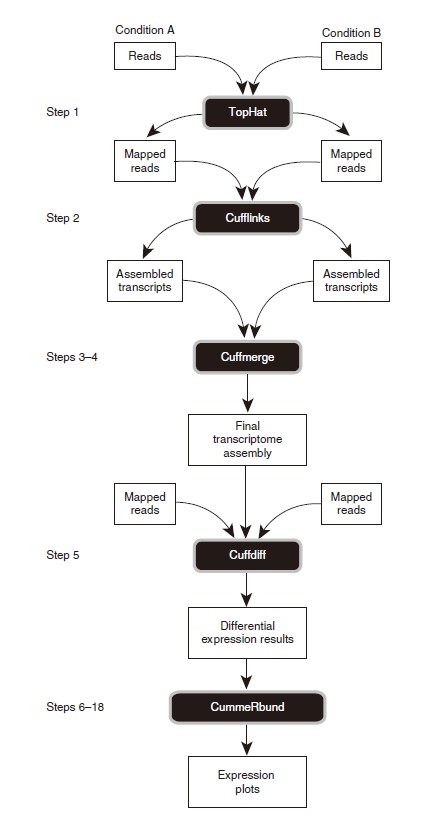
根据文章介绍的workflow,做转录组分析,无论是链特异性转录组数据(Strand-specific RNA-seq)还是非特异性数据,主要内容包括下面几个部分:
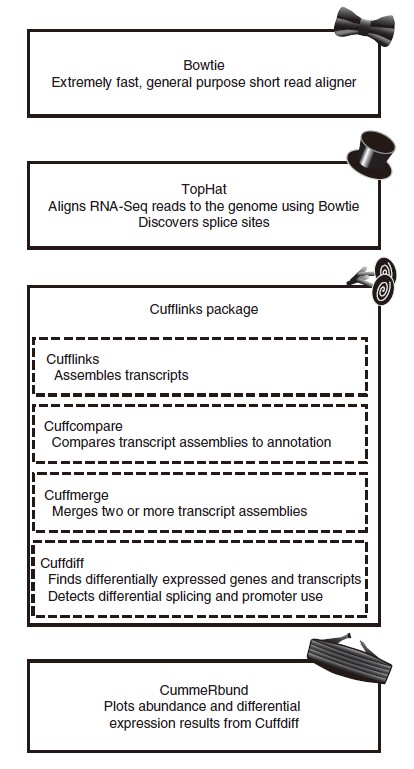
1)reads mapping,这里面推荐两款软件一个是Bowtie,另一个是tophat(此软件相对于Bowtie或者bwa,可以识别转录本的可变剪接)
2)转录组本组装(利用Cufflinks),转录本与已有基因组注释比较(利用Cuffcompare)、合并(利用Cuffmerge),转录组本差异表达分析(利用Cuffdiff)。
下面附上原文中的两张图片供大家快速预览转录组分析大致过程,其中图1是转录组分析中可能会用到的软件以及相关功能,图2:是转录本分析的一般流程。
图1
图2
关于转录组分析的相关软件在分析数据过程中的命令和参数,这里就不附加上来了,请大家直接阅读原文。
没有权限从nature method官方网站上面下载全文的同学,欢迎加入生物信息学QQ:235461986,从群共享下载。也可以考虑从这个链接里面下载:
打开网页后,链接前面有[PDF]这种标示的标示您可以点击直接下载全文。



来自外部的引用