新一代测序技术的出现改变了基因表达谱分析和转录组研究。在其他方法还依赖预知信息进行杂交探针或qPCR引物设计时,RNA-Seq技术可直接对测序文库中任一RNA分子进行无假设的分析。RNA-Seq技术不单能测定转录本数,研究表达水平差异 [1],还能检测序列特异的信息,如转录本的起始和终止位点[2]、SNV [3],和RNA编辑事件[4]。RNA-Seq技术还具有一些其他优势,包括更高的灵敏度、更广的动态范围,以及评估融合转录本和变异表达水平的能力。
Ion PGM™测序仪推动了RNA-Seq技术的普及。结合Ion Total RNA-Seq Kit v2[5]和Ambion ERCC Spike-In Control提供的额外质量控制[6],Ion半导体测序生成数据的灵敏度超过了传统微阵列芯片。Ion半导体测序能够在单次实验中对新转录本、基因融合和等位基因特异表达同时进行检测。Ion PGM™测序仪配有多种产量不同的芯片组合,适合多种转录组大小并能扩展到不同的RNA测序应用。Ion半导体测序提供最快的测序流程,100 base读取的测序运行时间只需约2小时,数据分析直观简便(可使用公共软件)。
用Ion PGM™测序仪与微阵列芯片开展基因表达谱分析的比较
研究显示杂交芯片可产生大约两百万个已定位的读取[7],但也有研究证实,即使读取数目过亿,提高读取数目仍然继续增加其信息量 [8]。
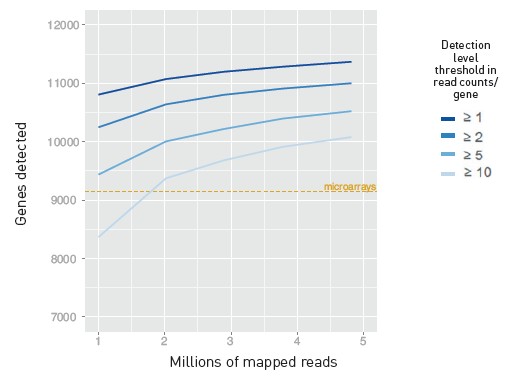
那么,究竟Ion PGM™测序仪生成多少个100 base读取能相当于一个基因表达微阵列芯片呢?在白皮书[9]中我们对Affymetrix GeneChip Human Genome U133 Plus 2.0 Arrays (HG-U133 Plus 2.0)的芯片质量控制(MAQC)的微阵列数据与Ion PGM™系统生成的RNA-Seq数据进行了比较。图1显示了Ion PGM™测序仪数据按照不同检测阈值下所得的基因数和微阵列芯片所能检测到的基因数。当采用最严谨的检测阈值即每个基因≥10读取时,两百万个来自Ion PGM™测序仪的已定位读取所能检测到的基因数超过了微阵列芯片能检测的基因。当读取计数阈值放宽到≥5读段数/基因时,仅仅1百万个Ion RNA-Seq已定位读取所能检测到的基因数就超过了芯片的基因检测数。利用来自Ion PGM™测序仪的2百万个平均读取所能检测到的显著差异表达基因数(sig DEGs)要多于微阵列芯片所能检测到的sig DEGs数。在p ≤0.05和倍数变化f≥2标准下,在HBRR和UHRR样本中Ion PGM™测序仪得到的sig DEGs数为4,630,而相比之下芯片得到的sig DEGs数为4,198。
将Ion PGM™测序仪生成的两百万个已定位读取所产生的基因差异表达结果与qPCR结果相比较,发现qPCR 和RNA-Seq得到的sigDEGs倍数变化的对数值的Pearson相关系数(R)超过0.95。
在Ion PGM™平台进行RNA-Seq实验的另一个优势在于,能够应用ERCC RNA Spike-In Mix对转录本检测灵敏度进行评估。这些ERCC转录本混合物是一些含多聚腺苷酸化且无标记的RNA,该混合物经过美国国家标准技术研究所(NIST)鉴定,是评估RNA测量系统性能的工具,能控制实验可变因素。这些ERCC转录本的GC含量经过平衡,更贴近真核生物内源性mRNA的特征,它们的长度范围在250 到2,000个核苷酸之间。ERCC转录本混合物的含量构成经特殊设计,能代表一个大动态范围的表达水平。ERCC ExFold RNA Spike-In Mix可用于评估基因差异表达的检测灵敏度。这些质控RNA使用户得以直接评估文库质量、检测灵敏度、动态范围以及表达倍数变化。剂量响应散点图显示,ERCC相对浓度的对数值与已定位读取数的对数值呈线性关系(图2)。利用这些外部RNA质控有助于将来自合作实验室所产生的结果进行直接比较。
图1. 在HBRR或UHRR样本间基因检测水平根据已定位读取数的变化。蓝色实线表示随已定位读取的数量增加的基因检测水平的变化。如图所示,RNA-Seq数据的检测水平阈值分别为1、2、5、和10个读取/基因。橙色虚线表示MAQC HG-U133 Plus 2.0 microarray 检测所得的9,140条基因。当采用最严谨的检测阈值≥10个读取/基因时,来自Ion PGM™测序仪的两百万个读取所检测到的基因数超过了microarray。当读取数阈值放宽到≥5个读取/基因时,来自Ion RNA-Seq的1百万个读取所能检测的基因数就超过了microarray的基因检测数。更详细讨论,请参见白皮书[9]。图2. ERCC剂量效应。图中展示了样本的ERCC剂量效应散点图和线性回归数据统计。来自HeLa细胞的Poly(A)富集的RNA加入Ambion ERCC RNA Spike-In Mix,并用于构建RNA-Seq文库,随后用Ion 318™芯片进行测序。原始读取数(y轴)代表总ERCC比对后的读取;x轴是混合液中每个ERCC转录本的相对含量。灰色阴影区域表示90%置信区间。样本量为64时R2为0.9475。
利用Ion PGM™测序仪检测新外显子、剪切突变、融合转录本和SNP
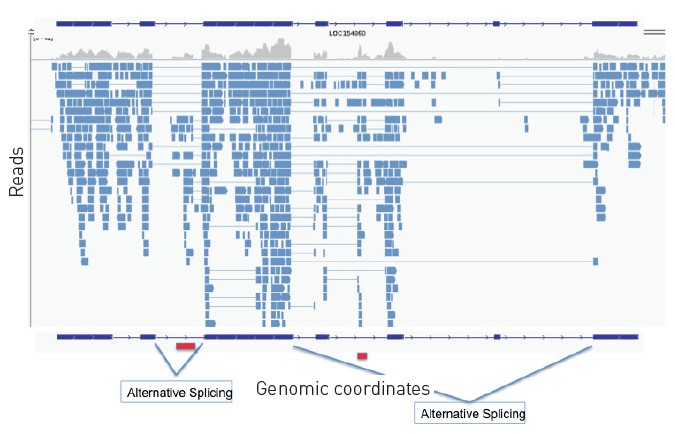
能发现未知的外显子和剪切突变是RNA-Seq的一个关键优势。通过分析RNA-Seq读取在基因组的分布有助于了解基因结构。这与微阵列芯片形成鲜明对比,微阵列芯片只能检测其阵列中已有相应寡核苷酸探针的转录本。图3中将Ion PGM™测序仪生成的RNA-Seq读取定位到基因组,在Ewing尤因肉瘤细胞系样本中揭示了两个新外显子和选择性剪切事件。
图3. 定位读取到基因组上实现了对可变剪切和新外显子的检测。图示为用Ion PGM™测序仪分析Ewing尤因肉瘤细胞系生成的RNA-Seq结果(数据承蒙洛杉矶儿童医院的T. Triche提供)。在这个特例中,测序数据揭示了外显子边界旁的两个新外显子(红色方框)和选择性剪切的发生。该文章正待发表,因此隐去该位点。
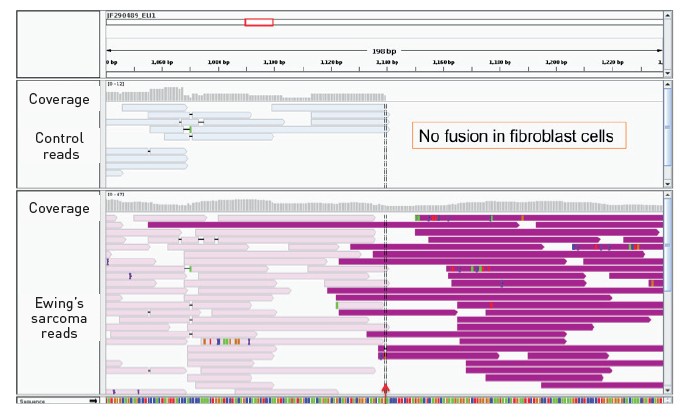
融合转录本被认为是多种癌症的重要致病因素。将Ion PGM™测序仪生成的精确RNA-Seq长读取定位到整个基因组,而非RefSeq中的转录本的参照组装,能在未知外显子边界的情况下,通过定位跨外显子边界的读取来直接检测融合转录本。图4为Ewing尤因肉瘤细胞系样本中22号染色体和11号染色体间的融合转录本,这是传统microarray检测不到的。
图4. 用RNA-Seq检测融合转录本。在Ion PGM™测序仪上用Ion 316™芯片进行100-base测序,将所生成的结果定位到基因组,如图所示在Ewing肉瘤细胞系存在一个跨越22号染色体(左)和11号染色体(右)的融合转录本,但未在成纤维细胞中检测到该融合转录本(数据承蒙洛杉矶儿童医院的T. Triche提供)。
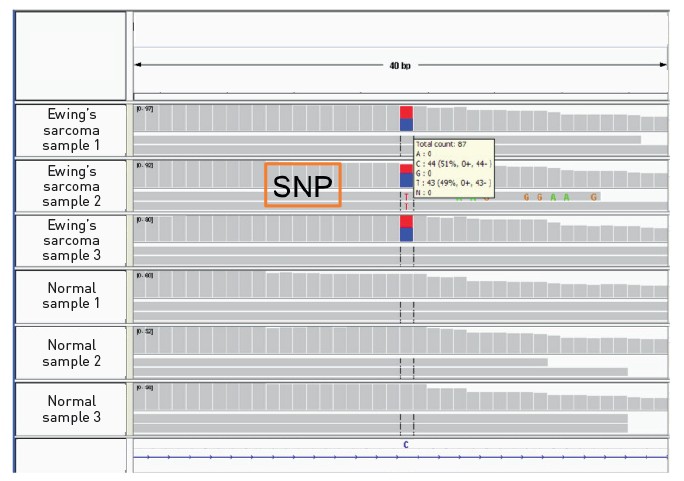
在转录本和等位基因特异性表达中发现的SNP,可实现对RNA或蛋白质结构进行直接分析。图5是Ion PGM™测序仪生成的数据中SNP检出示例。在这种出现杂合型SNP的情况下,研究人员可以直接检测突变转录本的水平并与疾病情况关联起来。
图5. RNA-Seq检测SNP。图为Ion PGM™测序仪分析Ewing尤因肉瘤细胞系得出的RNA-Seq结果(数据承蒙洛杉矶儿童医院的T. Triche提供),显示了一个表达的SNP(前三行数据来自癌症样本,后三行数据来自对照样本)。
应注意的是,同一个体的RNA中SNP检出比DNA中SNP检出要复杂的多。这是因为,每个转录本的表达水平会有变化,从而会影响为获得某个特定转录本的特定覆盖度而所需的总覆盖度。这样,表达量低的转录本比表达量高的转录本需要更多的总读取。其他可能存在因素包括等位基因特异性表达或RNA编辑。在该情况下,转录组数据可能与基因组数据重叠,因此能同时获得基因组序列和RNA序列信息就更有优势。
另外,若基因组中出现一个SNP,而转录组只出现一个等位基因或出现等位基因失衡现象,就意味着发生了等位基因特异性表达。
可扩展的技术
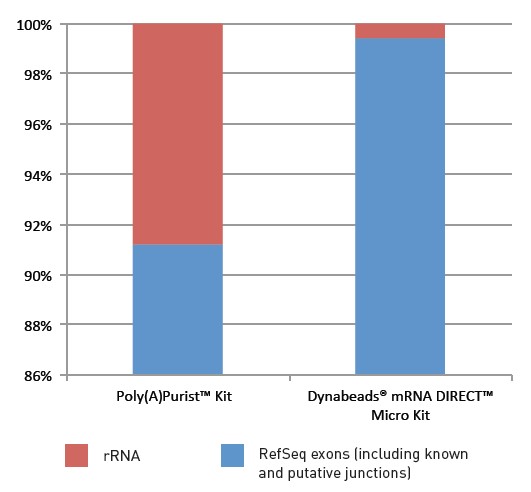
RNA-Seq的一个独特优势在于,通过生成的读取数目,可以轻而易举地调节实验灵敏度。Ion PGM™测序仪的配套芯片组合和新RNA barcodes(Ion Xpress™ RNA-Seq Barcode01-16试剂盒)让研究者能根据实验需要选择读取数目。具有6百万微孔的Ion 316™ 芯片适用于小型转录组研究。而超过一千一百万微孔的Ion 318™芯片能生成超过两百万定位RefSeq exons的读取[10],如上所述其灵敏度超越了HG-U133 Plus 2.0 array。随着Ion半导体测序技术的持续快速发展,转录组分析能力和方法也将随之改进。例如,经过优化的Dynabeads mRNA DIRECT™ Micro试剂盒在从总RNA抽提mRNA时,能进一步减少产物中的rRNA含量,增加定位于RefSeq的读取数。如图6所示,使用这个仅耗时30分钟的从总RNA中纯化poly(A) RNA的方法,定位于rRNA的读取数显著降低,而相应增加了定位于RefSeq exons的读取数。
图6. mRNA分离方法对RefSeq读取分布的影响。分别使用Poly(A) Purist™ 试剂盒和Dynabeads mRNA DIRECT™ Micro 试剂盒从HeLa总RNA中抽提mRNA。用得到的mRNA制备文库,并使用Ion Total RNA-Seq v2试剂盒和Ion 318™芯片进行测序。如图显示,定位于rRNA的读取数显著降低,而定位RefSeq外显子的读取数相应增加。
最快的测序流程和简洁直观的RNA-seq分析
Ion PGM™测序仪为RNA-seq实验提供了最快的测序流程,100-base测序仅需约2小时。数据分析过程分为两步。首先使用配套的Torrent软件将数据定位到参考基因组并将结果输出为BAM文件格式(包含所有已定位信息)。然后利用新一代测序基因表达分析软件包对该BAM文件进行分析。这样的软件包很多,包括学术部门的免费软件(如Connecticut大学的IsoEM),还有Strand Genomics 的Avadis软件、Partek Genomics Suite软件等商业化产品,传统microarray用户可能已在使用这些软件。
结论
Ion PGM™测序仪结合Ion芯片和Ion Total RNA-Seq Kit v2是一种快速、简单且可扩展的RNA测序解决方案。Ambion RNA制备试剂盒完全支持Ion工作流程,可为各种读长(不论是100还是200 bases)提供从样品到结果的最快解决方案。Ion PGM™测序仪得到的RNA测序结果与传统基因表达芯片相比更具优势,不仅灵敏度更高,还能生成无假设(hypothesis-neutral)的测序数据,帮助研究者发现融合转录本、新外显子和选择性剪切,以及检测SNP和等位基因特异性表达模式。
参考文献
1. Clonnan N. et al. 2008, Nat Methods, Stem cell transcriptome profiling via massive-scale mRNA sequencing, doi:10.1038/NMETH.1223
2. Hashimoto S. et al. 2009, PLoS One, High-Resolution Analysis of the 59-End Transcriptome Using a Next Generation DNA Sequencer, doi:10.1371/journal.pone.0004108
3. Tuch B. et al. 2010, PLoS One, Tumor Transcriptome Sequencing Reveals Allelic Expression Imbalances Associated with Copy Number Alterations. doi:10.1371/journal.pone.0009317
4. Picardi E. et al. 2010, Nucleic Acids Res, Large-scale detection and analysis of RNA editing in grape mtDNA by RNA deep-sequencing. doi:10.1093/nar/gkq202
5. http://products.invitrogen.com/ivgn/product/4475936
6. http://products.invitrogen.com/ivgn/product/4456740
7. Ramsköld D. et al. 2011, PLoS Comput Bio, An Abundance of Ubiquitously Expressed Genes Revealed by Tissue Transcriptome Sequence Data. doi:10.1371/journal.pcbi.1000598
8. Toung J.M. et al. 2011, Genome Res, RNA-sequence analysis of human B-cells, doi:10.1101/gr.116335.110
9. “Sensitivity of RNA-Seq using Ion semiconductor sequencing” ioncommunity.iontorrent.com
10. “C18-199” data set, ioncommunity.iontorrent.com
来自:生物通http://www.ebiotrade.com/newsf/2012-6/2012613102715265.htm



1F
希望能出安卓和苹果等智能机的终端。这样可以移动设备登陆。随时学习