导读
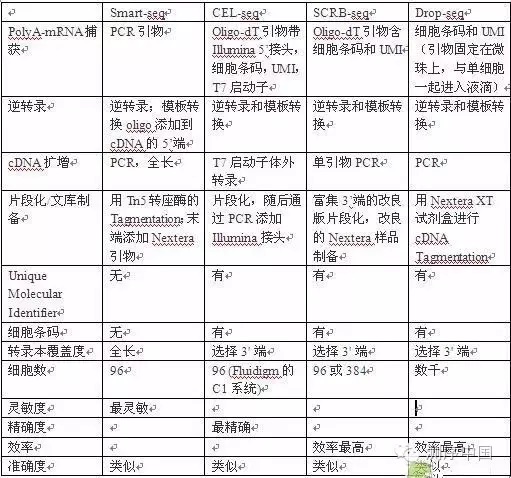
在单细胞研究的大潮中,新的测序方法层出不穷。不过,很少有人对这些方法进行系统的比对。慕尼黑大学生物学家Wolfgang Enard最近领导团队,在小鼠胚胎干细胞的基因表达研究中比较了一些常用的单细胞测序方法,包括Smart-seq、CEL-seq、SCRB-seq和Drop-seq。
Smart-seq
SMART(Switching mechanism at 5’ end of the RNA transcript)是一个具有里程碑意义的重要技术。实际上,能够从单细胞生成全长cDNA的测序方案并不多,Smart-seq就是其中之一。对于等位基因特异性表达或者剪接变体研究来说,覆盖整个转录组是一件非常重要的事情。
Fluidigm C1单细胞制备系统能够自动完成Smart-seq步骤。你只需要将制备好的细胞悬液加进去,仪器就会分离并裂解细胞,把mRNA逆转录为cDNA,再对cDNA进行扩增。扩增后的cDNA可以拿来测序,也可以进行qPCR检测。
在Enard的比较研究中,Fluidigm C1系统的Smart-seq最为灵敏,成本也最高。这一系统使用的微流体芯片不能重复使用,不过这种芯片可以放到显微镜下观察,证实健康单细胞的存在。
CEL-seq
CEL-seq(Cell expression by linear amplification and sequencing)是一种采用线性扩增的常用测序方法。线性扩增的主要优势是错误率比较低,不过线性扩增和PCR都存在序列依赖性偏好。
CEL-seq技术发表于2012年,主要是分离单细胞,逆转录带有poly-A 尾巴的mRNA片段,给它们贴上代表其细胞来源的条码。2014年Science杂志发布的MARS-seq与CEL-seq很类似。
CEL-seq和其他线性扩增方法生成文库花费的时间要稍微长一点。不过,CEL-seq在早期阶段就给样本贴上条码并进行混合,大大减少了手动操作的时间。PCR是在最后阶段使用的,主要是为了连接正确的测序接头,CEL-seq开发者纽约大学的Itai Yanai介绍到。
CEL-seq所需试剂都是现成的,大约两天时间生成测序文库和测序数据,Yanai指出。需要注意的是,CEL-seq与大多数方案一样,测序转录本的3’端。Enard等人并没有亲自尝试着一技术,只是在比较研究中用到了CEL-seq数据。从这项研究来看,CEL-seq的重现性最好。
GitHub网站提供有CEL-seq可用的生物信息学工具。Yanai研究团队正在开发新版本CEL-seq2,新版本的灵敏度将比旧版本高三倍。
SCRB-seq
Broad研究所开发的SCRB-seq(single-cell RNA barcoding and sequencing)技术采用的是PCR扩增。该技术需要结合流式细胞仪(FACS)或者其他细胞分选方法,把单细胞分配到微孔里去。
SCRB-seq与Smart-seq比较相似,只不过SCRB-seq会整合特异性的细胞条码,以分辨扩增分子的来源,更准确的定量转录本。此外,SCRB-seq并不生成全长cDNA,而是像CEL-seq一样富集RNA 3’端。
2014年,开发者们将SCRB-seq技术提前发布在BioRXiv上。随后他们对这一技术进行了更新,并将其更名为“high-throughput eukaryote 3’ digital gene expression”。Broad研究所仍提供有SCRB-seq技术服务,SCRB-seq也被整合到了Wafer GenBio systems公司的scRNA-seq平台上。据说Fluidigm公司的C1系统很快也将支持SCRB-seq方案。SCRB-seq目前是Enard最中意的测序方法,它不仅适用于单细胞RNA测序,还能支持大量细胞(bulk)的RNA测序。可惜的是SCRB-seq并不好DIY,研究者自己很难将测序流程与FACS对接起来。
DROP-seq和inDROP
哈佛医学院的研究人员开发了以微滴为基础的两种独立技术Drop-seq和inDrop。他们利用微流体装置将带有条码的微珠和细胞一起装入微小的液滴,建立了快速、廉价、高通量的单细胞RNA-seq方法。这两种技术将细胞隔离在微小的液滴中,装上用于扩增的条码引物,由此检测数以千计的细胞。研究者们认为,Drop-seq和inDrop能够帮助生物学家进一步发现和分类人体细胞,绘制大脑等复杂组织的细胞多样性图谱,更好地了解干细胞分化,获得更多疾病的遗传学信息。
Enard及其同事发现,Drop-seq在单个细胞中检测的基因数还不到Smart-seq/C1、CEL-seq和SCRB-seq的一半。不过,在统计学水平上研究差异性表达的时候,高通量Drop-seq和SCRB-seq是最划算的。哈佛医学院Steve McCarroll实验室去年下半年根据用户反馈,在自己网站上公布了3.1版的Drop-seq(mccarrolllab.com/dropseq)。
参与开发SCRB-seq的Stefan Semrau正在搭建自己的Drop-seq平台。Semrau从Whitehead生物医学研究所搬到Leiden物理研究所的时候,发现自己用FACS已经不那么方便了,于是他为新实验室选择了Drop-seq。建立微流体芯片和液滴系统是整个过程中最艰难的部分,不过Semrau实验室的一名微流体博士后仅用三周就搞定了这些问题。Drop-seq文库制备是任何分子生物学研究者都熟悉的标准操作。据Semrau估计,搭建和优化Drop-seq大概只需要六个月时间。
四种单细胞RNA-seq方法比较
本文来自:http://mp.weixin.qq.com/s?__biz=MzIzODE4OTAyNA==&mid=2651614750&idx=1&sn=851aeb6589a6c93c95190edf1da9b543&scene=2&srcid=0615MXX9COMLO2yzLDH6gazF&from=timeline&isappinstalled=0