Maftools系列文章:
上篇文章《肿瘤变异数据分析和可视化工具maftools:突变的数据分析》介绍了使用maftools分析MAF格式的突变数据,但maftools本身功能不限于此,它也可以针对拷贝数变异进行一些分析。
CNV数据下载和处理
还是继续之前TCGA-LUAD的例子。Maftools接受的CNV数据需要是GISTIC输出的结果,包括4个文件——all_lesions.conf_XX.txt、 amp_genes.conf_XX.txt、del_genes.conf_XX.txt以及scores.gistic(其中XX代表置信水平)。但是从TCGA官网能下载到的GISTIC的输出结果只有LUAD.focal_score_by_genes.txt,因此需要下载更上游的DNAcopy的输出结果,然后自己再根据TCGA的流程和参数跑一遍GISTIC。
但这样有点麻烦,更简单的办法就是下载其他数据库已经处理好的。这里我直接从Firehose下载GISTIC2的输出结果,把其中all_lesions.conf_99.txt、amp_genes.conf_99.txt、del_genes.conf_99.txt、scores.gistic四个文件挑出来。需要注意的是,Firehose的分析流程用的是hg19,而TCGA官方目前已经用hg38了,所以参考基因组的坐标是不一致的,因为后面画oncoplot需要用到突变数据,所以索性在Firehose也下载对应的临床数据和MAF文件。
不过怕麻烦就得付出代价,Firehose的数据更新速度实在太慢,直到今天9012年了还在用2016_01_28版的TCGA数据,导致TCGA-LUAD这个项目只有230个case的MAF文件(占总585个case的39%),而官网和UCSC Xena的数据虽然比较新,但是都下不了maftools需要的GISTIC2输出的4个文件,所以最好还是自己用DNAcopy的结果跑一下GISTIC2(如果有更好的办法可以在评论区留言!)。这里只是做演示,所以我还是偷下懒算了。
通过简单的数据整理后文件如下(包括各case的MAF文件合并以及《肿瘤变异数据分析和可视化工具maftools:突变数据下载和可视化》一文中提到的两个需要处理的地方):
$ ls -lhrt total 237M -rw-r--r-- 1 xiaofei xiaofei 459K Jun 7 19:26 all_lesions.conf_99.txt -rw-r--r-- 1 xiaofei xiaofei 14K Jun 7 19:26 amp_genes.conf_99.txt -rw-r--r-- 1 xiaofei xiaofei 63K Jun 7 19:26 del_genes.conf_99.txt -rw-r--r-- 1 xiaofei xiaofei 2.5M Jun 7 19:26 scores.gistic -rw-r--r-- 1 xiaofei xiaofei 223M Jun 7 19:34 TCGA.LUAD.maf -rw-r--r-- 1 xiaofei xiaofei 11M Jun 7 19:40 LUAD-TP.samplefeatures.txt
使用maftools读取GISTIC输出的CNV数据并统计
使用函数readGistic读入GISTIC2输出的4个文件:
> library(maftools) > luad.gistic <- readGistic(gisticAllLesionsFile="all_lesions.conf_99.txt", gisticAmpGenesFile="amp_genes.conf_99.txt", gisticDelGenesFile="del_genes.conf_99.txt", gisticScoresFile="scores.gistic", isTCGA=TRUE) ## -Processing Gistic files.. ## --Processing amp_genes.conf_99.txt ## --Processing del_genes.conf_99.txt ## --Processing scores.gistic ## --Summarizing by samples
直接键入GISTIC对象luad.gistic就能得到一些简单的统计:
> luad.gistic ## An object of class GISTIC ## ID summary ## 1: Samples 516 ## 2: nGenes 6057 ## 3: cytoBands 75 ## 4: Amp 177966 ## 5: Del 896197 ## 6: total 1074163
和之前介绍的MAF文件的统计方法类似,GISTIC对象可以使用getSampleSummary、getGeneSummary、getCytobandSummary进行统计,并用write.GisticSummary保存统计结果:
getSampleSummary(luad.gistic) getGeneSummary(luad.gistic) getCytobandSummary(luad.gistic) write.GisticSummary(gistic=luad.gistic, basename="luad_gistic2")
CNV数据的可视化
GISTIC的输出结果可以用染色体图、气泡图(点图)、oncoplot进行可视化。
1. 染色体图
使用gisticChromPlot可以绘制染色体各位点G-score:
gisticChromPlot(gistic=luad.gistic, markBands="all")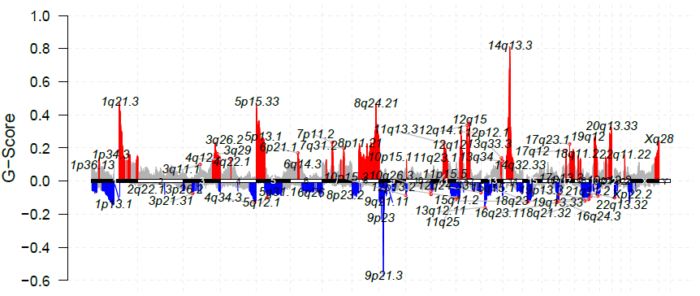
2. 气泡图
gisticBubblePlot函数可以将展示显著变化的cytobands对应的样本数(X轴)和包含的基因数(Y轴),圆点的大小为-log10(qvalue):
gisticBubblePlot(gistic=luad.gistic)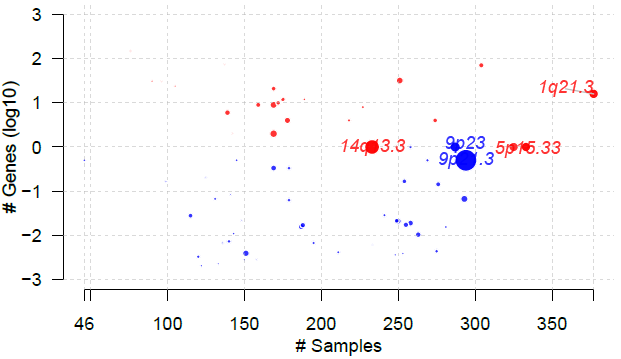
3. oncoplot
可以使用gisticOncoPlot函数以oncoplot的形式展示CNV。是否根据临床数据进行分类是可选的,如果要标注临床信息,需要先用read.maf建立MAF对象,这里选择性别:
luad <- read.maf(maf="TCGA.LUAD.maf", clinicalData="LUAD-TP.samplefeatures.txt") gisticOncoPlot(gistic=luad.gistic, clinicalData=getClinicalData(x=luad), clinicalFeatures="CLI_gender", sortByAnnotation=TRUE, top=10)
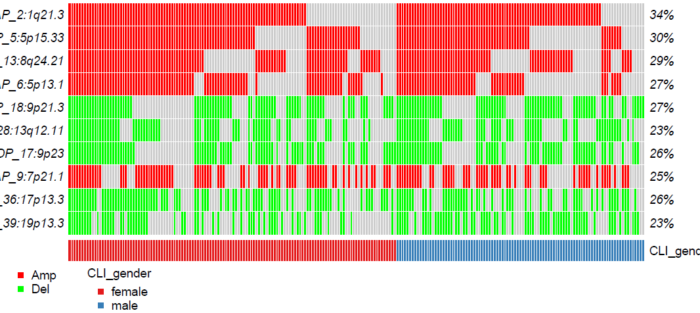
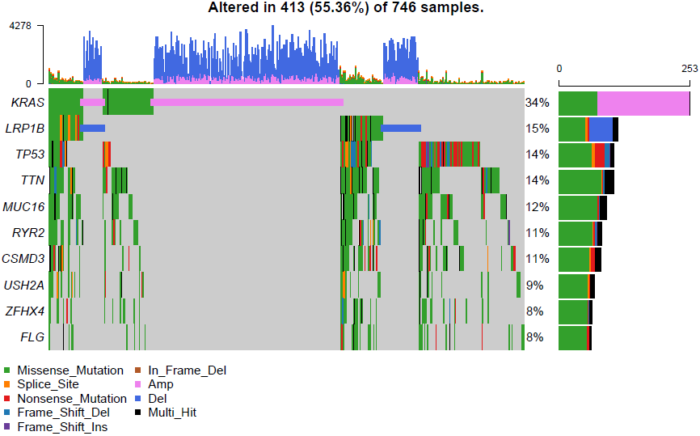
也可以用oncoplot同时展示CNV和突变:
luad.plus.gistic <- read.maf(maf="TCGA.LUAD.maf", gisticAllLesionsFile="all_lesions.conf_99.txt", gisticAmpGenesFile="amp_genes.conf_99.txt", gisticDelGenesFile="del_genes.conf_99.txt", gisticScoresFile="scores.gistic") # 因为样本数太多,使用borderCol=NULL不显示样本边界。可惜的是目前gisticOncoPlot还没有加上这个参数 oncoplot(maf=luad.plus.gistic, borderCol=NULL, top=10)
4. 可视化segment文件
Maftools除了可以可视化GISTIC的输出结果以外,还可以可视化DNAcopy输出的segment文件。这里以《肿瘤变异数据分析和可视化工具maftools:突变的数据分析》一文中已经处理过的文件TCGA-38-4625.seg为例:
plotCBSsegments("TCGA-38-4625.seg")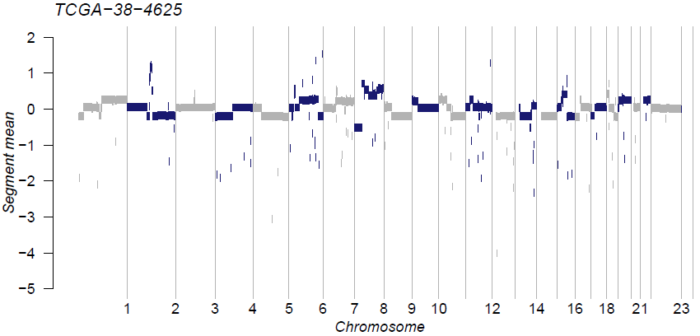
要注意的是,目前这个函数感觉还不太成熟,连help都没有写,而且对于女性,segment文件中没有Y染色体,会引发报错,因此需要在文件最后补上一行Y染色体才能正常绘图:
TCGA-38-4625Y 0 0 0 0


