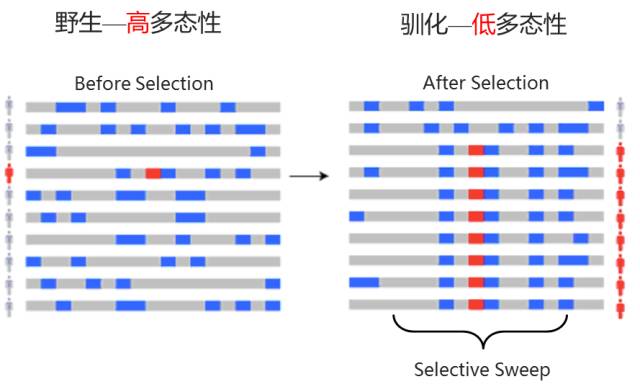
游戏“天天爱消除”曾经风靡一时,想必大家一定都玩过。其实在重测序分析中也有一个分析“爱消除”,那就是选择消除分析。群体进化选择消除分析:简单的说就是基因组某区域由于受到了选择而消除多态性。选择消除分析是正向选择在物种基因组上留下的印迹。与野生祖先相比,栽培或驯化的物种发生选择消除的区域遗传多样性显著降低,这是驯化区域的典型特征。
图1 物种驯化过程中多态性的变化趋势
选择消除分析可以做什么?
- 挖掘人工驯化过程中受选择的基因,如家猪与肉质相关的基因,大豆中与油含量相关的基因。
- 挖掘物种适应性进化中受选择的基因,如藏猪的高原适应性,毛果杨高低纬度气候适应性。
选择消除分析常用的方法
1、基于群体分化(Fst)
群体的固定系数F反映了群体等位基因杂合性水平。固定系数F是F统计量(Fst)的一个特例。Fst分析表示群体的分化程度,值越大,群体分化程度越高,受选择程度越高。
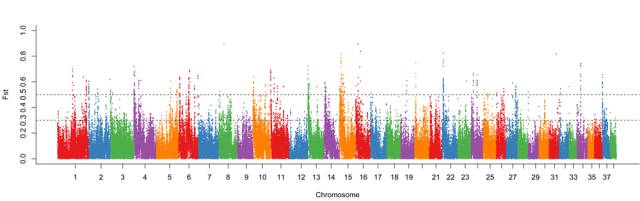
图2 Fst 分布图
Fst分布图,横坐标代表不同的染色体名称,纵坐标代表相应染色体窗口内Fst值,另外的两条虚线代表两种选择阈值(top 5%或1%)。
2、基于群体多样性分析
π用来分析碱基多态性,多态性越低,受选择程度越高。
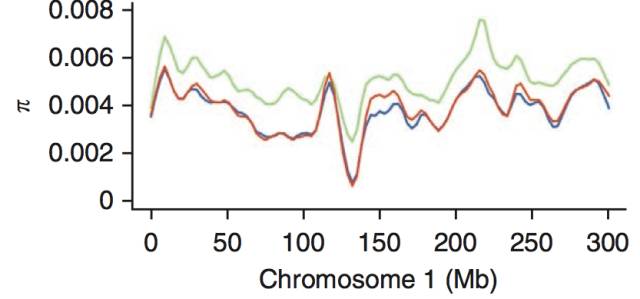
图3 碱基多样性分析[1]
π结果分析图中,横轴表示基因组的位置,纵坐标表示π值,图中的曲线表示3个群体的多态性水平。群体π值反映了群体基因组碱基多样性。
3、基于群体中性进化的分析
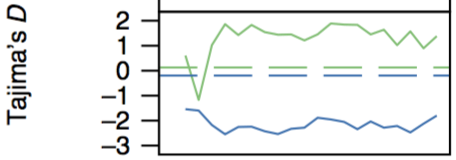
在标准中性进化模型下,Tajima’s D 的理论值为零。如果Tajima’s D 值为正,表明存在大量的中等频率的等位基因,这可能是由于群体瓶颈效应,群体结构,或者平衡选择引起的。如果 Tajima’s D 值为负,表明存在大量的低频等位基因位点。Tajima’s D分析表示中性进化,越偏离0,受选择程度越高。
图4 Tajima’s D分析[2]
选择消除分析多种算法的结合
1、Fst和θπ 的结合
Fst 和θπ已被证实是一种很有效力地检测选择消除区域的方法,特别是在挖掘与生存环境密切相关的功能区时,往往可以得到较强的选择信号,图5为Fst与θπ选择消除分析结果展示,二者共同筛选较强的选择信号,便于目标基因的筛选。
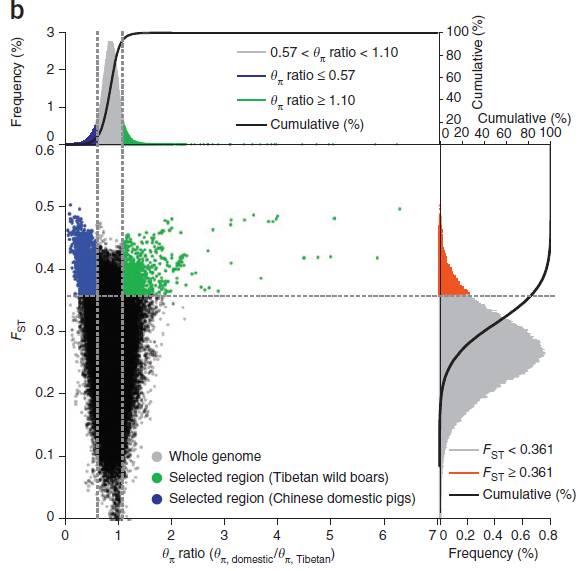
图5 Fst和θπ的选择消除分析[3]
如何看懂这张图:
横坐标为θπ 的比值(驯化/野生),纵坐标为Fst值,分别对应上面的频率分布图和右侧的频率分布图,中部的点图则代表不同窗口内的相应的Fst和θπ 比值。其中最上方蓝色和绿色区域为θπ选择出来的top 5%区域,红色区域为Fst所选择top 5%区域,中间蓝色和绿色区域为Fst和θπ的交集,即为候选的位点。
2、Tajima’s D和θπ的结合
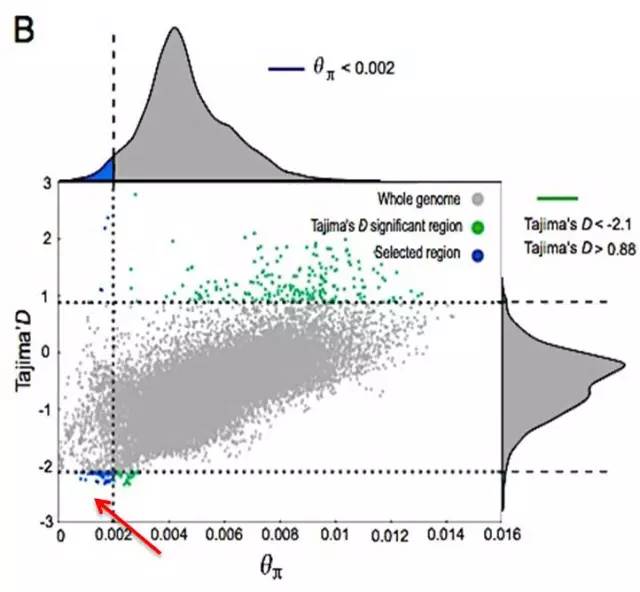
图6 Tajima’s D和θπ的选择消除分析
如何看懂这张图:
横坐标为θπ 值,纵坐标为Tajima’s D值,分别对应上面的频率分布图和右侧的频率分布图,中部的点图则代表不同窗口内的相应的θπ和Tajima’s D值。其中蓝色区域为θπ选择出来的top 5%区域,绿色区域为Tajima’s D选择出的top 5% 区域,红色箭头所指的区域为θπ和Tajima’s D的交集,即为候选的位点。
3、Fst和Hp的结合
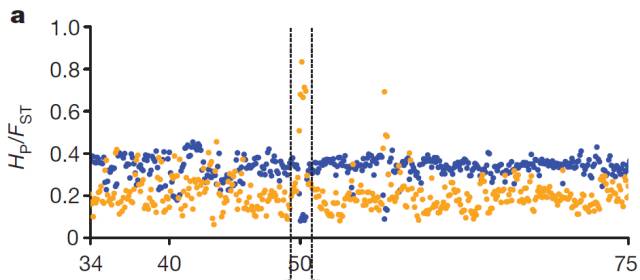
图7 Fst和Hp的选择消除分析[4]
如何看懂这张图:
横坐标表示染色体上的某个区间,纵坐标表示Hp、Fst,图中的蓝点表示杂合率(Hp),图中的橙黄色点表示固定系数(Fst)。通常选择杂合率Hp较小,群体分化系数Fst较大的点,图中两虚线间的点即为候选位点。
参考文献:
[1] Hufford M B, Xu X, van Heerwaarden J,et al. Comparative population genomics of maize domestication andimprovement. Nature Genetics, 2012, 3(6),44(7): 808-11.
[2] Qiu Q, Wang L, Wang K,et al. Yak whole-genome resequencing reveals domestication signatures and prehistoricpopulation expansions. Nature Communications, 2015,22(12), 6:10283.
[3] Li M,Tian S,Jin L,et al. Genomic analyses identify distinct patterns of selection in domesticatedpigs and Tibetan wild boars. Nature Genetics, 2013,45(12): 1431-8.
[4] Axelsson E, Ratnakumar A, Arendt M L,et al. The genomic signature of dog domestication reveals adaptation to astarch-rich diet. Nature, 2013 Mar 21(3),495(7441): 360-4.