规范的命名是基因变异解读中不可或缺的一部分。ACMG变异分类标准建议对变异进行统一的标准化命名以确保定义明确并实现基因组信息的有效共享和下游使用。1998年由人类基因组变异协会(HGVS)、人类变异项目组(HVP)和人类基因组组织(HUGO)联合成立序列变异描述工作组(SVD-WG),旨在建立一个稳定、有意义、且明确的命名系统,并不断更新和修正。
HGVS的宗旨是明确定义,避免出现易混淆的概念或定义。在实际应用环境中有些学科将不能引起疾病的变异定义为多态性,有些则将人群发生频率大于1%的变异统称为多态性;同时,突变有时仅单纯表示序列改变,也还被特指为能够引起疾病的变异,并逐渐被认为具有负面含义。为此2016年更新版本中建议取消这两个专业术语,使用“变异”作为替代词。
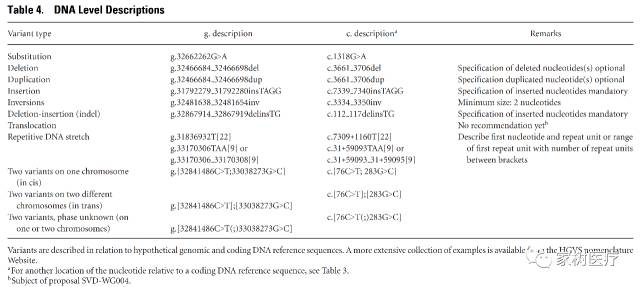
为了提高准确度、促进序列变异的计算机分析和描述,必须严格定义变异的基本类型。更新版将变异类型分为7类(下表)。
- 置换(>):一个核苷酸被另一个核苷酸替代,使用“>”来表示;例如g.1318G>T;
- 缺失(del):一个或多个核苷酸被移除,使用“del”进行描述;例如g.3661_3706del;
- 倒置(inv): 与原始序列反向互补的新的核苷酸序列(大于1个核苷酸)替换原始序列,例如由CTCGA变为TCGAG,使用”inv“表示;
- 重复(dup):一个或多个核苷酸拷贝直接插入原始序列的下游,使用“dup”表示;
- 插入(ins):序列中插入一个或多个核苷酸,并且插入序列并非上游序列拷贝;
- 缺失-插入(delins/indel):一个或多个核苷酸被其他核苷酸替代,但并不是发生替代、倒置和转置;
- 转换(con):一种特殊类型的缺失-插入,其中替代原始序列的核苷酸序列是来自基因组中另一个位点的序列拷贝;
更新后定义更加明确,有效避免了概念混淆。例如A>T表示置换而非倒置,倒置应包含多个核苷酸序列;一个核苷酸被一个以上的核苷酸序列替代时,不应描述为置换,应属于缺失-插入变异;重复是指上游序列的串联拷贝,当插入的位置不在原始序列的下游时,应将变异类型描述为插入。插入的序列通常是短的、新生成的,并非基因组上现有序列的拷贝。较大序列的重复插入,指的是该序列在基因组其他位置有拷贝,描述时应定义原始序列的参考序列和核苷酸范围。
在使用或提交变异信息时,应关注参考序列数据库(Genbank、EMBL、DDJB、SWISS_PROT)的检索号(例如M18533),变异描述需包含DNA、RNA和蛋白质水平,并明确标注变异是通过实验确定还是仅为理论推断。HGVS强烈建议使用确定的LRG数据库作为参考序列,若当前没有该序列应及时提出,并推荐使用RefSeq序列或转录本。由于基因组序列信息更为完善,包含多个启动子、可变剪接位点、不同的poly-A 信号,不同的翻译起始位点及长度变化等,通常将基因组作为首选参考序列。但大多数情况下特别是在疾病诊断时,以cDNA为参考序列的报告更受欢迎,鉴于其与RNA、蛋白质的相关性,可以直观便捷的确定变异的位置及受影响的氨基酸数目。为避免序列变异描述中出现混淆,通常使用一个字母表示参考序列的类型(下表)。
g:基因组序列;
c:cDNA序列
m:线粒体序列
n:非编码DNA序列
r:RNA序列
p:蛋白质序列
DNA水平
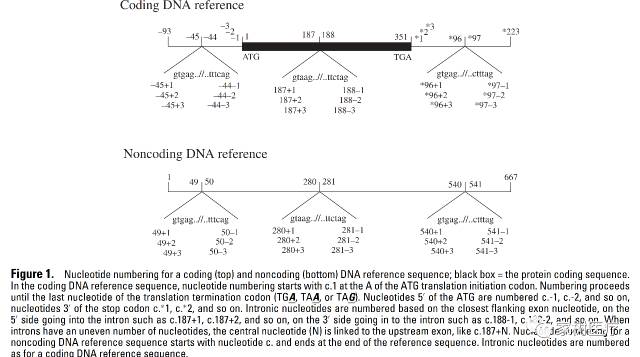
DNA水平的变异描述根据参考序列的不同可以分为三类:基因组、cDNA和非编码DNA。使用大写字母表示核苷酸。对于编码序列来说,将参考序列中翻译起始密码子ATG中的A编号为c.1,编码进行到翻译终止密码子的最后一个核苷酸。非编码区中ATG上游依次编号为“c.-1、c.-2……”,终止密码子下游依次编号为“c.*1、c.*2……”至参考序列结尾处结束编号。内含子是根据相邻外显子核苷酸进行编码的,如图编码区187-188核苷酸间的内含子,其5’端编号为“c.187+1、c.187+2……”,3’端编号为“c.188-1、c.188-2……”,当内含子包含不均匀的核苷酸数目时,使用“N”表示中央核苷酸,并连接上游序列,例如c.187+N,内含子序列的编号至参考序列的编码区(下图)。
此外,HGVS还明确声明撤回之前推荐的使用c.IVS #和c.EX#替代内含子编码的声明,这样的描述方式容易造成混淆并且不利于软件系统的开发和识别,建议所有编号都具有唯一性且只包含数字(下表)。
变异描述示例:
- c.76A>C76A>C:76位的核苷酸A变异为C;
- c.82_83delTG:位于82和83位点上的核苷酸TG缺失,ACTTTGTGCC变异为ACTTTGCC(A是第76位);
- c.83_84dupTG: ACTTTGTGCC(A为第76位)的83-84位之间插入短的串联重复序列TG,变为ACTTTGTGTGCC;
- g.333_590con1844_2011:基因组中编号为333-590的核苷酸序列替代1844-2011原有序列,插入其中;
- g.112_117delinsTG:在基因组序列编号为112-117之间的6个核苷酸被TG替换;
- 多个变异使用”[]”标注变异,并用“;”链接
*同一等位基因发生多个变异
- c.[76A >C;83G>C]:同一染色体上76位和83位发生两个变异(順式);
*不同等位基因发生多个变异
- c.[76A >C];[83G>C]:两个变异发生在不同染色体上(反式);
*不确定多个变异发生的位置
- c.[76A >C](;)[83G>C]:两个变异可能发生在同一染色体,也可能发生在不同染色体上,用(;)来链接;
- 重复序列的变异描述
*定义重复序列的核苷酸范围及重复单位的数量,并用“[]”表示
- g.123_124[4]:基因组序列中第123-124间的核苷酸重复出现4次;
*对于短的/简单的重复,可以展示重复序列
- g.123TG[4]:基因组序列中从123位开始TG核苷酸重复出现4次;
*当重复序列长度不确定时,使用括号进行指定
- g.-128GGC[(600-800)]:基因组编码区上游128位核苷酸处重复插入GGC,重复次数在600-800之间;
RNA水平
RNA使用“r”标注参考序列,从编码RNA或非编码RNA参考序列的第一个核苷酸开始编号,使用小写字母表示核苷酸(a、c、g、u)。变异描述方式与DNA相同。当DNA变异影响到多个转录本时,使用“[]”标注并使用“,”连接。例如r.[76a>c,70_77del]:DNA序列上76位核苷酸由A置换为C,导致产生两个RNA产物,其中1条携带76a>c变异,另一条RNA链发生70-76位点间核苷酸的缺失(下表)。
蛋白水平
蛋白质“p”标注参考序列,强调蛋白水平的结果描述应标注是实验验证还是功能预测。最新版本遵循IUPAC-IUB命名方式,强烈推荐使用三个字母缩写表示氨基酸,并限制单字母描述方式在长序列变异中的应用。此外,在IUPAC-IUB中定义“X”表示任意氨基酸,为此HGVS建议使用“Ter(三字母缩写氨基酸)”或“*(单字母缩写氨基酸)”表示终止密码子,例如p.Trp123Ter或p.W123*。在移码突变的描述中增加了影响起始密码子和终止密码子变异的描述方式,对于预测的移码突变可以用两种方式展示,例如p.(Arg97fs)或p.(Arg97Profs*23)。“fsTer#”或“fs*#”使用“#”表示新的读码框以终止密码子结束,新的读码框编号从改变的氨基酸开始到终止密码子结束。同时新增变异对起始和终止密码子在N端或C端延伸的影响,使用“ext”表示,例如p.Met1ValextMet-12表示DNA序列上c.1A>G的变异导致最终蛋白产物在N端延伸了12个氨基酸,Met变异为Val。同理p.Ter110GlnextTer17表示DNA c.331T>C变异导致蛋白产物在C端多生成了17氨基酸,同时终止密码子变异为Gln。对于存在基因变异但未预测到蛋白变化的,可用“=”描述,如p.(Arg152=)(下表)。
HGVS变异命名得到了广泛的认可,但是随着不断发展和演变,对大多数遗传学专家尤其是非专业人员来说完全掌握变得非常困难,这就需要开发辅助性工具,HGVS在变异更新的过程中也将计算机分析的识别和兼容纳入考虑范围。目前已发表文章中介绍一款基于Python语言的开源文件包(https://github.com/counsyl/hgvs),协助从业人员本地化识别患者的变异信息,基于解析器的功能利用解析表达语法和变异映射器适应参考序列和转录本中的插入缺失变异,并持续不断的扩展、更新数据资源。
参考文献
[1] den Dunnen et al. HGVS Recommendations for the Description of Sequence Variants: 2016 Update.Hum Mutat.2016 Jun;37(6):564-9.
[2] Hart RK et al. A Python package for parsing, validating, mapping and formatting sequence variants using HGVS nomenclature.Bioinformatics.2015 Jan;31(2):268-70.
[3] den Dunnen et al. Nomenclature for the description of human sequence variations.Hum Genet.2001 Jul;109(1):121-4.