本篇主要内容
本篇主要介绍两种方法,搜索并批量下载ENCODE数据。
方法一:在页面中搜索并获取下载地址
- 进入ENCODE portal主页:https://www.encodeproject.org/。
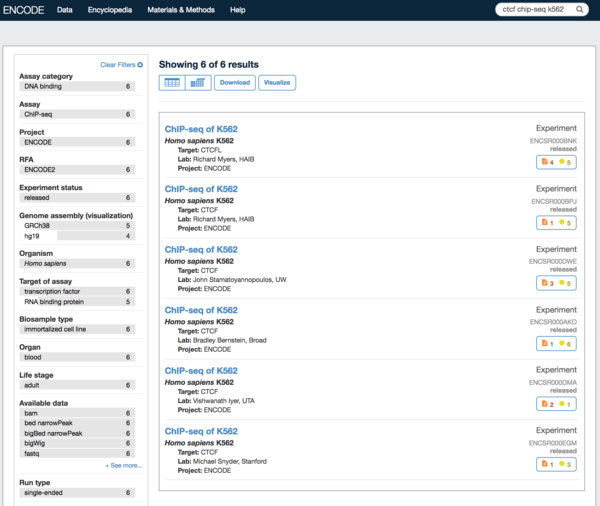
- 点击matrix,在搜索框搜索。比如“ctcf chip-seq k562” 。同时可以点击页面左方导航栏进行过滤。
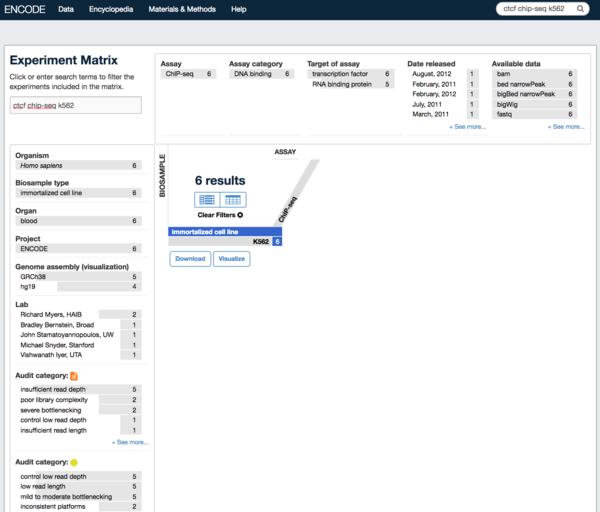
- 点击切换到列表模式。
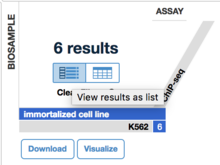
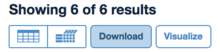
4. 点击页面中的Download,下载一个名为files.txt的文件。这个文件的第一行是页面中所有文件的metadata。从第二行开始就是下载链接了。使用wget或者curl可以直接下载。
方法二:写代码搜索后批量下载
可以发现方法一中的files.txt的URL有相同的模式(BASE_URL + QUERY)。
BASE_URL= 'https://www.encodeproject.org'
QUERY = '/files/[accession number]/@@download/[accession number].[format]'
那么对于一个Experiment,我们如何获取所有原始文件和结果文件(fastq,bam,bed等)的下载地址(QUERY)呢?
举例来说,我们点击方法一中搜索得到第一个实验(Experiment),accession number为ENCSR000BNK,可以通过在URL后面加上?format=json查看这个实验的所有metadata(JSON格式),页面地址为
https://www.encodeproject.org/experiments/ENCSR000BNK/?format=json
我们需要的下载链接(QUERY),就是其中的一个名为href的metadata。
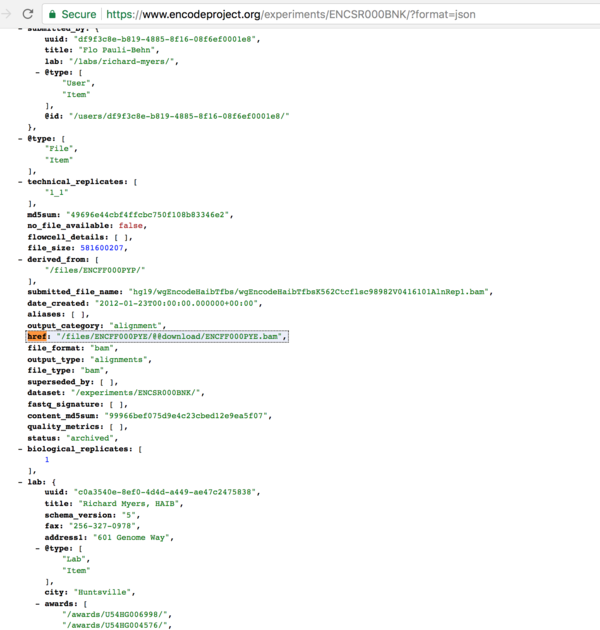
写代码搜索下载的实质就是通过RESTful API同ENCODE的metadata数据库交互,取得我们需要的metadata(以JSON的格式),然后获取文件下载地址“href”。
直接上代码,使用上述的ENCSR000BNK为例:
import requests, json
# 要求服务器返回JSON格式数据
HEADERS = {'accept': 'application/json'}
# BASE_URL是通用前缀,QUERY是定制的查询
# type=file 查询的是file类型的对象
# file_format=bed 指定下载文件内容
# dataset=/experiments/ENCSR000BNK/ 指定下载的Experiment accession number
# limit=all 不加这个参数只能返回前25个结果
# frame=object 获取object的全部metadata
BASE_URL = 'https://www.encodedcc.org/search/?'
QUERY ='type=file&dataset=/experiments/ENCSR000BNK/&file_format=bed&limit=all&frame=object'
# 用GET命令向服务器请求结果
response = requests.get(BASE_URL+QUERY, headers=HEADERS)
# 将JSON格式转换成python的字典dict格式。response_json_dict储存所有的metadata
response_json_dict = response.json()
# 打印查看结果
print(json.dumps(response_json_dict, indent=4, separators=(',', ': ')))
# 定义下载函数
def download_file(url):
local_filename = url.split('/')[-1]
r = requests.get(url, stream=True)
with open(local_filename, 'wb') as f:
for chunk in r.iter_content(chunk_size=1024):
if chunk:
f.write(chunk)
f.flush()
return local_filename
# 返回href信息并下载
for file_dict in response_json_dict['@graph']:
print(file_dict['href'])
fn = download_file(FILE_URL+file_dict['href'])
# /files/ENCFF002CLT/@@download/ENCFF002CLT.bed.gz
# /files/ENCFF653WEF/@@download/ENCFF653WEF.bed.gz
# /files/ENCFF678TFE/@@download/ENCFF678TFE.bed.gz
# /files/ENCFF001UJN/@@download/ENCFF001UJN.bed.gz
# /files/ENCFF001UJO/@@download/ENCFF001UJO.bed.gz
# /files/ENCFF081QMM/@@download/ENCFF081QMM.bed.gz
# /files/ENCFF036DBK/@@download/ENCFF036DBK.bed.gz
# /files/ENCFF943BRX/@@download/ENCFF943BRX.bed.gz
这样文件就下载到本地了。
参考信息:
ENCODE REST API:https://www.encodeproject.org/help/rest-api/
ENCODE DCC github:https://github.com/ENCODE-DCC