经过了第四节的长文,我想大家基本上已经知道了一个WGS流程该如何构建起来了吧。但在那一节中限于篇幅有两个很重要的文件我没能展开来讲,分别是:BAM和VCF文件。这篇我们先说BAM文件。
什么是BAM
BAM是目前基因数据分析中最通用的比对数据存储格式,它既适合于短read也适合于长read,最长可以支持128Mbp的超大read!除了后缀是.bam之外,有些同学可能还会看到.cram,甚至.sam后缀的文件,其实它们一个是BAM的高压缩格式(.cram)——IO效率比原来的BAM要略差;另一个是BAM的纯文本格式(.sam)。当然格式都是一样的,因此为了描述上的清晰,我下面都统一用BAM。
BAM文件格式
其实一开始它的名字是SAM(The Sequencing Alignment/Map Format的英文简称),第一次出现的时候,它是bwa比对软件的标准输出文件,但原生的SAM是纯文本文件,十分巨大(比如:一个人30x全基因组测序的sam大小超过600G),非常容易导致存储空间爆满!为了解决这个问题,bwa的开发者李恒(lh3)设计了一种比gz更加高效的压缩算法,对其进行压缩,这就是我们所说的BAM,它的文件大小差不多只有原来的1/6。
在2007年,NGS技术刚刚兴起之时,各类短序列比对软件层出不穷,输出格式也是各有特点,各家各有一套,并没有什么真正的标准可言,可以说那是一个谁都说我最好的时期。
但逐渐的,研究者们发现BAM格式对Mapping信息的记录是最全面的,用起来也是最灵活的。bwa的作者还为BAM文件开发了一个非常好用的工具包——Samtools,使得人们对BAM文件的处理变得十分便利,拓展性也变得非常强,后来还有类似于IGV等专门支持BAM的工具也越来越多,因此它就逐渐成为了主流。
现在基本上所有的比对数据都是用BAM格式存储的,俨然已经成为了业内的默认标准。
在2013年,研究者们还专门将Samtools的处理核心剥离出来,并将其打包成为一个专门用于处理高通量数据的API——htslib,除了C语言版本之外还有Java和Python版本,这些在github上都能直接找到。后续许多与NGS数据处理有关的工具基本都会使用这个API进行相关功能的开发,可见其影响力。
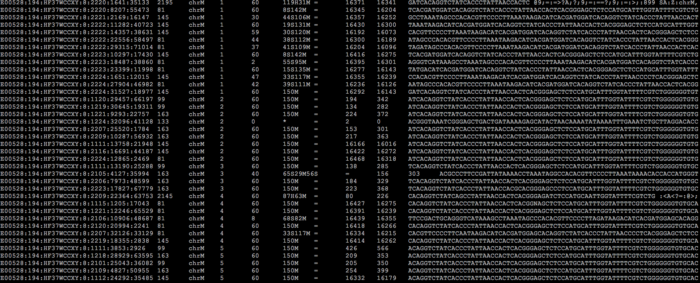
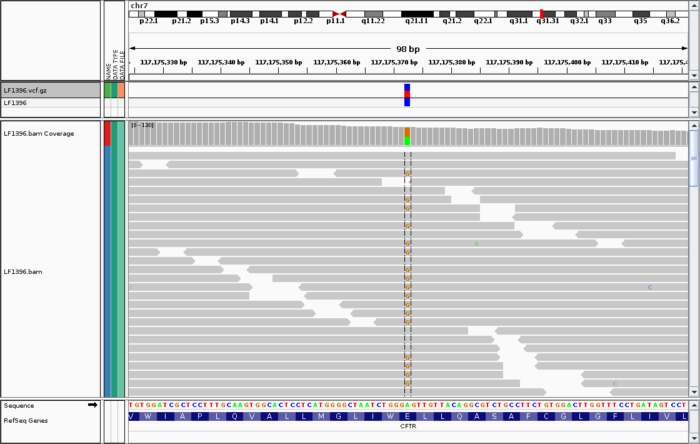
ok,背景的介绍就先到此为止了,我们回归主题。下面这个图是我从一份刚刚完成比对的bam文件中截取出来的内容:
由于屏幕所限,无法把全部的内容都包含进来,特别是header信息,贴在这里仅是为了让还没见过BAM文件的同学们能够对它有一个总体的感觉。
如果是SAM文件,同时你也熟悉linux操作的话,直接在linux终端用less打开即可(注意:不要试图在本地使用文本编辑器,如vim等直接打开文件,会撑死机子的),但如果我们要查看的是BAM,那么必须通过Samtools(可以到samtools的网站下载并安装)。
$ less -SN in.sam # 打开sam文件 $ samtools view -h in.bam # 打开bam文件
BAM文件分为两个部分:header和record。这里额外说一句,许多NGS组学数据的存储格式都是由header和record两部分组成的。
以上例子,在samtools view中加上-h参数目的是为了同时把它的header输出出来,如果没有这个参数,那么header默认是不显示的。
header内容不多,也不会太复杂,每一行都用‘@’ 符号开头,里面主要包含了版本信息,序列比对的参考序列信息,如果是标准工具(bwa,bowtie,picard)生成的BAM,一般还会包含生成该份文件的参数信息(如上图),@PG标签开头的那些。这里需要重点提一下的是header中的@RG也就是Read group信息,这是在做后续数据分析时专门用于区分不同样本的重要信息。它的重要性还体现在,如果原来样本的测序深度比较深,一般会按照不同的lane分开比对,最后再合并在一起,那么这个时候你会在这个BAM文件中看到有多个RG,里面记录了不同的lane,甚至测序文库的信息,唯一不变的一定是SM的sample信息,这样合并后才能正确处理。
其实,关于这一点我在上一篇文章中(引用第四节)讲序列比对时的也特意强调了这些方面,不记得的同学们也可以翻看上一篇的相关内容。
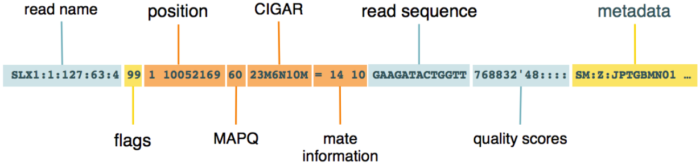
接下来重点要说的是BAM的核心:record(有时候也叫alignment section,即,比对信息)。这是我们通常所说的序列比对内容,每一行都是一条read比对信息,它的记录看起来是这样的:
我这里借用了网上的一张图片来辅助说明,recoed中的每一个信息都是用制表符tab分开的。
下面我们就来仔细瞧瞧这里的每一个信息分别都是什么。
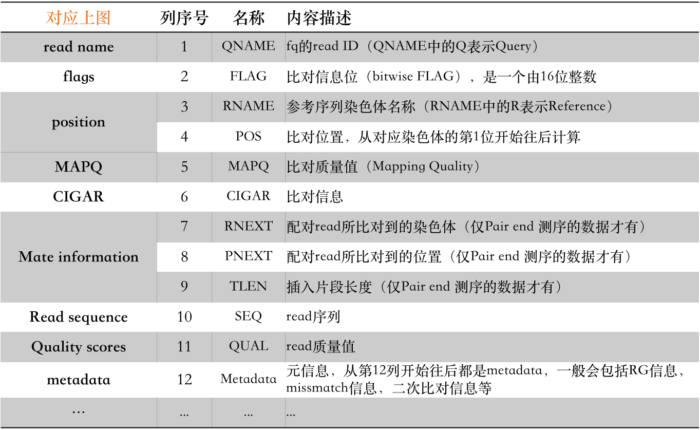
以上,前11列是所有BAM文件中都必须要有的信息,而且从描述中我们也能够比较清楚地知道其所代表的含义。但其中,有几个信息实在太重要了,以至于我认为有必要对其进行详细说明。
第一,Flag信息
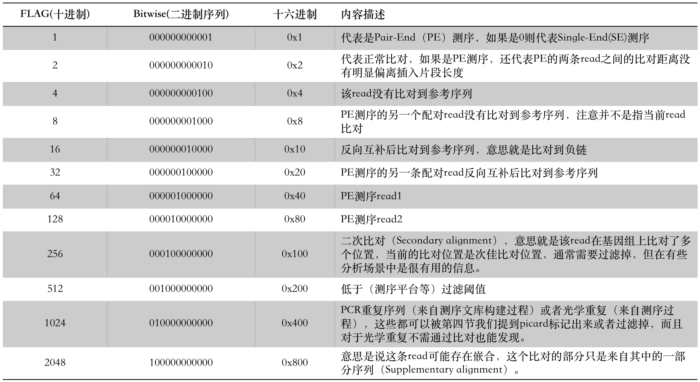
这是一个非常特别并且重要的数字,也是一个容易被忽视的数字,这可能和许多生信工程师也并不完全理解这个值有关。许多同学在第一次看到其官方文档中的描述之后依然会觉得十分困惑,但它里面实际上记录了许多有关read比对情况的信息。想要读懂它的一个关键点是我们不能够将其视为一个数字,而是必须将其转换为一串由0和1组成的二进制码,这一串二进制数中的每一个位(注意是“位”,bit的意思)都代表了一个特定信息,它一共有12位(以前只有8位),所以一般会用一个16位的整数来代表,这个整数的值就是12个0和1的组合计算得来的,因此它的数值范围是0~2048(2的12次方,计算机科学的同学对这种计算应该不陌生)。
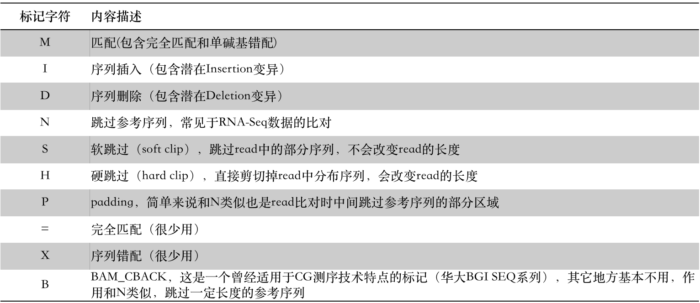
那么下面我就结合其文档和自己的实践经验对这12个位的含义用更加通俗易懂的语言来重新描述,如下表:
所以,通过上面这个表的信息,我们就可以清楚地知道每一个FLAG中都包含了什么信息。比如看到FLAG = 77时,我们第一步要做的就是将其分解为二进制序列(也可以理解为分解成若干个2的n次方之和):
77 = 000001001101 = 1 + 4 + 8 +64,这样就得到了这个FLAG包含的意思:PE read,read比对不上参考序列,它的配对read也同样比不上参考序列,它是read1。
当然,如果你希望自己在程序中写一段处理FLAG的代码,那么显然是不会像我们这个例子那样去分解这个整数的,多麻烦啊!那么该如何做呢?其实也很简单,比如我们 要获得其中某个位(假设第N位)的值——只需要将这个FLAG值和2的N次方做与的运算即可。在与运算时,FLAG值首先会被转换成一串二进制序列(如77=000001001101),而2的N次方除了第N位是1之外,其它的都是0,“与”了之后其它信息就会被屏蔽掉。比如,我们想知道该read是否比对上了参考序列,那么只需要计算FLAG & 4 的值就行了,如果结果是1那么就是比对上了,如果是0则代表没有比上。
不过,在实际工作中,除非遇到特殊的情况,否则我一般更推荐调用官方的htslib这个包来协助处理,它是一个C语言库,如果你用Python,则是pysam——htslib的python包(Java则是htsjdk),包中已经帮我们做了这些处理,可以直接得到结果,下一篇文章里我会用pysam举例说明如何用它来操作bam文件。
另外,下面这一段代码是htslib(samtools的核心库)中定义的12个与flag值进行与操作获取对应位信息的变量,感兴趣的同学可以再htslib里面的sam.h文件中找到,在做一些需要触达基础性原理的开发时或许你会用到。
/*! @abstract the read is paired in sequencing, no matter whether it is mapped in a pair */ #define BAM_FPAIRED 1 /*! @abstract the read is mapped in a proper pair */ #define BAM_FPROPER_PAIR 2 /*! @abstract the read itself is unmapped; conflictive with BAM_FPROPER_PAIR */ #define BAM_FUNMAP 4 /*! @abstract the mate is unmapped */ #define BAM_FMUNMAP 8 /*! @abstract the read is mapped to the reverse strand */ #define BAM_FREVERSE 16 /*! @abstract the mate is mapped to the reverse strand */ #define BAM_FMREVERSE 32 /*! @abstract this is read1 */ #define BAM_FREAD1 64 /*! @abstract this is read2 */ #define BAM_FREAD2 128 /*! @abstract not primary alignment */ #define BAM_FSECONDARY 256 /*! @abstract QC failure */ #define BAM_FQCFAIL 512 /*! @abstract optical or PCR duplicate */ #define BAM_FDUP 1024 /*! @abstract supplementary alignment */ #define BAM_FSUPPLEMENTARY 2048
第二,CIGAR
CIGAR是Compact Idiosyncratic Gapped Alignment Report的首字母缩写,称为“雪茄”字符串。
作为一个字符串,它用数字和几个字符的组合形象记录了read比对到参考序列上的细节情况,读起来要比FLAG直观友好许多,只是记录的是不同的信息。比如,一条150bp长的read比对到基因组之后,假如看到它的CIGAR字符串为:33S117M,其意思是说在比对的时候这条read开头的33bp在被跳过了(S),紧接其后的117bp则比对上了参考序列(M)。这里的S代表软跳过(Soft clip),M代表匹配(Match)。CIGAR的标记字符有“MIDNSHP=XB”这10个,分别代表read比对时的不同情况:
除了最后‘=XB’非常少见之外,其它的标记符通常都会在实际的BAM文件中碰到。另外,对于M还是再强调一次,CIGAR中的M,不能觉得它代表的是匹配就以为是百分百没有任何miss-match,这是不对的,多态性碱基或者单碱基错配也是用M标记!
第三,MAPQ,比对质量值
这个值同样非常重要,它告诉我们的是这个read比对到参考序列上这个位置的可靠程度,用错误比对到该位置的概率值(转化为Phred scale)来描述:
。
因此MAPQ(mapping quality)值大于30就意味着错比概率低于0.001(千分之一),这个值也是我们衡量read比对质量的一个重要因子。
剩下的几列在上面的格式表中描述的也比较清楚,基本没有过于隐藏的信息,因此我就不打算再一一细说了,如果大家依然有困惑可以到后台留言。
此外,细心的同学可能也已经发现了:fastq的所有信息都被涵盖到了BAM文件中了,包括比对不上的read也在,因此获得了BAM其实也等于获得了所有的read。而且,fastq有时也会被转换成一种uBam文件,指的就是un-mapping BAM——没有做过比对的BAM文件。它相比于Fastq可以用metadata存储更多有用的信息,不过这不是我们这篇文章想说的内容。
最后,还是再说明一次:BAM文件中除了必须的前11列信息之外,不同的BAM文件中后面记录metadata的列是不固定的,在不同的处理软件中输出时也会有所不同,我们也可以依据实际的情况增删不同的metadata信息。
使用samtools view查看BAM文件
BAM文件由于是特殊的二进制格式,因此没办法通过文本的形式直接打开,要用samtools的view功能在终端上进行查看(上文也已经说到这里在进行系统补充),如:
samtools view in.bam
如果不想从头开始看,希望快速地跳转到基因组的其它位置上,比如chr22染色体,那么可以先用samtools index生成BAM文件的索引(如果已经有索引文件则不需该步骤),然后这样操作:
$ samtools index in.bam # 生成in.bam的索引文件in.bam.bai $ samtools view in.bam chr22 # 跳转到chr22染色体 $ samtools view in.bam chr22:16050103 # 跳转到chr22:16050103位置 $ samtools view in.bam chr22:16050103-16050103 # 只查看该位置
IGV或者samtools tview查看比对情况
以上,我基本上列举了我们会在终端上如何查看BAM文件的几个最常用操作。但如果你想更直观查看的BAM文件,IGV是目前最好的一个选择,但仅适合于文件还比较小的情况,效果如下:
如果你的BAM文件很大,都超过了你的本地电脑磁盘了,你还是想看该怎么办?你有两个选择:
第一,把你想查看的那部分区域用samtools view提取出来,生成一份小一些的BAM,然后下载下来,在导入到IGV中。
$ samtools view -h in.bam chr22:16050103-16050203 | samtools view -Sb - > small.bam
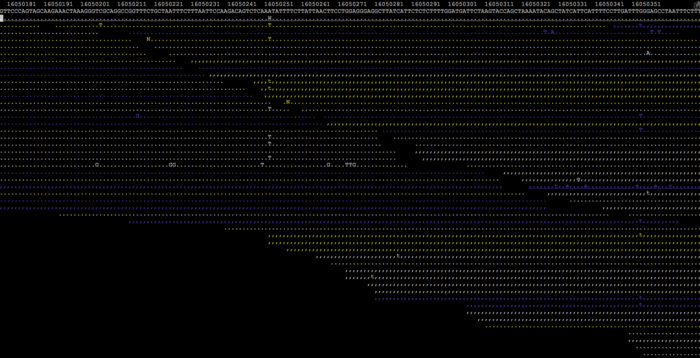
第二,不下载,直接在终端用samtools tview进行查看。samtools tview有类似于IGV的功能,虽然体验会稍差一些。
$ samtools tview --reference hg38.fa in.bam
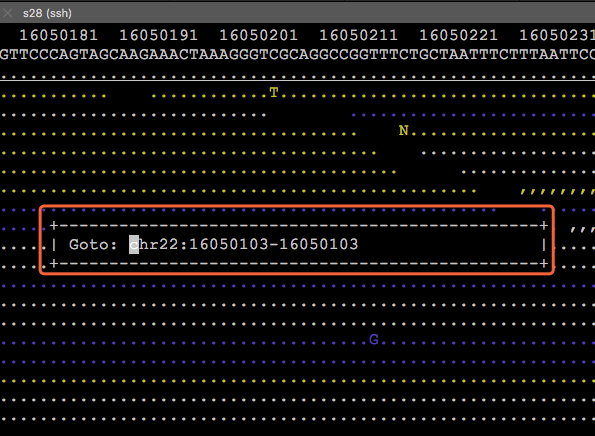
在该模式下,按下键盘‘g’后,会跳出一个Goto框,在里面输入想要调整过去的位置,就行了,比如:
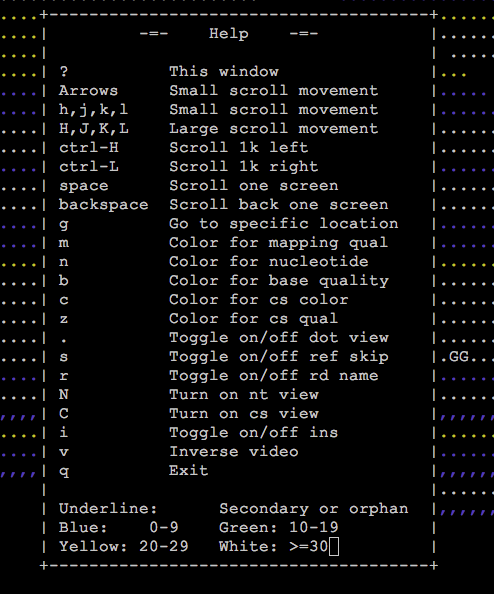
按下esc键则可以取消。另外,为了节省空间,加快查询效率,read中与参考序列相同的部分被用一串串不同颜色的点表示,只留下miss-match的碱基和发生indel变异的区域。其中圆点表示正链比对,逗号表示负链比对。不同的颜色代表不同的比对质量值:白色>=30,黄色20-29,绿色10-19,蓝色0-9。如果你还想知道的其他的功能,可以在tview模式里按下“?”问号,就会弹出类似下面这样的帮助窗口,然后按照指引做就行了。
虽然看起来不如IGV体验那样好,功能也比较单一(仅可以查看比对情况),但可贵之处在于可以在终端里面直接操作,当需要快速查看某个位置的比对情况时,操作效率非常高。而如果要退出该模式,也非常简单,按下q键就可以了。
小结
那么,有关BAM格式的内容我们就暂且先到这里吧,大家如果有疑惑或者感兴趣的内容都可以到后台留言,我都会定时进行回复。在下一篇文章中,我们将重点介绍如何使用pysam来操作bam文件了。