简介
1、概述
BEDTools是可用于genomic features的比较,相关操作及进行注释的工具。而genomic features通常使用Browser Extensible Data (BED) 或者 General Feature Format (GFF)文件表示,用UCSC Genome Browser进行可视化比较。该工具的主要功能如下图
2、与BEDTools使用相关的基本概念
已有的一些genome features信息一般由BED格式或者GFF格式进行存储。
- genome features: 功能元素(gene), 遗传多态性 (SNPs, INDELs, or structural variants), 已经由测序或者其他方法得到的注释信息,也可以是自定义的一些特征信息。
- genome features的基本信息: 染色体或者scaffold的位置, 起始位置,终止位置,哪条链,feature的name
- Overlapping / intersecting features: 两个genome features的区域至少有一个bp的共同片段
3、BED和GFF文件的一个差异
BED文件中起始坐标为0,结束坐标至少是1,; GFF中起始坐标是1而结束坐标至少是1。
安装
注意:下面的代码中是需要下载的工具的版本号
[shell]
curl http://bedtools.googlecode.com/files/BEDTools.***<version>***.tar.gz > BEDTools.tar.gz
tar -zxvf BEDTools.tar.gz
cd BEDTools
make clean
make all
ls bin
cp /bin/* usr/local/bin/
[/shell]
一些简单的使用
1、从两个BED文件中得到genome feature的交集
intersectBed -a reads.bed -b genes.bed
2、从两个BED文件中得到只在第一个文件中有而不在第二个文件中的genome features
intersectBed -a reads.bed -b genes.bed -v
相关格式
1) BED format
BEDTools主要使用BED格式的前三列,BED可以最多有12列。BED格式的常用列描述如下:
- chrom: 染色体信息, 如chr1, III, myCHrom, contig1112.23, 必须有
- start: genome feature的起始位点,从0开始, 必须有
- end: genome feature的终止位点,至少为1, 必须有
- name: genome feature的官方名称或者自定义的一个名字
- score: 可以是p值等等一些可以刻量化的数值信息
- strands: 正反链信息
2) BEDPE format
可以用于描述不连续的genome features, 例如structural variations或者paired-end sequence alignments。和BED文件格式相比,就是一个记录中要有两个chrom, start,end
3) GFF format
类似于BED
4) genome files
BEDTools中的一些工具(genomeCoverageBed, complementBed, slopBed)需要物种的染色体大小的信息,genome file一般就是每行都是tab隔开,两列,一列为染色体的名字,第二列为这个染色体的大小。一般常用物种的genome file在BEDTools安装目录的/genome里面。
5) SAM/BAM format
BEDTools的两个工具:intersectBed, pairToBed支持BAM格式的输入和输出。有两个工具有助于:
- Find BAM alignments that overlap (or not) with BED annotation and report them in BED format
- Create a new BAM file of BAM alignments that overlap (or not) with BED annotations. This serves as a powerful way to refine alignment datasets based on biological interest.
BEDTools suite使用详细
1、intersectBed
用来求两个BED或者BAM文件中的overlap,overlap可以进行自定义是整个genome features的overlap还是局部。
默认的结果描述如下图
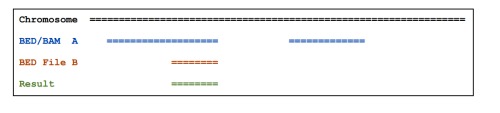
加-wa参数可以报告出原始的在A文件中的feature, 如下图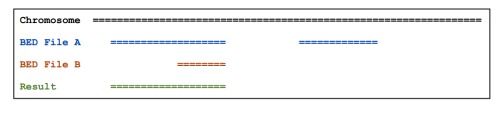
加-wb参数可以报告出原始的在B文件中的feature, 加-c参数可以报告出两个文件中的overlap的feature的数量, 参数-s可以得到忽略strand的overlap。
示例:
intersectBed -a A.bed -b B.bed intersectBed -a A.bed -b B.bed -wa intersectBed -a A.bed -b B.bed -wb intersectBed -a A.bed -b B.bed -wa -wb intersectBed -a A.bed -b B.bed -c intersectBed -a A.bed -b B.bed -f 0.50 -r -wa -wb
2、pairToBed
将BEDPE或者paired-end的BAM文件与BED文件进行比较,搜索overlaps。默认结果如下图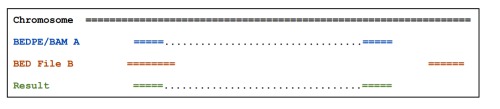
加-type both报告A中两个end都在B中存在overlap的;加-type notboth报告A中任一end都在B中存在overlap的; -type ispan报告A的pair是否是横跨B的两侧,还有类似的-type ospan, -type notispan;加参数-f可以指定最小的overlap的比例,超过这个比例则报告;参数-s可以得到忽略strand的overlappairToBed -a A.bedpe -b B.bed -type bothpairToBed -a A.bedpe -b B.bed -f 0.5pairToBed -abam pairedReads.bam -b simreps.bed -bedpe
- pairToPair
比较BEDPE文件搜索overlaps, 类似于pairToBed。
- bamToBed
将BAM文件转换为BED文件或者BEDPE文件。bamToBed -i reads.bam
- windowBed
类似于intersectBed, 但是可以指定一个数字,让A中的genome feature增加上下游去和B中的genome features进行overlap。默认情况这个值为1000,可以使用-w加定义,可以用-l指定是上游,用-r指定下游windowBed -a A.bed -b B.bed -w 5000
windowBed -a A.bed -b B.bed -l 200 -r 20000
- subtractBed
在A中去除掉B中有的genome features
- coverageBed
加-s参数表明根据正负链计算
coverageBed computes both the depth and breadth of coverage of features in file A across the features in file B. For example, coverageBed can compute the coverage of sequence alignments (file A) across 1 kilobase (arbitrary) windows (file B) tiling a genome of interest. One advantage that coverageBed offers is that it not only counts the number of features that overlap an interval in file B, it also computes the fraction of bases in B interval that were overlapped by one or more features. Thus, coverageBed also computes the breadth of coverage for each interval in B.
genomeCoverageBed
genomeCoverageBed computes a histogram of feature coverage (e.g., aligned sequences) for a given genome. Optionally, by using the –d option, it will report the depth of coverage at each base on each chromosome in the genome file (-g ).
软件相关论文:
参考来源:http://caoyaqiang.diandian.com/post/2012-09-12/40039807769




1F
这个网站真是牛,学到了很多,非常感谢楼主的分享!!!
不知道楼主用没用过GSC(Genome Structrue Correction)的软件,感觉和bedtools好像挺相似的,不知道有没有类似的资料分享下?
B1
@ bioaggressor 非常抱歉,目前还没有Genome Structrue Correction相关的资料,如果后续有相关的资料会放到PLoB上来的
B2
@ ybzhao 哦,好的,看过GSC的原paper,太难懂了,但是又感觉和bedtools很像。。感觉这个网站做的真心好,哈哈
2F
版主好,我用bedtools getfasta遇到了WARNING. chromosome (chr01) was not found in the FASTA file. Skipping. 这样的问题。bed文件和fasta文件中的染色体名称对了好几遍,实在没发现有空格或者其他的什么不同之处。国外论坛例子不多,而且好像都没有解决就停贴了。望解答,谢谢!
B1
@ joyjane88 ,我用bedtools getfasta遇到了WARNING. chromosome (chr01) was not found in the FASTA file. Skipping. 这样的问题。bed文件和fasta文件中的染色体名称对了好几遍,实在没发现有空格或者其他的什么不同之处。国外论坛例子不多,而且好像都没有解决就停贴了。望解答,谢谢! 你好,你这个怎么解决的?可以联系我18404982559
3F
楼主,你好。问一下将多个bed文件整合在同一个bed文件中,怎么整理。整理的时候,报错显示是,It looks as though you have less than 3 columns at line: 1. Are you sure your files are tab-delimited?
这是什么意思啊?