唐碧霞 王彦青 陈旭 庞博 赵文明
(中国科学院北京基因组研究所,北京,100029)
摘 要 高通量测序技术的发展使得测序数据量大规模的增长,从而为研究人员的数据分析工作带来了 挑战。基因组可视化工具使用图形界面的方式将数据进行可视化,从而在一定程度上加快了数据分析 速度。本文介绍了一种比较常用的基因组可视化工具GBrowse,并结合实际描述了GBrowse在水稻基因 组数据库的应用。最后本文总结了基因组可视化工具发展所面临的挑战。
关键词 基因组,可视化,GBrowse
1.引言
随着第二代高通量的测序技术的发展,测序通量在以超过摩尔定律增长趋势快速增 长,而成本却直线下降,这无疑对科学家通过分子水平开展科学研究提供了一个最有力 的支持和帮助。但海量的数据对从事生物信息分析的人员却提出了巨大的挑战,如何及 时、高效并准确的处理和分析这些数据,也是生物信息工作者开口必谈的话题。
在生物信息数据分析的众多过程中,如序列拼接、序列比对、SNP检查、表达量分析 等,都出现了一些专业的、自动化的软件工具,用于帮助研究人员进行数据分析,从而 大大提高了数据分析的效率,但是数据分析结果的正确有效性,仍依赖于研究人员的人 工参与,而面对杂乱无章的数据文件时,人工参与的效率往往较低。相比之下,图形或 图表能很直观的表示数据特征,更易于被人阅读和理解,如果能将分析结果数据以图形 或图表的方式进行可视化,并提供一些交互性操作界面,将会极大的提高数据分析效率。
目前,已经有许多应用于生物数据分析不同阶段的基因组可视化工具可供科研工作者选择[1],如在拼接领域的Consed[2]等,在基因组序列领域的GBrowse[3]、JBrowse[4]等, 在比较基因组领域中的Circos[5]等,在国际上,也出现了一些专门的基因组浏览器,如NCBI的Map viewer[6]、UCSC的UCSC Genome Browser[7],这些基因浏览器除提供数据的可视化功能外,还与公共数据库的应用结合在一起,从而为用户提供更多有用的数据信息。
本文详细介绍了GBrowse基因组浏览器的功能、特点,并在后面结合实际应用案例介绍了GBrowse的使用方法。
2.GBrowse 介绍
GBrowse是Genetic Model Organism Database(GMOD) Project[8]开发的一个基于Web 的基因组浏览器工具,因其灵活的定制功能,而被广泛使用。目前有许多模式生物数据 库使用GBrowse构建了自己的基因组浏览器,如小鼠、果蝇、NCBI的HapMap等等。GBrowse 基因组浏览器的基本功能是提供一个可视化的基因组浏览界面,该界面是一个以序列长 度作为横坐标,以各数据项作为纵坐标的二维显示界面,目前支持基因组序列以及基因、 SNP等常见注释数据的显示。
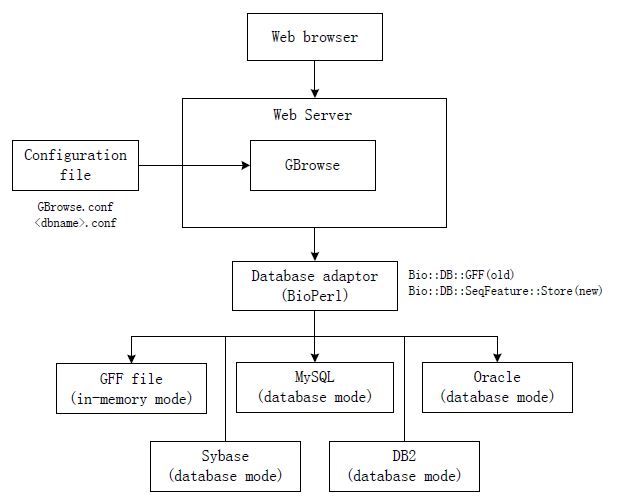
图1是GBrowse的架构图。
图 1 GBrowse 架构图
GBrowse的浏览界面是基于WWW的,需要Apache服务器的支持。GBrowse收到用户的数 据请求后,会先根据请求的数据集名从GBrowse.conf配置文件中找到该数据集对应的配 置文件<dbname>.conf,接着从该配置文件指定的数据库中取出对应的数据,并将其按照 配置的数据显示方式进行显示。从上述这个过程可以看出,GBrowse的运行与配置文件具 有很大的关系,基于配置文件的机制,使得GBrowse具有较好的扩展性。
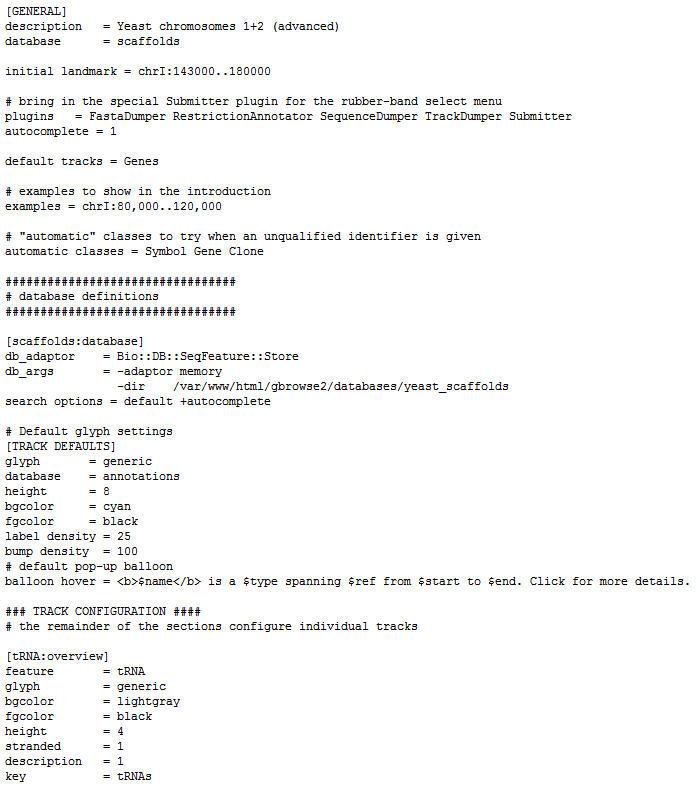
GBrowse配置文件分为两种,一种是全局的配置文件GBrowse.conf,该配置文件的配 置项将应用于所有的数据集,另一种是各数据集自己的配置文件<dbname>.conf,由开发 人员根据需要按给定格式自行编写。另外,由于GBrowse是基于perl语言实现的,开发人 员可以在配置文件中编写perl脚本从而实现更为复杂的功能。一个比较简单的GBrowse 数据集的配置文件如下图2所示。
GBrowse数据集配置文件可对界面显示语言、界面整体布局、界面提示信息、搜索方 式(精确搜索/模糊搜索/全文检索)、数据项、数据项的显示外观等进行配置。
GBrowse支持文件和数据库两种数据存储方式,提供了多种数据库类型如MySQL、DB2、 Oracle、Sybase等,开发人员可以根据自己的数据量以及数据类型,进行数据库的选择, 在配置文件中只需设置相应的数据库适配器即可。一般来说,GFF[9]文件内存模式支持小 于2万条的数据记录,Mysql数据库则支持千万条以上的数据库记录。
一个一般的GBrowse基因组浏览器的开发过程如下:
1)根据实际的数据量和数据类型,选择一种合适的数据库存储模型,如Mysql
2)将实际数据生成GFF格式的数据文件,使用提供的数据装载perl脚本,将数据装 载进数据库中
3)编写该数据库的配置文件如dbname.conf,并配置各项
4)在GBrowse.conf文件加入该数据库的配置文件,并为该数据库命名一个数据集 名
5)在浏览器中输入该数据集名url地址,即可访问。不需要重启Apache。
GBrowse发展到现在,经过了从1.x到2.0版本的发展,并且仍在持续更新,支持多种 应用平台如windows、linux等。GBrowse2.0在数据获取与更新方面使用Ajax技术,较1.x 版本能给用户带来更好的体验。此外,GBrowse2.0还支持数据的分布式架构,只需在各从节点上安装slave模块,即可达到数据分布的目的,从而为数据的集成提供了方便。
3.GBrowse2.0 应用
系统生物学中多组学综合数据库是中国科学院北京基因组研究所开发的一个集基因 组学、转录组学等组学领域数据为一体的综合性数据库系统,目前主要集成了水稻、家 鸡、家蚕、流感病毒的基因组数据,其中,水稻、家鸡、家蚕数据库都提供了基于GBrowse2.0 的基因组数据浏览方式,从而方便用户根据自己的需求定制浏览数据。下面本文将详细 介绍GBrowse2.0在水稻基因组中的简单应用,关于GBrowse2.0更多配置项的应用,用户 可到GBrowse网站查阅相关信息。
水稻基因组数据库包括两个水稻亚种即indica 9311与syngenta japonica,其中 indica 9311含有部分转录组数据。indica 9311与japonica都包含Scaffold、Gene等数 据类型,但各数据类型由于个体的差异性,其具体的数据内容有所不同。如果能在同一界面上提供两个水稻亚种的不同数据类型的序列数据或者基因结构的可视化显示工具,并能提供基于染色体坐标位置的数据范围定位操作,则用户可以很方便的了解两株水稻 的差异性。基于此目的,使用GBrowse2.0建成的水稻基因组浏览器集成了两个水稻亚种 的Scaffold、Gene以及序列的GC含量等数据,用户可以根据自己的需求选择想要查看的 数据类型,并比较两株水稻的差异性。
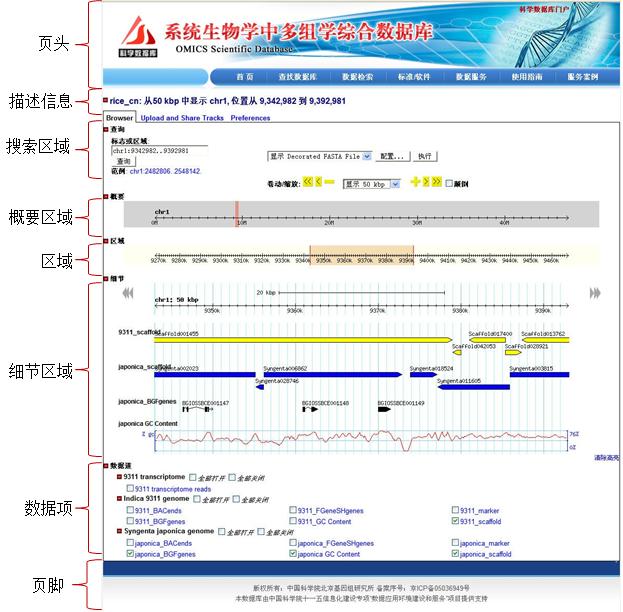
水稻基因组浏览器的界面如下图3所示。以下将结合水稻的GBrowse配置文件对如何 实现该浏览界面进行说明。
3.1 页头与页脚等基本项的设置
GBrowse支持中、英文等多种语言的界面显示,只需配置相应的语言选项即可。对于 显示页面的页头与页脚,开发人员可以根据自己的需要自行配置,页头与页脚支持常见的html标签,并支持样式表。整个页面的显示宽度、背景颜色等都可以自行配置。水稻基因组浏览器的页头、页脚等的配置样例如下图4所示,其中stylesheet指定了页面使用 的css文件。如果是自行编写的样式文件或自己的图片要先上传到GBrowse安装目录下的 images与css文件夹下。所有配置项的格式均为“配置项名 = 配置值”的格式。
图4水稻基因组浏览器基本项配置
3.2 数据的浏览
水稻基因组浏览器是以水稻染色体序列长度为坐标,其他各种数据类型如Scaffold等的坐标位置都是相对于染色体的。 对于序列数据浏览,GBrowse界面上给出不同粒度上的数据浏览区域。概要区域是整个序 列长度范围的数据显示区域,序列长度坐标根据用户在GFF文件中指定的长度自动显示。 区域是比较细粒度范围的数据显示范围,可根据用户选择的区域范围进行变化。而细节 区域则是更细粒度范围的数据显示,最小可以显示出碱基序列。开发人员可以根据自己 的需要设置数据项的显示区域。区域一般放序列相对较长的数据项,如基因、转录,或 者需要在较大粒度范围内查看的数据项,如SNP每20kp的密度分布等,而细节区域则放 序列相对较短的数据项如SNP、Reads,或者需要在细粒度范围内查看的数据项,如基因 的碱基序列信息、GC含量分布等。
GBrowse中可以配置各区域显示的数据粒度参数,水稻基因组浏览器关于数据浏览界 面的配置如下图5所示,这些配置项说明了水稻基因组浏览器的区域能显示的数据粒度为200kb,细节区域最大能显示的数据粒度为50Mb,用户可以进行放大或缩小的数据粒度从100bp到10Mb,最多能显示的检索结果为1000个等信息。
3.3 数据定位
水稻基因组浏览器界面提供多种方式供用户定位数据查看区域。搜索区域中基于搜 索输入框的数据查看方式,除了能基于位置范围搜索数据外,还能输入数据项名称如BGIOSSBCE017670进行精确检索,支持名称模糊检索,需在数据集中进行配置。
基于染色体坐标位置的点选方式,能快速定位想查看的数据范围。基于中心区域的范围选择框能快速显示某一区域范围内的数据,当中心区域设置为100bp时,界面上将会 显示实际的碱基序列。另外,辅助的坐标左移、右移、放大、缩小功能方便的支持数据范围的切换,这些功能在GBrowse安装后就具备了,不需要进行配置。
3.4 数据集
水稻基因组浏览器集成了两个数据集即indica 9311与syngenta japonica,GBrowse支持将多个数据集显示在一个界面上,但是必须给出数据集配置。在数据集配置中,每 一个数据集配置以[数据集名:database]开始,接着需要配置所使用的数据库适配器 db_adaptor,以及数据库访问参数选项db_args,在该db_args中需指定具体的数据库类 型(adaptor)、数据库名(dsn)、数据库访问用户名(user)、数据库访问密码(pass),以 及对于该数据库中的数据使用什么检索策略(search options)。
图6给出了水稻基因组浏览器的数据集配置项,从图中可以看出,水稻基因组浏览器 配置了newrice与syngenta两个数据集,使用的数据库类型为mysql,搜索数据策略使用 的默认全库检索类型,并支持输入框输入的自动提示功能。
图 6 水稻基因组浏览器的数据集配置
3.5 数据项
水稻基因组浏览器的各数据项使用图标和颜色进行了标示。GBrowse中数据项都是基 于图标显示的,一共有80多种图标供开发人员选择。有一些图标对应着特定的数据项,如gene图标对应着Gene数据,只需将Gene结构数据整理成良构的GFF文件格式即可,GBrowse将自动画出Gene的结构图。数据项配置中,支持使用不同的颜色、字体、高度、 超链接、提示信息以及数据项名字、出现面板区域等基本配置选项,其中最为重要的配置选项为feature与glyph,feature描述了要使用的具体数据(在GFF文件有对应的 feature列),而glyph则描述了数据显示使用的图标。其中一些通用数据配置选项可以写 在[TRACK DEFAULTS]选项中。每一个数据项都以[数据项名]开始。
图7给出了水稻基因组浏览器中默认配置项以及Gene、Scaffold的配置内容,对于9311_scaffold数据项表达的意思是从newrice数据集中获取feature为supercontig的数 据行,并将数据以黄色的矩形显示(矩形高度为10px),且标示出正负链信息,该数据项 应显示在数据项区域中的indica 9311 genome目录中,在细节区域中以9311_scaffold 进行标识,效果见图3细节区域中的9311_scaffold。
图 7 水稻基因组浏览器的数据项配置
4.总结
基因组可视化工具是生物学领域一个比较重要的数据辅助分析工具,到目前为止, 已有许多满足各种不同需求的可视化工具被开发出来并被使用。本文介绍了GBrowse的功 能、特点,并结合实际给出了GBrowse在水稻基因组浏览器中的简单应用。随着测序技术 的发展,如何在支持大规模数据量的情况下,提高数据的显示速度,提供更加友好的用 户体验,是可视化工具的开发一个挑战。另外,随着生物学研究领域的发展,可视化工 具也需要相应的支持越来越多的数据类型,如疾病的临床信息,如何将这些多种多样的 数据类型以更好的方式在一个界面上显示出来,也是基因组可视化工具所面临的另外一 个挑战。
参考文献
[1] Nielsen, C. B. et al., Visualizing genomes: techniques and challenges, Nature methods, Vol. 7, No. 3s, Mar. 2010
[2] Gordon, D., Abajian, C. & Green, P., Consed: a graphical tool for sequence finishing, Genome Res., Vol. 8, No. 3, Mar. 1998.
[3] Donlin,M.J., Using the Generic Genome Browser(GBrowse),Curr. Protoc. Bioinformatics, Chapter 9, Unit 9.9.,Dec. 2009.
[4] Skinner, M.E., Uzilov, A.V. et al., JBrowse: a next-generation genome browser, GenomeRes., Vol. 19, No. 9, Sep. 2009.
[5] Krzywinski, M. et al., Circos: an information aesthetic for comparative genomics, GenomeRes., Vol. 19, No. 9, Sep. 2009.
[6] http://www.ncbi.nlm.nih.gov/mapview/ [7] http://genome.ucsc.edu/
[8] http://gmod.org/wiki/Main_Page
[9] http://gmod.org/wiki/GFF
Introduction to a genomic visualization tool GBrowse and its application
Tang Bixia, Wang Yanqing, Chen Xu, Pang Bo, Zhao Wenming
(Beijing Institute of Genomics, Chinese Academy of Sciences, Beijing, 100029)
Abstract The advent of High Throughput Sequencing (HTS) technologies generates large volumes of data, which brings a challenge to the computational analysis on the sequencing data. Genomic visualizat ion tools use a graphic interface to represent the data and could make the procedure of data analysis more efficiently to some extent. This paper introduces GBrowse, a common genomic visualization tool, and describes an application of rice genome database which uses GBrowse to show the data. Finally, the paper discusses the challeng es about the development of genomic visualization tools.
Key Words Genome; Visualization; GBrowse