在基因组de novo组装后,为了进一步提高组装连续性经常对初步组装结果进行scaffolding。这时候可以直接利用手头已有的二代PE/MP/BAC文库测序(SSPACE)、三代单分子测序(SSPACE-LongRead、LRScaf)甚至转录组数据(L_RNA_scaffolder)。但是仅靠这些通常很难在保证准确性的前提下大幅度提升基因组连续性,因此通常不得不投入更多经费加测10X genomics、BioNano光学图谱或者Hi-C数据。
如果研究的物种已有较高质量的参考基因组时,比如水稻、拟南芥、人等常见模式生物,且又不关心测序个体可能存在的结构变异时,可以直接利用参考基因组scaffolding到染色体水平(依赖参考基因组的连续性)。这里整理了几个可以利用参考基因组进行scaffolding的一些工具。
Ragout
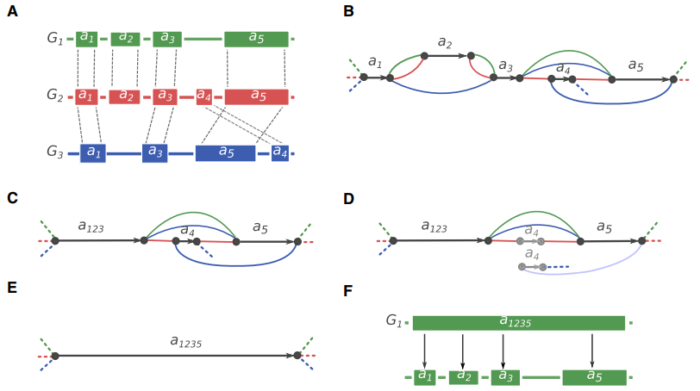
Ragout(Reference-Assisted Genome Ordering Utility)是一款可以利用一个或多个参考基因组将初步组装结果进行染色体水平scaffolding的工具,支持Linux以及Mac OS。Ragout目前更新到第2版,主要增加了对大型复杂基因组的支持,论文发表在Genome Research上。其基本原理是通过调用Cactus进行基因组比对得到共线性blocks构建A-Bruijn graph,再重构成染色体水平的基因组。
- GitHub:https://github.com/fenderglass/Ragout
- Reference:Kolmogorov et al., "Chromosome assembly of large and complex genomes using multiple references", Genome Research, 2018
PyScaf
第一次了解到PyScaf是作为Redundans的依赖之一。PyScaf的基因组比对使用LAST进行,运行速度非常快——可以在几分钟内完成拟南芥基因组的scaffolding(16 cpus),并且可以支持20%以内相对于参考基因组的碱基差异。此外Pyscaf除了可以利用参考基因组(reference-based)以外,还可以使用二代PE/MP(NGS-based)以及PacBio、Nanopore三代测序数据(long-reads-based)进行scaffolding。
Chromosomer
Chromosomer与PyScaf和Ragout整理类似,都是先共线性比对后重构基因组。首先通过fragmentmap可以调用blastn将初步组装的基因组和参考基因组进行比对,并将比对结果转化为fragment map。然后assemble命令可以根据fragment map重构scaffolding后的最终基因组。
值得一提的是,Chromosomer自带了transfer功能,可以把基于原版基因组的注释结果转换为scaffolding后的坐标。或者使用fragmentmapbed将fragment map转为bed格式。通过bed文件在genome browser里面检查scaffolding的正确性,同时理论上就可以使用UCSC的LiftOver转换坐标了(我还没有测试过)。不过如果后续还要再做gap filling或者polishing这类改变基因组序列的操作,建议还是得到最终基因组之后再做注释吧!
- GitHub:https://github.com/gtamazian/chromosomer
- Reference:Tamazian G, et al., "Chromosomer: a reference-based genome arrangement tool for producing draft chromosome sequences", GigaScience, 2016
ALLMAPS
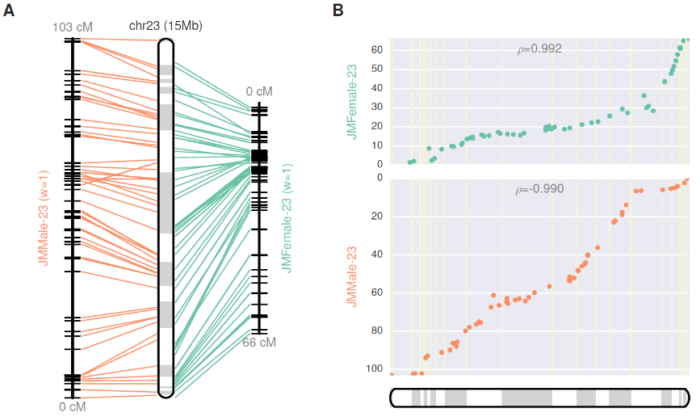
ALLMAPS是一个很常用的可以将一个或者多个遗传谱图构建染色体水平基因组的工具。除了遗传图谱外,也可以使用物理图谱和基因组比对的图谱。使用方法也很简单:首先自行使用LAST/NUCmer等软件先进行基因组比对,然后将比对的结果整理成和遗传谱图类似的csv文件就可以使用了(需要自己能编程处理比对格式)。ALLMAPS也可以生成一个.chain文件,使用LiftOver可以将基因组的坐标/注释信息进行转换。具体操作参见ALLMAPS的wiki:
- GitHub:https://github.com/tanghaibao/jcvi/wiki/ALLMAPS
- Reference:Tang H, et al., "ALLMAPS: robust scaffold ordering based on multiple maps", Genome Biology, 2015
利用参考基因组进行scaffolding提高组装连续性的工具就整理到这里。虽然相似功能的工具还有很多,但是上面介绍的这几个基本上能满足绝大多数场景,比如基于多参考基因组scaffolding、设定权重、坐标/注释信息转换等,根据自己的实际需求进行选择就可以了。


