接下来,我们看看诊断试验meta分析的步骤。
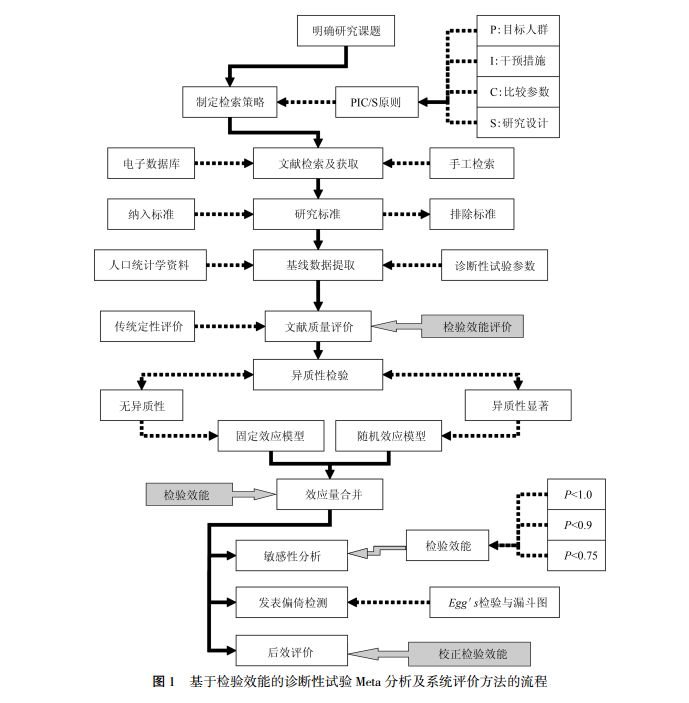
与其他类型的meta分析不同的是,诊断试验meta分析中,阈值效应是异质性的重要来源。阈值效应是因为单个诊断试验中采用不同的诊断界值引起的。当存在阈值效应时,随着灵敏度的增加,特异度逐渐减小,反之亦然。
我们常通过计算灵敏度与特异度的Spearman相关系数探讨阈值效应,二者之间若存在强负相关则提示存在阈值效应。但需要注意的是,灵敏度与特异度的相关并不一定都是由阈值效应引起。
当不存在阈值效应时,可以直接合并灵敏度、特异度等单一评价指标;当存在阈值效应时,应采用SROC曲线法,计算SROC曲线下面积和Q指数。
目前,可用来进行诊断试验meta分析的软件有RevMan、meta-DiSc、Stata、R软件等。今天,我们以R软件的meta4diag程序包为例,演示下如何做诊断试验的meta分析。
meta4diag程序包的功能是基于INLA实现的, 因此在使用时还需同时安装相关程序包,即INLA 程序包和sp程序包,程序如下:
(1)下载程序包,若运行出错,可通过R/RStudio的工具栏,手动安装。
install.packages("meta4diag")
install.packages("INLA", repos="http://www.math.ntnu.no/inla/R/testing")
(2)加载程序包。library(meta4diag);library(INLA)。
(3)读取数据。mydata<-read.csv(file.choose())。数据至少应包括studynames,真阳性例数TP、假阴性例数FN、真阴性例数TN、假阳性例数FP。
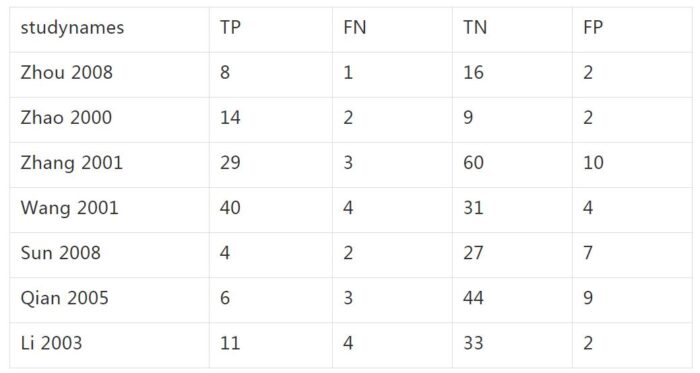
(4)整理数据。需要将录入的数据整理成函数要求的格式,这个不用担心,直接运行下列程序即可。将res<-meta4diag(mydata)中的mydata改成您自己的数据即可。
if(requireNamespace("INLA", quietly = TRUE)){
require("INLA", quietly = TRUE)
res <- meta4diag(mydata)
}
(5)探讨阈值效应。计算灵敏度(真阳性)与特异度(真阴性的)Spearman相关系数,若二者之间Spearman相关系数呈现强负相关,且p<0.05,提示存在阈值效应,则不适合合并灵敏度、特异度等,而应该拟合SROC曲线。cor.test(mydata$TP,mydata$TN,method = "spearman")。
(6)绘制森林图。可通过accuracy.type选择绘制哪个指标的森林图,如
"sens", "spec", 分别代表灵敏度、特异度。
forest(res, accuracy.type = "sens");forest(res, accuracy.type = "spec")。
(7)绘制SROC曲线,计算其曲线下面积。
SROC(res, est.type="mean");AUC(res,est.type="mean")。
诊断试验meta分析流程图
引自:刘鸿, 周洁, 冯巧灵, 顾海涛,基于检验效能的诊断性试验Meta分析及系统评价方法《转化医学杂志》2015年1期。
复制下列程序至R或RStudio软件,按要求整理数据,选择存储数据的路径即可。
#下载程序包
install.packages("meta4diag")
install.packages("INLA", repos="http://www.math.ntnu.no/inla/R/testing")
#加载程序包
library(meta4diag)
library(INLA)
#读取数据
mydata<-read.csv(file.choose())
mydata
#数据整理
if(requireNamespace("INLA", quietly = TRUE)){
require("INLA", quietly = TRUE)
res <- meta4diag(mydata)
}
#探讨阈值效应
cor.test(mydata$TP,mydata$TN,method = "spearman")
#绘制森林图
forest(res, accuracy.type = "sens")
forest(res, accuracy.type = "spec")
#拟合SROC曲线,计算其曲线下面积
SROC(res, est.type="mean")
AUC(res,est.type="mean")

