什么是批次效应?
大型的单细胞测序项目一般都会产生许多细胞,这些样本制备过程很难保持时间一致、试剂一致,另外上机测序的时候也不一定在同一个测序仪上
具体可以看这篇文章:https://www.nature.com/articles/nrg2825
Batch effects are sub-groups of measurements that have qualitatively different behaviour across conditions and are unrelated to the biological or scientific variables in a study. For example, batch effects may occur if a subset of experiments was run on Monday and another set on Tuesday, if two technicians were responsible for different subsets of the experiments or if two different lots of reagents, chips or instruments were used.
简而言之,不同时间、不同操作者、不同试剂、不同仪器导致的实验误差,反映到细胞的表达量上就是批次效应,这个很难去除但可以缩小。如果效应比较还可以接受,但是批次效应很严重,就可能会和真实的生物学差异相混淆,让结果难以捉摸。我们需要辨别到底存在多大程度的批次效应,对我们真实的生物学样本会不会产生影响。
校正批次效应的目的就是:减少batch之间的差异,尽量让多个batch的数据重新组合在一起,这样下游分析就可以只考虑生物学差异因素
目前有种方法:removeBatchEffect()(Ritchie et al. 2015),它属于limma包,假定细胞群体组成在批次中是已知或相同的 https://dash.harvard.edu/bitstream/handle/1/15034877/4402510.pdf?sequence=1
本文中使用的方法是mnnCorreect() (Haghverdi et al. 2018) https://www.nature.com/articles/nbt.4091,它基于高维表达空间中最近邻居(MNN)检测的批量修正策略,不需要预先定义或者已知全部细胞群体组成,它只需要在批次之间有关联的一小部分群体
下面将会利用三个人类胰腺scRNA-seq数据集,而且它们来自不同的组,使用不同的方法得到,因此预测会存在较大的批次效应
处理不同的数据集
第一个数据:CEL-seq, GSE81076
加载数据
数据是Grun et al. (2016) 利用CEL-seq方法,加入了UMI、ERCC,表达矩阵可以从GEO获取(https://www.ncbi.nlm.nih.gov//geo/query/acc.cgi?acc=GSE81076)
这里要学会组织R数据的方式:先新建一个文件夹,随便从其他地方复制一个
.Rpoj到这个目录,修改一下名称(最好将文件夹和这个文件都设置为英文)。双击打开.Rpoj就好,那么以后直接将数据下载到这个目录下,它就成为了我们整个项目的"根据地“
# 首先清空变量和设置默认不要因子型
rm(list = ls())
options(stringsAsFactors = F)
# 压缩文件可以直接读取
gse81076.df <- read.table("GSE81076_D2_3_7_10_17.txt.gz", sep='t',
header=TRUE, row.names=1)
> dim(gse81076.df)
[1] 20148 1728
看一下样本信息
很不幸,这个数据的列名是压缩的,包含了多种注释信息
> head(colnames(gse81076.df)) [1] "D2ex_1" "D2ex_2" "D2ex_3" "D2ex_4" "D2ex_5" "D2ex_6"
我是怎么发现的?
因为GEO描述中写道donor的编号就这么几种,因此列名的D加紧随的数字就是donor信息

工欲善其事必先利其器
需要使用sub,不理解可以看一下:https://en.wikibooks.org/wiki/R_Programming/Text_Processing#Detecting_the_presence_of_a_substring

sub包含三部分:要匹配的内容、要替换成的内容、操作的内容,两种形式:
# 第一种:不利用正则 > text <- "abc def ghk" > sub(pattern = " ", replacement = "", x = text) [1] "abcdef ghk" #可以看到,要匹配空格,替换为无(也就是去掉空格) # 第二种:利用正则匹配 sub(pattern = regexp, replacement = "\1", x = string) # 结果返回就是regexp中第一个小括号包围的内容,同理\2返回第二个小括号返回的内容
最后注意一下:
sub是替换第一个匹配到的内容,gsub是一次性全部替换匹配到的内容(即:global substitute)
好,了解了需要用的工具,先将donor ID提取出来:
donor.names <- sub("^(D[0-9]+).*", "\1", colnames(gse81076.df))
# 理解一下这个代码:
# 先看第一部分,^(D[0-9]+).* 这是利用正则表达式匹配的,主要看小括号,这里的是一会要返回的内容。意思就是说D后面是0-9数字,可以是1个或多个
# 检查一下
> table(donor.names)
donor.names
D101 D102 D10631 D17 D1713 D172444 D2 D3 D71
96 96 96 288 96 96 96 480 96
D72 D73 D74
96 96 96
同理,列名中还包括了细胞板ID,也利用sub提取
# 可以看到这个ID位于donor ID后面(当然有的样本名不包含plate ID)
plate.id <- sub("^D[0-9]+(.*)_.*", "\1", colnames(gse81076.df))
# 还是看小括号中包含的,意思是位于donor ID和下划线之前,可以是0到多个字符
# 检查一下
> table(plate.id)
plate.id
All1 All2 en1 en2 en3 en4 ex TGFB
864 96 96 96 96 96 96 192 96
看一下基因信息
同样是比较混乱的,并不是我们想要的标准名称(如Ensembl ID、gene symbol等)
> head(rownames(gse81076.df)) [1] "A1BG-AS1__chr19" "A1BG__chr19" "A1CF__chr10" "A2M-AS1__chr12" [5] "A2ML1__chr12" "A2MP1__chr12"
看起来这个比较容易替换,只要将__chr及后面的内容去掉就好(采用上面所写的sub第一种方法即可),并且注意是gsub ,全部替换的意思
gene.symb <- gsub("__chr.*$", "", rownames(gse81076.df))
# 注意要加一个结尾标志符号 $
看看有没有ERCC
目前已经有了基因名,那么看看有没有ERCC,直接grep搜索即可
需要注意的是,grep返回的结果是一个字符串(也就是匹配到的ERCC位置);另外grepl返回逻辑值,也就是全部基因中哪些是ERCC(标记TRUE),哪些不是(标记为FALSE)
is.spike <- grepl("^ERCC-", gene.symb) #结果返回大量的TRUE和FALSE
> table(is.spike)
is.spike
FALSE TRUE
20064 84
# 看到共有84个ERCC
# 以前10列为例,看看ERCC内容
View(gse81076.df[grep("^ERCC-", gene.symb),1:10])
高ERCC含量与低质量数据相关,通常是排除的标准。https://scrnaseq-course.cog.sanger.ac.uk/website/cleaning-the-expression-matrix.html也有提及:

如果ERCC的reads数很高,则表示起始内源性RNA总量低(可能发生了细胞凋亡或者其他胁迫因素导致的RNA降解;另外还可能是细胞体积小,一般来讲小细胞比大细胞有更高比例的ERCC)。
可能你会想,ERCC是内参基因吗?
其实并不是,它相对于内参基因会更稳定,看:https://cofactorgenomics.com/6-changes-thatll-make-big-difference-rna-seq-part-3/
Spike-in controls are inherently advantageous to endogenous housekeeping genes for normalization, as potential housekeeping genes such as ACTB, GAPDH, HPRT1, and B2M, etc. vary considerably under different experimental conditions
转化基因ID
library(org.Hs.eg.db)
gene.ids <- mapIds(org.Hs.eg.db, keys=gene.symb, keytype="SYMBOL", column="ENSEMBL")
> length(gene.ids)
[1] 20148
> length(gene.symb)
[1] 20148
> head(gene.symb)
[1] "A1BG-AS1" "A1BG" "A1CF" "A2M-AS1" "A2ML1" "A2MP1"
> head(gene.ids)
A1BG-AS1 A1BG A1CF A2M-AS1
"ENSG00000268895" "ENSG00000121410" "ENSG00000148584" "ENSG00000245105"
A2ML1 A2MP1
"ENSG00000166535" "ENSG00000256069"
转换完id后,记住:这里代码只是将标准的基因名转换过去,还有一些ERCC没有转换,找个例子就知道,在新的gene.ids中ERCC全部显示为NA
> grep("^ERCC-",gene.symb)
[1] 5179 5180 5181 5182 5183 5184 5185 5186 5187 5188 5189 5190 5191 5192
[15] 5193 5194 5195 5196 5197 5198 5199 5200 5201 5202 5203 5204 5205 5206
[29] 5207 5208 5209 5210 5211 5212 5213 5214 5215 5216 5217 5218 5219 5220
[43] 5221 5222 5223 5224 5225 5226 5227 5228 5229 5230 5231 5232 5233 5234
[57] 5235 5236 5237 5238 5239 5240 5241 5242 5243 5244 5245 5246 5247 5248
[71] 5249 5250 5251 5252 5253 5254 5255 5256 5257 5258 5259 5260 5261 5262
> gene.ids[5179]
ERCC-00002
NA
# 使用一句代码即可替换
gene.ids[is.spike] <- gene.symb[is.spike]
# 再检查一下
> gene.ids[5179]
ERCC-00002
"ERCC-00002"
去重复基因和没有表达量的(NA)
# 去重复和NA(这里采用反选的方法) keep <- !is.na(gene.ids) & !duplicated(gene.ids) gse81076.df <- gse81076.df[keep,] rownames(gse81076.df) <- gene.ids[keep] > summary(keep) Mode FALSE TRUE logical 2071 18077 # 结果过滤掉了2071个基因
创建单细胞对象
创建SingleCellExperiment这个对象,将count矩阵和注释信息放在一起
# 创建对象存储count和metadata
library(SingleCellExperiment)
sce.gse81076 <- SingleCellExperiment(list(counts=as.matrix(gse81076.df)),
colData=DataFrame(Donor=donor.names, Plate=plate.id),
rowData=DataFrame(Symbol=gene.symb[keep]))
# 重新设定一下ERCC位置
isSpike(sce.gse81076, "ERCC") <- grepl("^ERCC-", rownames(gse81076.df))
# 看下结果
> sce.gse81076
class: SingleCellExperiment
dim: 18077 1728
metadata(0):
assays(1): counts
rownames(18077): ENSG00000268895 ENSG00000121410 ... ENSG00000074755
ENSG00000036549
rowData names(1): Symbol
colnames(1728): D2ex_1 D2ex_2 ... D17TGFB_95 D17TGFB_96
colData names(2): Donor Plate
reducedDimNames(0):
spikeNames(1): ERCC
质控和标准化
对每个细胞进行质控,并鉴定出文库很小/表达基因少/ERCC含量高的细胞
library(scater)
sce.gse81076 <- calculateQCMetrics(sce.gse81076, compact=TRUE)
QC <- sce.gse81076$scater_qc
low.lib <- isOutlier(QC$all$log10_total_counts, type="lower", nmad=3) # 这个nmad意思就是偏离MAD计算结果几位,被认为是偏离值(用lower向左找更小的,higher向右找更大的)
low.genes <- isOutlier(QC$all$log10_total_features_by_counts, type="lower", nmad=3)
high.spike <- isOutlier(QC$feature_control_ERCC$pct_counts, type="higher", nmad=3)
data.frame(LowLib=sum(low.lib), LowNgenes=sum(low.genes),
HighSpike=sum(high.spike, na.rm=TRUE))
# 看下结果
LowLib LowNgenes HighSpike
1 55 130 388
绝对中位差(MADs)是用原数据减去中位数后得到的新数据的绝对值的中位数,可以用来估计标准差
上面返回的结果,被认为是低质量的样本数据,需要被移除
另外还有许多QC的检验标准,后续再探索
discard <- low.lib | low.genes | high.spike sce.gse81076 <- sce.gse81076[,!discard] > summary(discard) Mode FALSE TRUE logical 1292 436 # 看到总共过滤掉了400多个细胞
正式标准化之前需要先做几项工作:
- 先进行细胞聚类,利用
quickCluster(),这样做可以避免将差异很大的细胞混在一起导致误差。通过看帮助文档得知,这个函数有两种方法:hclust和igraph,基于spearman相关性分析,这种方法相对于pearson方法,是非线性的,可以针对不同量纲数据计算。我们只关心每个数值在变量内的排列顺序,如果两个变量的对应值,在各组内的排位顺序是相同或类似的,那么就认为有显著地相关性library(scran) clusters <- quickCluster(sce.gse81076, min.mean=0.1) > table(clusters) clusters 1 2 3 4 445 356 252 239
- 专用的标准化方法:之前听过的CPM/FPKM/TPM/TMM都是适用于bulk RNA-seq,分析时也经常移植到单细胞数据。但是单细胞数据有自己的特点,例如存在细胞差异和基因差异两类系统偏差,而上述方法主要考虑了基因差异。为了校正细胞间的差异,
scran包特意利用去卷积法(deconvolution) 开发了computeSumFactors函数(Lun, Bach, and Marioni 2016)。它将聚类后的多组细胞合并在一起屏蔽0值分散的问题,并采用类似CPM的方法计算标准化因子(size factor)关于scran的描述:https://www.stephaniehicks.com/2018-bioinfosummer-scrnaseq/cleaning-the-expression-matrix.html
scranpackage implements a variant on CPM specialized for single-cell data (L. Lun, Bach, and Marioni 2016). Briefly this method deals with the problem of vary large numbers of zero values per cell by pooling cells together calculating a normalization factor (similar to CPM) for the sum of each pool. Since each cell is found in many different pools, cell-specific factors can be deconvoluted from the collection of pool-specific factors using linear algebra.补充:CPM为原始reads除以一个样品总的可用reads数乘以
1,000,000,但这种方法容易受到极高表达且在不同样品中存在差异表达的基因的影响,有点"牵一发动全身"的感觉 - 代码实现
sce.gse81076 <- computeSumFactors(sce.gse81076, min.mean=0.1, clusters=clusters) # 另外对ERCC也单独计算size factor(用general.use=FALSE表示单独计算) sce.gse81076 <- computeSpikeFactors(sce.gse81076, general.use=FALSE) # 最后计算标准化的log表达量,用作下游分析 sce.gse81076 <- normalize(sce.gse81076)
鉴定高变异基因
利用函数trendVar()和decomposeVar() 根据表达量计算highly variable genes (HVGs) ,并利用spike-in的偏差为技术噪声提供参考(因为本来应该很稳定的spike-in如果方差很大,也就是波动很明显,说明实验或测序环节出了问题)
设置block是为了确保我们不感兴趣的差异(比如plate、donor之间)不会扩大真实数据偏差
block <- paste0(sce.gse81076$Plate, "_", sce.gse81076$Donor)
两行代码进行统计
fit <- trendVar(sce.gse81076, block=block, parametric=TRUE) dec <- decomposeVar(sce.gse81076, fit)
最后作图表示
plot(dec$mean, dec$total, xlab="Mean log-expression",
ylab="Variance of log-expression", pch=16)
OBis.spike <- isSpike(sce.gse81076)
points(dec$mean[is.spike], dec$total[is.spike], col="red", pch=16)
curve(fit$trend(x), col="dodgerblue", add=TRUE)
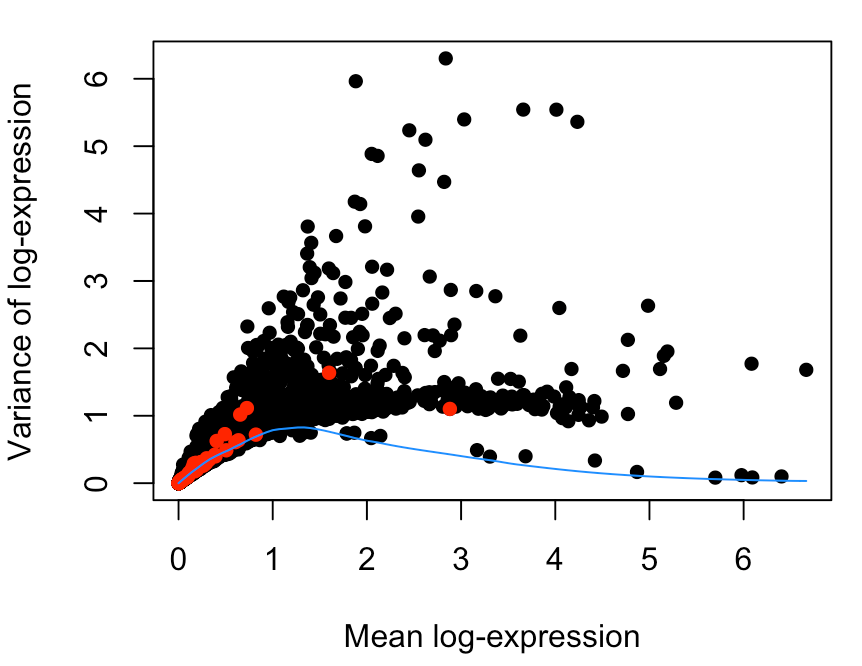
这张图不简单,我理解到的信息如下:
- 每个点表示一个基因;横坐标表示基因表达量高低(因为用log进行了缩放,所以标度变小);纵坐标表示基因表达量的波动大小;红点表示spike-in;蓝色线表示针对spike-in的波动情况做的趋势线
- 首先大部分红点在横坐标的1附近,表达量普遍处于低水平,按照之前介绍的:高ERCC含量与低质量数据相关,那么ERCC一般保持低水平就表示数据不错
- 然后大部分红点在纵坐标的1附近,表示方差(波动情况)并不大,因此可以认为技术误差在可控范围内(如果说存在很大的技术误差,那么应该会有几个红点在纵坐标较大的位置,然后趋势线也会比较陡峭)
- 在横坐标2-4这个范围内,一部分黑点(也就是一些基因)的纵坐标很大,表示它们在样本中的离散程度很大,有的样本中该基因表达量很小,有的却相当大,这就是部分要找的HVGs
挑出HVGs
在decomposeVar函数帮助文档中有一句描述:Highly variable genes (HVGs) can be identified as those with large biological components. The biological component is determined by subtracting the technical component from the total variance. (HVGs是具有高的biological components数值,而biological components取决于从总体方差中减去技术因素导致的方差)
dec.gse81076 <- dec
dec.gse81076$Symbol <- rowData(sce.gse81076)$Symbol
dec.gse81076 <- dec.gse81076[order(dec.gse81076$bio, decreasing=TRUE),]
> head(dec.gse81076,2)
DataFrame with 2 rows and 7 columns
mean total bio
<numeric> <numeric> <numeric>
ENSG00000254647 2.83712754306791 6.30184692631371 5.85904290864641
ENSG00000129965 1.88188510741958 5.96360144483475 5.5152391307155
tech p.value FDR Symbol
<numeric> <numeric> <numeric> <character>
ENSG00000254647 0.442804017667299 0 0 INS
ENSG00000129965 0.448362314119254 0 0 INS-IGF2
# 可以看到这里表达量变化最大的基因是INS,和胰岛素相关


