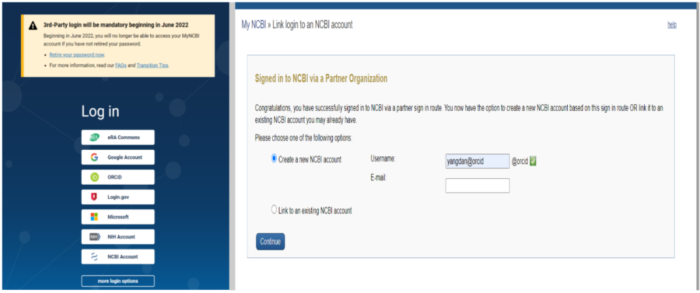
1 NCBI 账号注册

研究物种信息
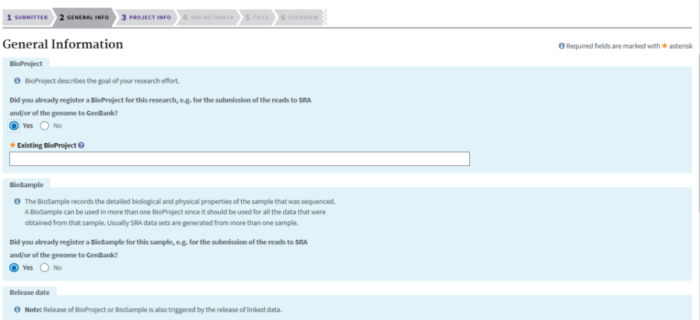
信息释放时间节点,项目的主要概况,研究目的等
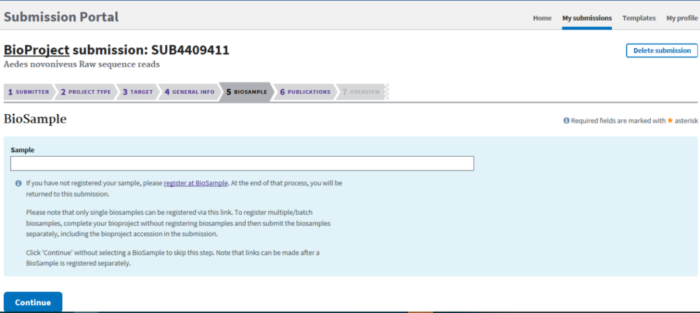
如果已经创建Biosample,则填写BioSample编号,否则创建BioSample

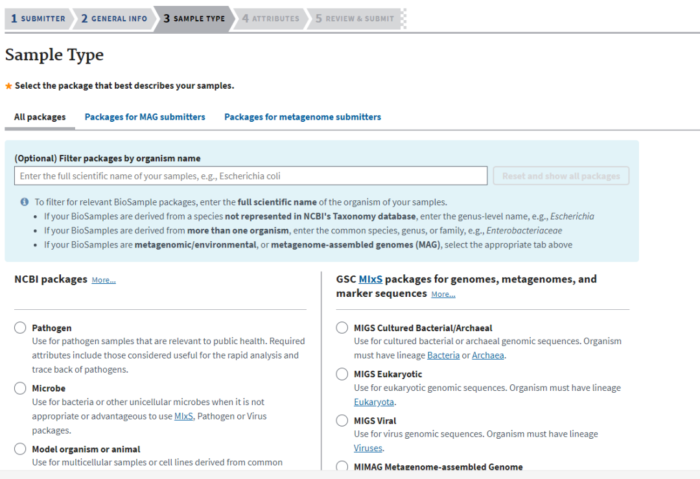
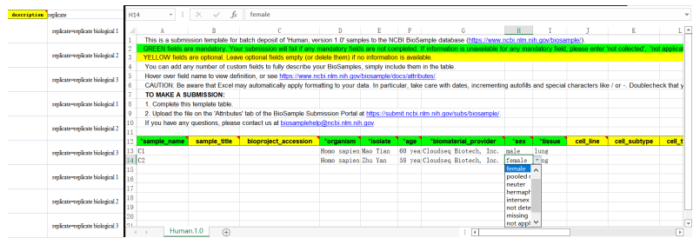
(1) 样品名称(sample name):无特定格式要求。 (2) 物种(organism):实验物种的拉丁名。 (3) 个体(isolate):为该实验样品个体取一个名字,无特定格式要求。 (4) 年龄(age):格式为【数字】 【时间单位】。 (5) 单位(biomaterial_provider):实验室或PI的名称和地址等信息,无特定格式要求。 (6) 性别(Sex):请从下拉菜单中选取符合条件的选项,而不要手动填写。 (7) 组织(tissue):样品来源的组织类型,无特定格式要求。 蓝色字段为至少一个必填项,可参考绿色字段填写方法;黄色字段则为非必填项。注意,除样本名外,需要可以有信息将每个样本区分开,可以通过添加description信息,或者另外增加replication的信息来进行区分(注意,这里信息如果填写不符合规范,上传后会报错,只需根据提示的信息对表格内容进行相应修改后重新上传即可)
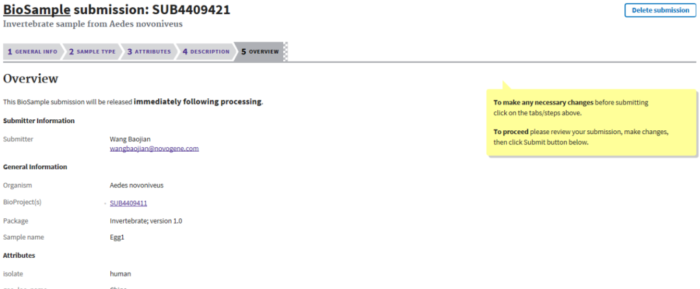
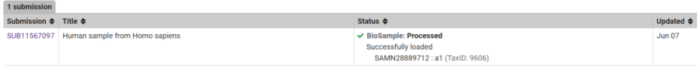
BioSample号
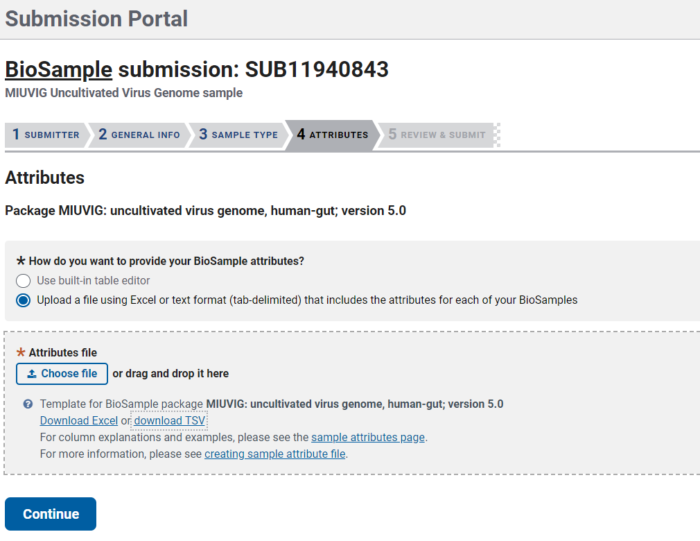
选择添加metadata 的方式,是在线填写或者下载表格填写后上传
我们建议您勾选“Upload a file using Excel or text format (tab-delimited)”并点击下图红框内的“Download Excel spreadsheet”超链接下载模板表格文件,以便在本地电脑上慢慢填写表格。
下载的“表格文件”内含三个工作表:Contact info and instructions、SRA_data、Library and Platform Terms。其中**个和第三个工作表是说明文档,实际填写 SRA 元数据的位置再第二个工作表“SRA_data”。完成填写后,回到网页点击“Choose file”按钮上传文件。SRA 官方建议您将第二个工作表另为 txt 格式再上传,然而实际使用中 Excel 格式的文件基本也能被成功读取识别。
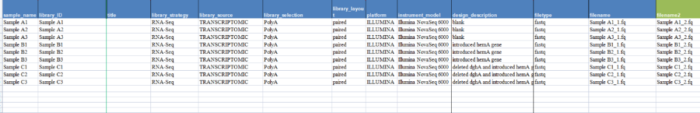
(1) 样品名称(sample name):无特定格式要求,但需与第五步中上传表格使用过的样品名称保持一致。 (2) 文库ID(library ID):无特定格式要求。 (3) 标题(title):建议采用 {methodology} of {organism}: {sample info} 这样的格式,例如“RNA-Seq of mus musculus: adult female spleen”。 (4) 文库方法(library_stratagy):请从下拉菜单中选取。点击标题超链接,您可以查看到各个选项的说明。对于转录组的测序类项目,请选择 RNA-seq。 (5) 文库来源(library_source): 请从下拉菜单中选取。点击标题超链接,您可以查看到各个选项的说明。对于转录组的测序类项目,请选择 TRANSCRIPTOMIC。 (6) 文库筛选(library_selection):请从下拉菜单中选取。点击标题超链接,您可以查看到各个选项的说明。 (6)文库端数(library_layout):请从下拉菜单选取单端测序(single)或双端测序(paired)。 (7)测序平台(platform):请从下拉菜单中选取。点击标题超链接,您可以查看到各个选项的说明。诺禾致源绝大多数的测序项目,都请选择 ILLUMINA。 (8)测序仪器(instrument_model):请从下拉菜单中选取。点击标题超链接,您可以查看到各个选项的说明。例如 Illumina NovaSeq 6000。 (9)方法描述(design_description):无特定格式要求。 (10)文件类型(filetype):请从下拉菜单中选取。绝大多数的测序项目是提供 fastq 格式文件。 (11)文件名(filename):此行所描述的文件的名称(含文件后缀名)。如果存在多个文件,例如对于双端测序而言,请您将R1测序文件的文件名填写在“filename”一列(例如“Sample A1_1.fastq.gz”),然后再将R2测序文件的文件名填写在“filename2一列(例如“Sample A1_2fastq.gz”)。 (12)参考基因组(assenbly 或 fasta_file):此行上传的数据是 bam 格式等与基因组比对后生成的文件,请提供参考基因组的信息。如果是 NCBI 上的公开基因组,请在 assembly 一列填写基因组版本名称;如果使用的是自定义的本地基因组文件,请在 fasta_file 一列填写基因组文件的名称。如果此行数据描述的文件不涉及基因组比对(alignment)操作,这两列都可以留空
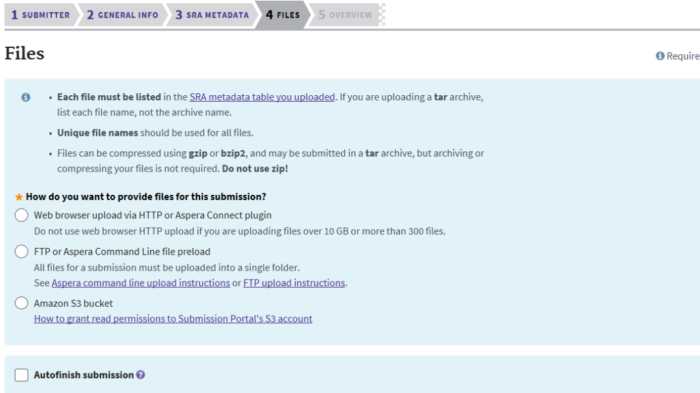
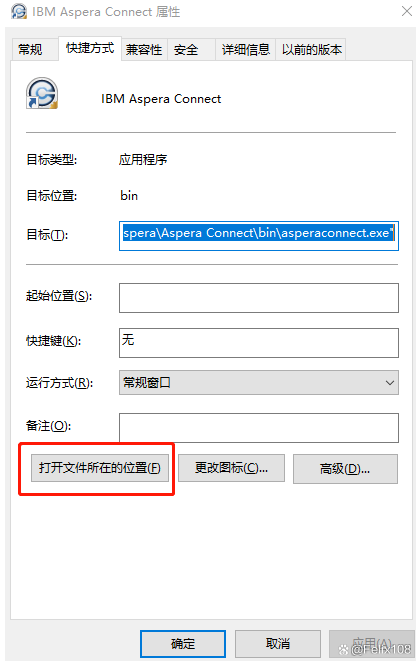
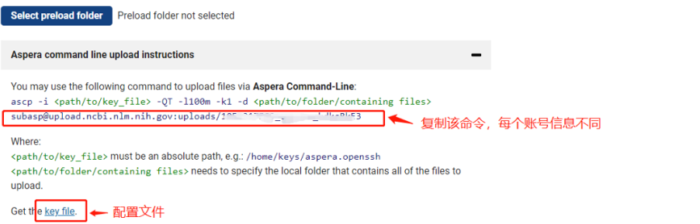
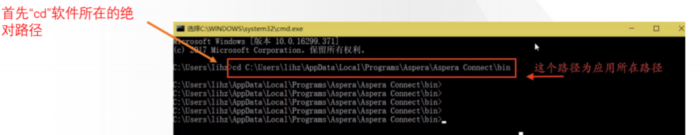
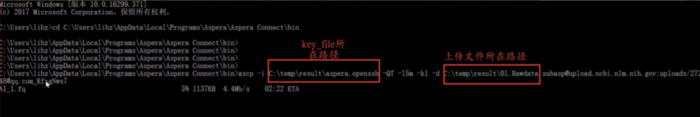
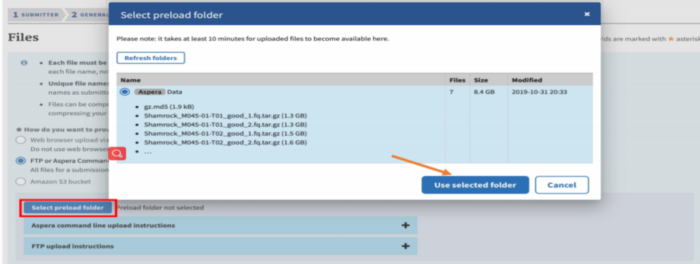
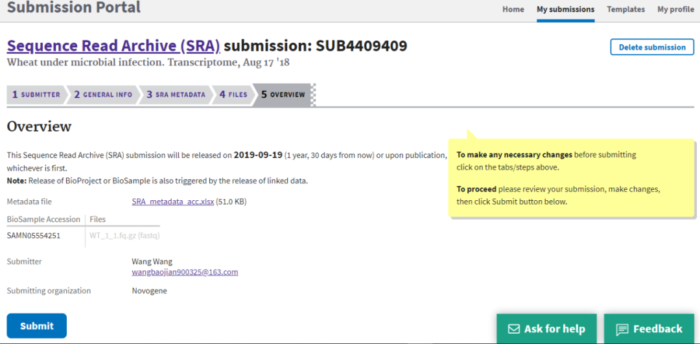
二SRA数据上传常见问题
(error:key passphrase)
以上就是本次数据上传指南的主要内容,希望本次的分享可以给大家带来帮助。


