1 定义
聚类分析(Cluster Analysis)是一类将数据所对应的研究对象进行分类的统计方法,它是将若干个个体集合,按照某种标准分成若干簇,并且希望簇内的样本尽可能地相似,而簇与簇之间要尽可能的不相似。根据分类对象的不同,可将聚类分为两大类,一类是按照变量对观测进行聚类,称为Q型聚类,即对样本(样品)聚类;另一类是根据观测对变量进行聚类,称为R型聚类,即对变量(特征)聚类。
首先说一下聚类和分类的区别,简单来说聚类是尽量把类似的样本聚在一起,在机器学习中属于无监督学习(unsupervised learning);而分类通常需要已知具体可以分为某几类,属于监督学习(supervised learning)。
2 分类
目前聚类分析常见的分类主要如图所示
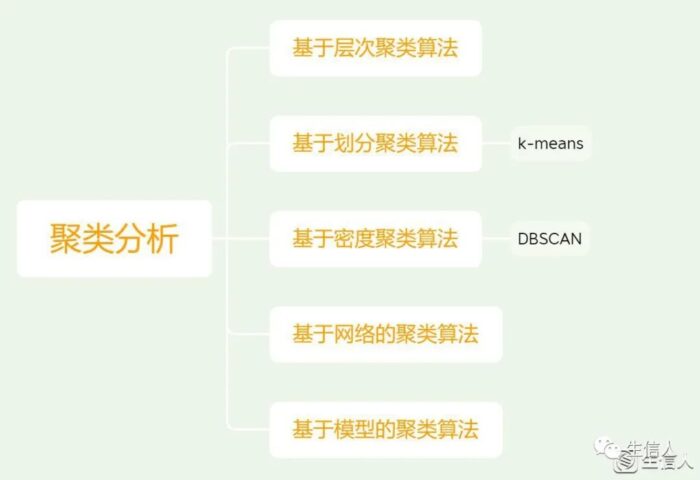
聚类分析分类
3 常见聚类分析算法
聚类分析算法很多,其中最经典的是层次聚类和k-means聚类。
3.1 层次聚类
层次聚类又称为系统聚类,是在不同层级上对样本进行聚类,并逐步形成树状的结构。在R中,hclust()函数提供了系统聚类的计算,plot()函数可画出系统聚类的树状图(也称为谱系图dendrogram),也可以使用cluster包中相关函数进行聚类结果可视化。层次聚类法的原理更简单。它的基本过程如下:
- 每一个样本点视为一个簇;
- 计算各个簇之间的距离,最近的两个簇聚合成一个新簇;
- 重复以上过程直至最后只有一簇。
3.2 k-means聚类
k-means聚类是划分方法中比较经典的聚类算法之一。它相对于系统聚类来说是一种快速聚类算法,适用于大样本的聚类分析。原理比较简单,它是基于距离,也就是欧式距离来实现的。基本过程如下:
① 首先任取k个样本点作为k个簇的初始中心;
② 对每一个样本点,计算它们与k个中心的距离,把它归入距离最小的中心所在的簇;
③ 等到所有的样本点归类完毕,重新计算k个簇的中心;
④ 重复以上过程直至样本点归入的簇不再变动。
虽然k-means聚类的原理简单收敛速度快,聚类效果较优,但缺点也比较明显,首先k值得自己定;其次它是采用迭代方法,所以得到的结果只是局部最优;再者就是对于不是凸的数据集比较难收敛;还有就是数据不均横、异常点影响大。
4 适用场景
- 使用分层聚类的情况:(1)层次聚类适合于小型数据集的聚类;(2)在不清楚数据集可以聚成几类的情况下,分层聚类可以在不同梯度水平上对数据进行探测,从而能发现类之间的层次关系。
- 使用k-means聚类的情况:当簇与簇之间区别明显时,使用k-means聚类效果会很好。因为这种聚类方法的算法比较快,所以当数据集大的时候一般也会使用到k-means聚类算法。
5 优化
5.1 K-Means
在k-means聚类中 k个初始化的质心的位置选择对最后的聚类结果和运行时间都有很大的影响,K-Means 的实质就是对K-Means随机初始化质心的方法的优化,其目的时让选择的质心尽可能的分散。距离平方进行求解,保证下一个质心到当前质心的距离最远。
5.2 二分k-means
类似于决策树的思想,通过误差平方和设置阈值,然后进行划分。聚类的误差平方和能够衡量聚类性能,该值越小表示数据点越接近于他们的质心,聚类效果就越好。
5.3 elkan K-Means
在传统的K-Means算法中,每轮迭代时,要计算所有的样本点到所有的质心的距离,这样会比较的耗时。elkan K-Means算法利用了两边之和大于等于第三步,以及两边之差小于第三边的三角形之来减少了不必要的距离计算。
5.4 Mini Batch K-Means
通常当样本量大于1万做聚类的时候就需要考虑用Mini Batch K-Means算法了,这种算法使用了Mini Batch(分批处理)的方法对数据点之间的距离进行计算。计算过程中不需要使用所有的数据样本,而是从不同类别的样本中抽取部分样本来代表各自类型进行计算,所以可以很好的减少运行时间,但准确度也会随之下降。
5.5 k-medoids
与 K-means 算法一样, K-medoids也是采用欧几里得距离来衡量某个样本点到底是属于哪个类簇。不同的地方在于中心点的选取,k-means中,选取当前簇中所有数据点的平均值为中心点,所以对异常点会非常敏感;而K-medoids中是从当前簇中选取到其他所有(当前簇中的)点的距离之和最小的点作为中心点。但K-medoids只能对小样本起作用。
6 案例
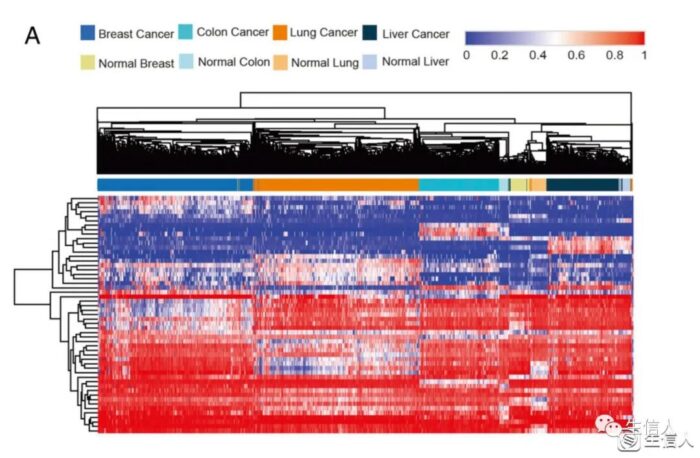
6.1 根据 CpG 位点的差异甲基化对样本进行分层聚类以区分不同的癌组织[1]。
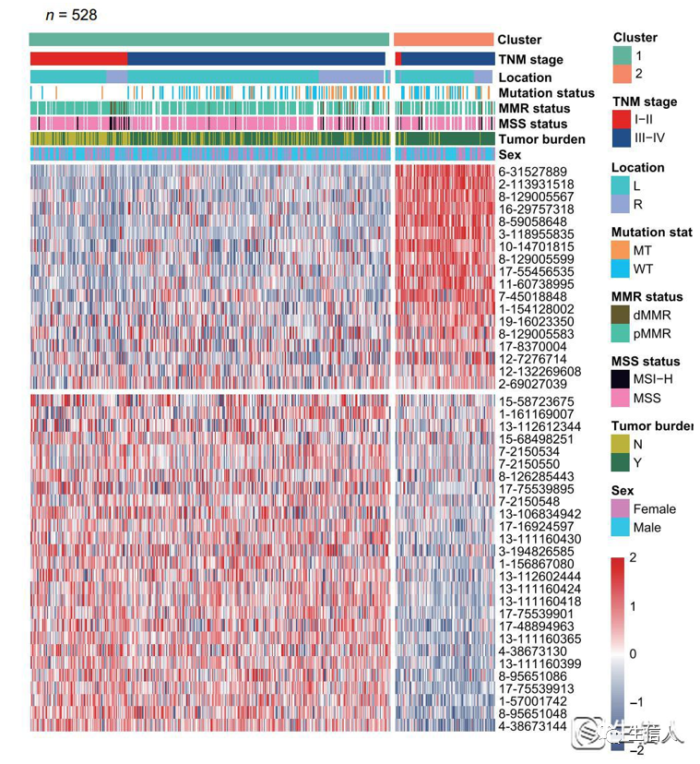
6.2 利用cfDNA甲基化标记采用迭代无监督聚类在结直肠癌中识别了两个亚型[2]。
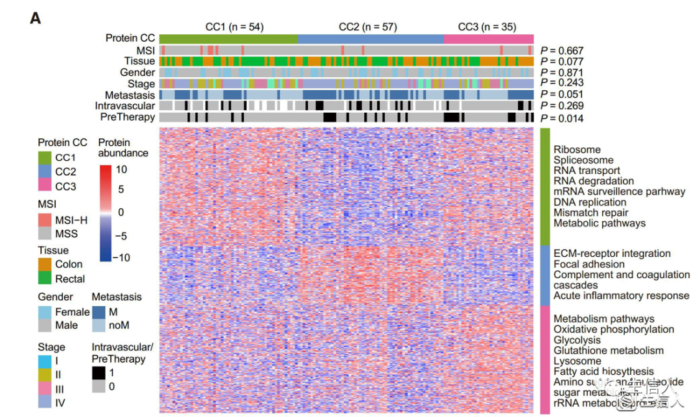
6.3 基于肿瘤和远端正常组织之间差异表达的蛋白质利用共识聚类将肿瘤样本分为三个亚型[3]。
参考文献:
- Hao X, Luo H, Krawczyk M, Wei W, Wang W, Wang J, Flagg K, Hou J, Zhang H, Yi S, Jafari M, Lin D, Chung C, Caughey BA, Li G, Dhar D, Shi W, Zheng L, Hou R, Zhu J, Zhao L, Fu X, Zhang E, Zhang C, Zhu JK, Karin M, Xu RH, Zhang K. DNA methylation markers for diagnosis and prognosis of common cancers. Proc Natl Acad Sci U S A. 2017 Jul 11;114(28):7414-7419.
- Luo H, Zhao Q, Wei W, Zheng L, Yi S, Li G, Wang W, Sheng H, Pu H, Mo H, Zuo Z, Liu Z, Li C, Xie C, Zeng Z, Li W, Hao X, Liu Y, Cao S, Liu W, Gibson S, Zhang K, Xu G, Xu RH. Circulating tumor DNA methylation profiles enable early diagnosis, prognosis prediction, and screening for colorectal cancer. Sci Transl Med. 2020 Jan 1;12(524):eaax7533.
- Li C, Sun YD, Yu GY, Cui JR, Lou Z, Zhang H, Huang Y, Bai CG, Deng LL, Liu P, Zheng K, Wang YH, Wang QQ, Li QR, Wu QQ, Liu Q, Shyr Y, Li YX, Chen LN, Wu JR, Zhang W, Zeng R. Integrated Omics of Metastatic Colorectal Cancer. Cancer Cell. 2020 Nov 9;38(5):734-747.e9.


