尽管我个人非常不喜欢人们被划分圈子,因为这样就有了歧视、偏见、排挤和矛盾,但“物以类聚,人以群分”确实是一种客观的现实——这其中就蕴含着聚类分析的思想。
前面所提到的机器学习算法主要都是分类和回归,这两类的应用场景都很清晰,就是对分类型变量或者数值型变量的预测。聚类分析是一种根据样本之间的距离或者说是相似性(亲疏性),把越相似、差异越小的样本聚成一类(簇),最后形成多个簇,使同一个簇内部的样本相似度高,不同簇之间差异性高。
有人不理解分类和聚类的差别,其实这个很简单:分类是一个已知具体有几种情况的变量,预测它到底是哪种情况;聚类则是尽量把类似的样本聚在一起,不同的样本分开。举个例子,一个人你判断他是男是女这是分类,让男人站一排女人站一排这是聚类。
聚类分析算法很多,比较经典的有k-means和层次聚类法。
k-means聚类分析算法
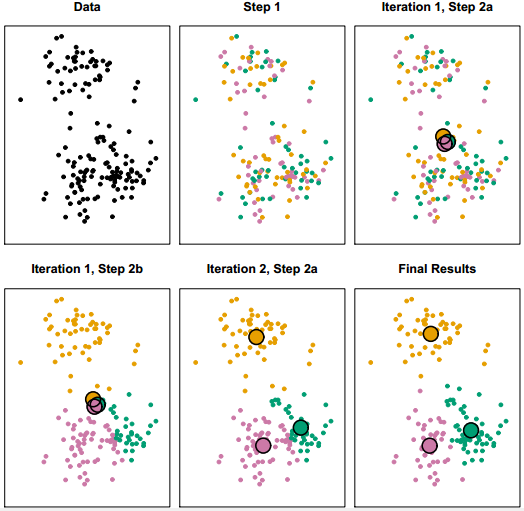
k-means的k就是最终聚集的簇数,这个要你事先自己指定。k-means在常见的机器学习算法中算是相当简单的,基本过程如下:
- 首先任取(你没看错,就是任取)k个样本点作为k个簇的初始中心;
- 对每一个样本点,计算它们与k个中心的距离,把它归入距离最小的中心所在的簇;
- 等到所有的样本点归类完毕,重新计算k个簇的中心;
- 重复以上过程直至样本点归入的簇不再变动。
k-means的聚类过程演示如下:
k-means聚类过程
k-means聚类分析的原理虽然简单,但缺点也比较明显:
- 首先聚成几类这个k值你要自己定,但在对数据一无所知的情况下你自己也不知道k应该定多少;
- 初始质心也要自己选,而这个初始质心直接决定最终的聚类效果;
- 每一次迭代都要重新计算各个点与质心的距离,然后排序,时间成本较高。
值得一提的是,计算距离的方式有很多种,不一定非得是笛卡尔距离;计算距离前要归一化。
层次聚类法
尽管k-means的原理很简单,然而层次聚类法的原理更简单。它的基本过程如下:
- 每一个样本点视为一个簇;
- 计算各个簇之间的距离,最近的两个簇聚合成一个新簇;
- 重复以上过程直至最后只有一簇。
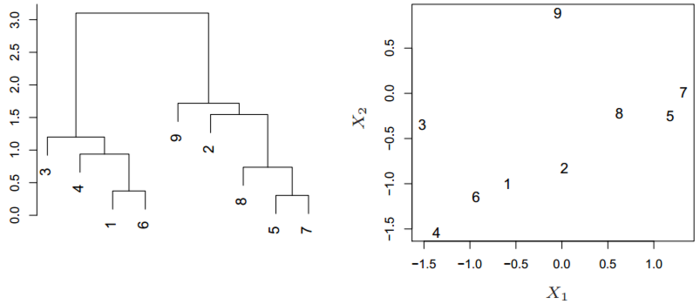
层次聚类不指定具体的簇数,而只关注簇之间的远近,最终会形成一个树形图。
层次聚类示例
通过这张树形图,无论想划分成几个簇都可以很快地划出。
以下以癌细胞细据为例,演示K-means和层次聚类法的过程。
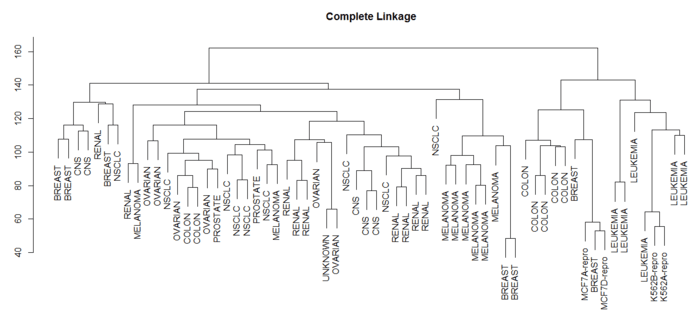
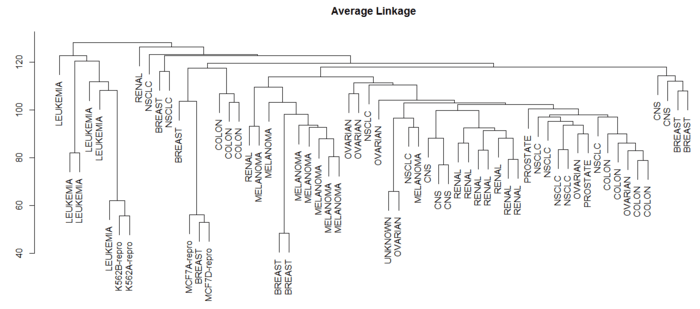
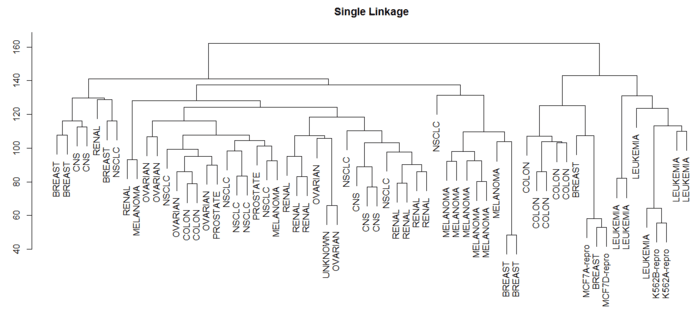
> library(ISLR) > nci.labels = NCI60$labs > nci.data = NCI60$data > > sd.data = scale(nci.data) > data.dist = dist(sd.data) > plot(hclust(data.dist),labels = nci.labels, main = "Complete Linkage", xlab = "", sub = "", ylab = "") # 默认按最长距离聚类 > plot(hclust(data.dist,method = "average"),labels = nci.labels, main = "Average Linkage", xlab = "", sub = "", ylab = "") # 类平均法 > plot(hclust(data.dist),labels = nci.labels, main = "Single Linkage", xlab = "", sub = "", ylab = "") #最短距离法
Complete Linkage
Average Linkage
Single Linkage
可见选择不同的距离指标,最终的聚类效果也不同。其中最长距离和类平均距离用得比较多,因为产生的谱系图较为均衡。
> # 指定聚类数
> hc.out = hclust(dist(sd.data))
> hc.clusters = cutree(hc.out,4)
> table(hc.clusters,nci.labels)
nci.labels
hc.clusters BREAST CNS COLON K562A-repro K562B-repro LEUKEMIA MCF7A-repro
1 2 3 2 0 0 0 0
2 3 2 0 0 0 0 0
3 0 0 0 1 1 6 0
4 2 0 5 0 0 0 1
nci.labels
hc.clusters MCF7D-repro MELANOMA NSCLC OVARIAN PROSTATE RENAL UNKNOWN
1 0 8 8 6 2 8 1
2 0 0 1 0 0 1 0
3 0 0 0 0 0 0 0
4 1 0 0 0 0 0 0
>
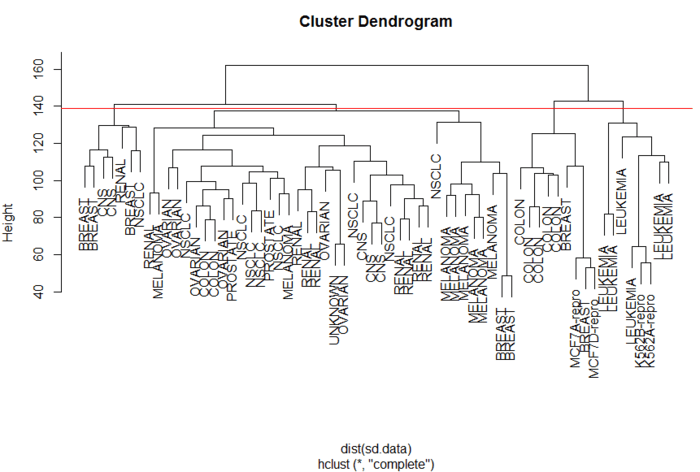
> plot(hc.out,labels = nci.labels)
> abline(h=139,col="red") # 切割成4类层次聚类划分成4类
图中一条红线将簇划分成4类,很容易看出哪些样本各属于哪一簇。
以上是层次聚类法的结果,但如果用k-means聚类的话,结果很可能就不一样了。
> # k-means聚类
> set.seed(2)
> km.out = kmeans(sd.data,4,nstart = 20)
> km.clusters = km.out$cluster
> table(km.clusters,hc.clusters) # 两种聚类结果的确有差异,k-means的第2簇与层次聚类的第3簇一致
hc.clusters
km.clusters 1 2 3 4
1 11 0 0 9
2 0 0 8 0
3 9 0 0 0
4 20 7 0 0