R语言在处理和分析数据上有其独有的优势,但也有一些让人一言难尽的智障操作,比如下面想讲的R语言中表格(矩阵)行名重复的处理。事情的起因是这样的,我最近在帮另一个研究组处理一些RNA-seq的数据,他们只提供了一张fpkm表,行名是NM_开头的Refseq数据库ID。那么很自然的操作是读入数据,将Refseq的ID转成gene symbol(毕竟画了图是要给人看的),再去画热图看下差异表达情况。然后就出现了诡异的一幕。(为了课题保密,下面用的都是自己生成的随机数)
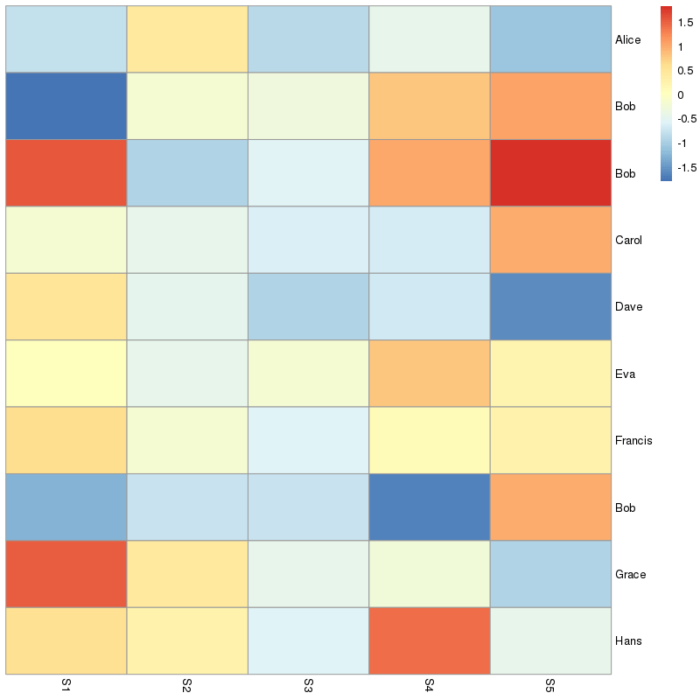
横轴是各个样本,纵轴是差异表达基因,诡异的是,竟然有三个Bob。在R语言中,行名是不能重复的,我也没修改pheatmap中相关的参数,照理来说不应该出现这种热图中重名的现象。那么很自然的,去检查一下fpkm表。
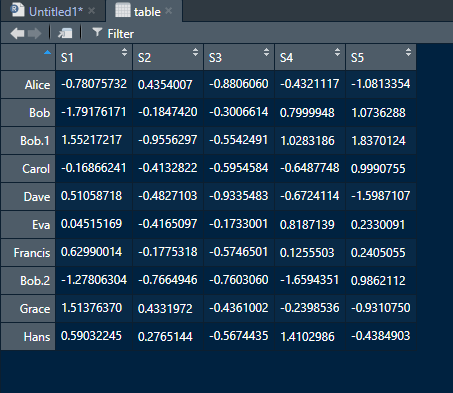
讲道理,他确实是没有重名,分别为Bob,Bob.1和Bob.2,那他为什么不在热图上也这么显示行名呢?问过学长后才知道,R语言的表格如果重名的话,他可能会报错;也可能不报错,然后自动在重名的行名后加.1,.2,.3等后缀加以区分。而在pheatmap中,它则是直接将后缀认为是重名给吃了。这就很一言难尽了,不知道该说他是智障还是智能了。来看下我上面的代码。
#生成随机数矩阵
table <- rbind(S1=rnorm(10,0,1),S2=rnorm(10,0,1),S3=rnorm(10,0,1),S4=rnorm(10,0,1),S5=rnorm(10,0,1))
table <- t(table)
rownames(table) <- c('Alice','Bob','Bob','Carol','Dave','Eva','Francis','Bob','Grace','Hans')这一步给行名赋值的操作就出现了上面加后缀的情况。
#热图
library('pheatmap')
pheatmap(table,cluster_cols = F,cluster_rows = F)pheatmap则自动把后缀给吃了。
所以让我们回到之前的RNA-seq的情况,推测事情应该是这样的。Fpkm表中的Refseq数据库ID会有多个ID指向同一个symbol(并且我发现这些同样的symbol,在fpkm表中的表达情况也一摸一样,怀疑是公司测序后给他们研究组算的fpkm表本身就有重复),然后我将ID转成symbol后,行名就出现了重复,并且被R‘机智的’识别了出来。然后我就要熬夜先去除重复然后全部重新做一遍,太难过了。总之,我们可以吸取的经验和教训有:1.R语言对表格重名有一些神奇的操作。2.拿到gene list后一定要先做一步查重(可以用duplicated函数进行操作)。
> duplicated(rownames(table)) [1] FALSE FALSE TRUE FALSE FALSE FALSE FALSE TRUE FALSE FALSE ################################################################


