今天跟大家分享的是一个在线TCGA基因表达和生存分析的工具(GEPIA2),2019年发表在NAR上,目前已更新到2.0版本,访问网址是http://gepia2.cancer-pku.cn/#index,做TCGA数据的基因差异表达、相关性、预后等相关研究的小伙伴可以作为参考,结果和图可以直接导出。
GEPIA2: an enhanced web server for large-scale expression profiling and interactive analysis.
背景介绍和意义就不多说了,直接上使用说明,能进行基因差异表达分析,生存,泛癌中表达热图绘制,降维等,网站主页如图1所示。
图1 GEPIA网站主页
1. 基因表达的一般性分析
输入框中输入感兴趣的基因名,得到在不同组织中正常和癌症中的表达热图,如图2。继续往下拉会有散点图(图3)、柱图(图4),以及共表达基因列表。
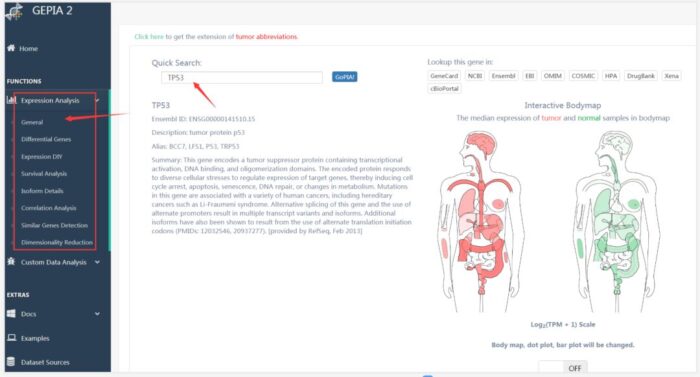
图2基因表达的一般性展示
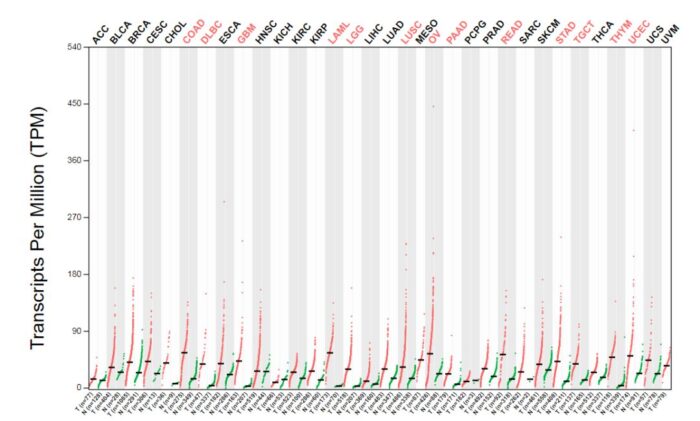
图3 基因表达散点图
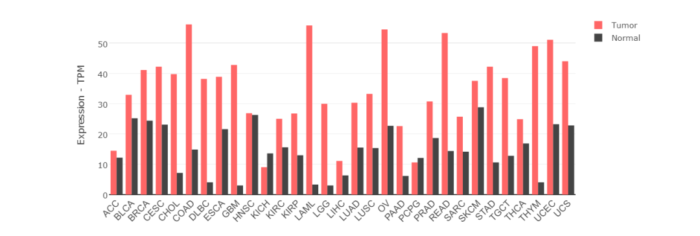
图4 基因表达柱状图
2. 差异表达分析
接下来是差异表达分析,下拉框选择癌症类型,筛选差异的阈值,筛选差异表达的方法,点击plot可得到差异基因的不同染色体分布(图5)。点击list得到差异基因列表(图6)。可以对差异结果进行下载。
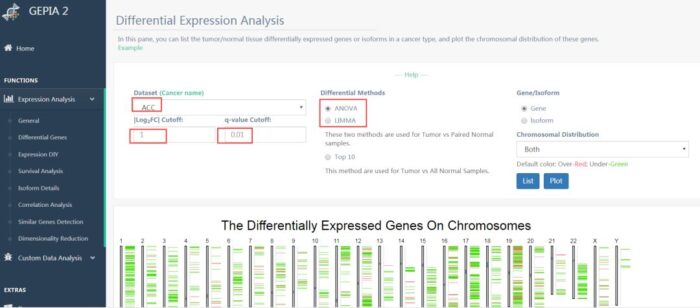
图5 差异表达基因分布
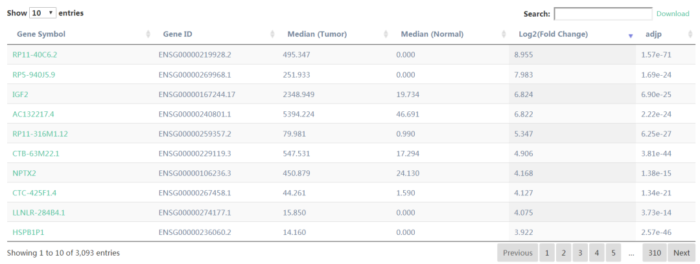
图6 差异基因列表
3. 表达DIY
这个功能比较实用,点击expression DIY,会发现有四个功能,如图7所示。例如在
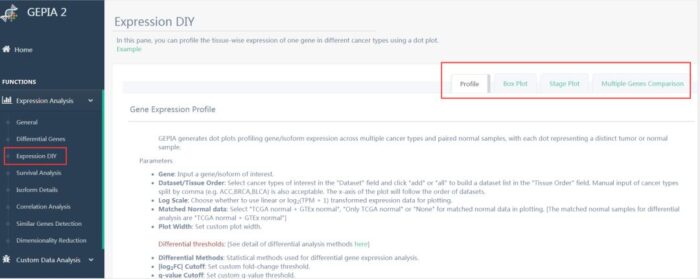
图7 expression DIY
往下拉可以输入基因名,差异的阈值和选中癌症类型,可以全选或者选择感兴趣的癌症类型,这里我们全选并点击add,点击plot,得到所有癌症与正常组织中的表达情况(图9)。
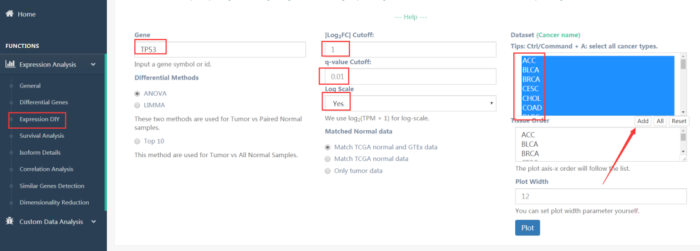
图8 表达DIY的选择
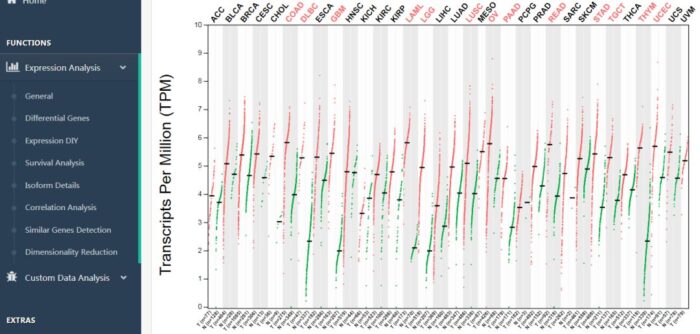
图9 癌症与正常基因谱
此外同样可以进行箱式图的绘制,选中感兴趣的癌症类型,方法类似,得到boxplot(图10)。可以下载pdf版本的图,以及DIY颜色配置。
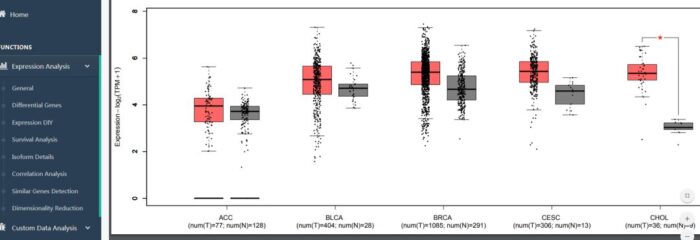
图10 差异表达箱式图
对于不同肿瘤分期,添加需要研究的肿瘤类型,同样可以对不同的分期中基因表达进行分析(图11)。
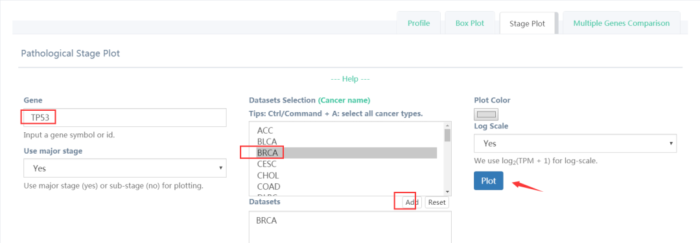
图11.Aviolinplot
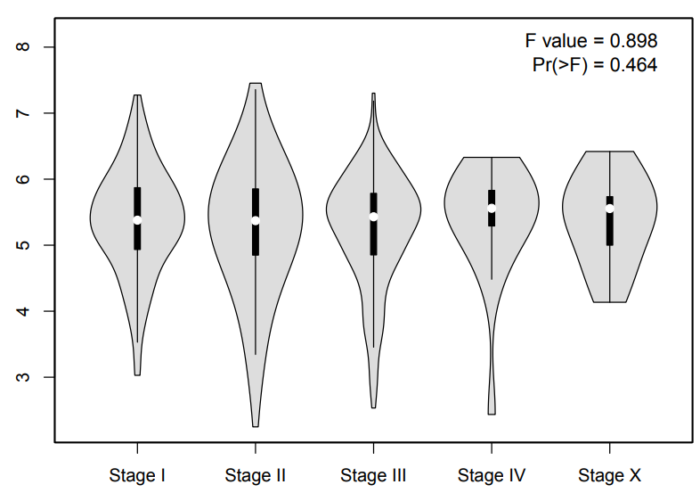
图11.B 不同分期基因表达violinplot
最后,可以对感兴趣的基因列表在多个癌症中以及正常组织绘制表达热图,输入感兴趣的基因列表,ctrl 鼠标左键选中癌症类型并点击add添加,点击plot即可出结果(图12,13)。且可以用鼠标框选后放大。
图12 多基因泛癌表达热图
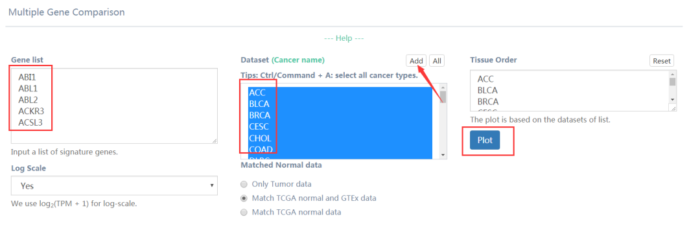
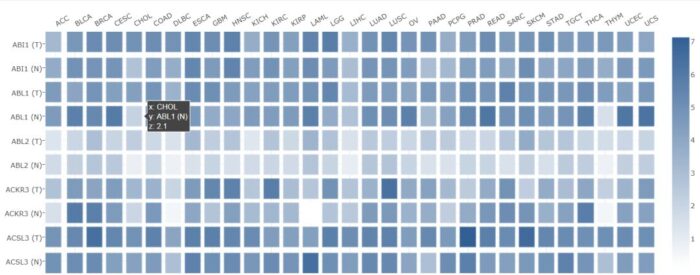
图13 多基因泛癌表达热图
4. 生存分析
我们在研究基因表达与生存之间的关联时,输入基因名以及是OS还是RFS,以及自定义颜色等,点击add添加感兴趣的癌症类型,点击plot就可以得到最终的生存分析结果(图14,15)
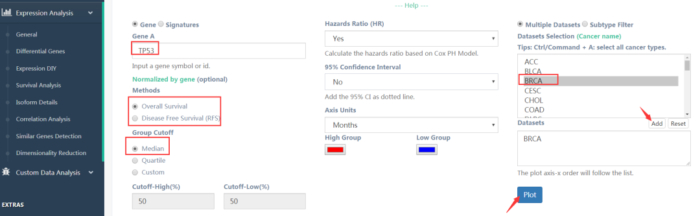
图14 生存分析
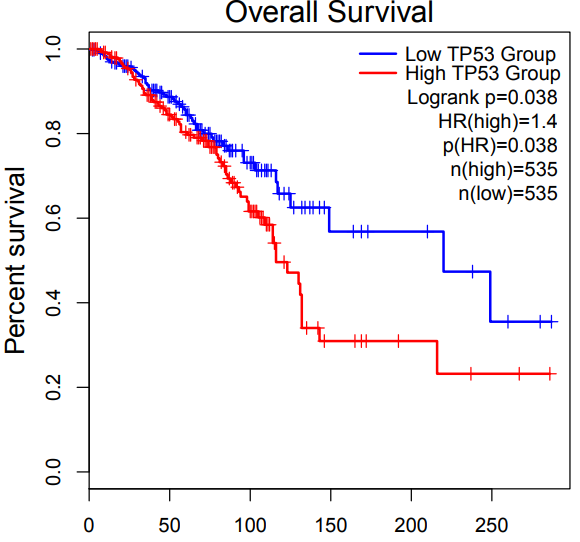
图15 生存分析结果
如果不知道癌症中哪些基因与生存有关,点击mostdifferential survival genes,得到与生存有关基因的列表,可以进行结果下载(图16)。
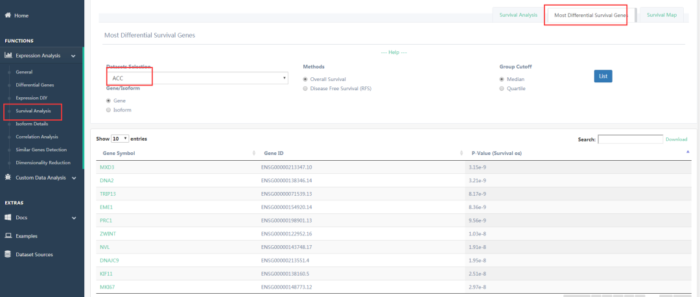
图16 生存相关基因列表
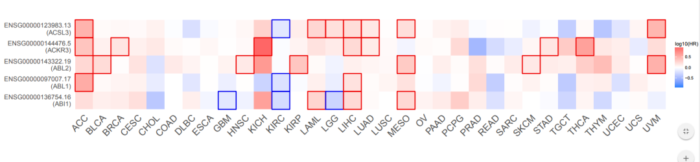
若在多个想看多个基因在多个癌症中与生存的关联,可以利用survival map工具,输入基因列表和癌症列表,得到每个癌症中每个基因与生存的显著性p值(图17,18)
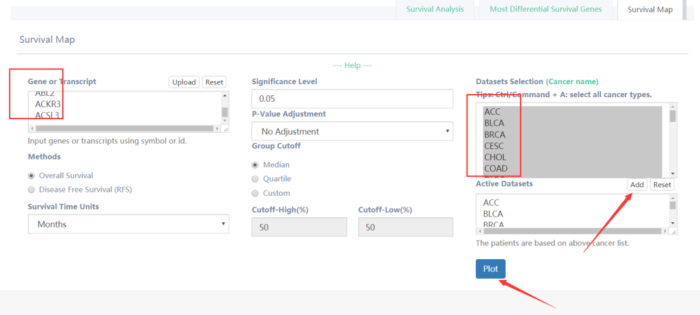
图17survival map
图18 泛癌中多基因生存p值热图
5. Isoform分析
众所周知,一个基因存在多个转录本,我们对一个基因的不同的转录本的表达同样可以研究,以观测不同转录本的表达丰度(图19)。
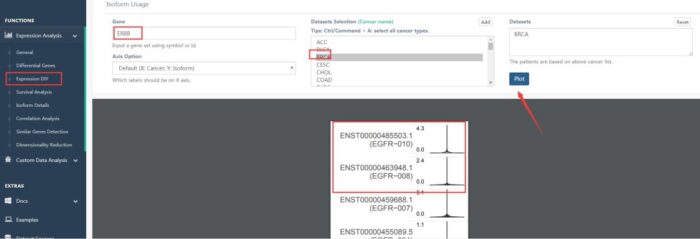
图19 不同转录本的表达
6. 相关性分析
在计算基因或signature之间相关性时,我们可以利用该工具进行在线绘图,在这里,我们对两个signature进行相关性分析,得到基因集之间相关性图(图20)。
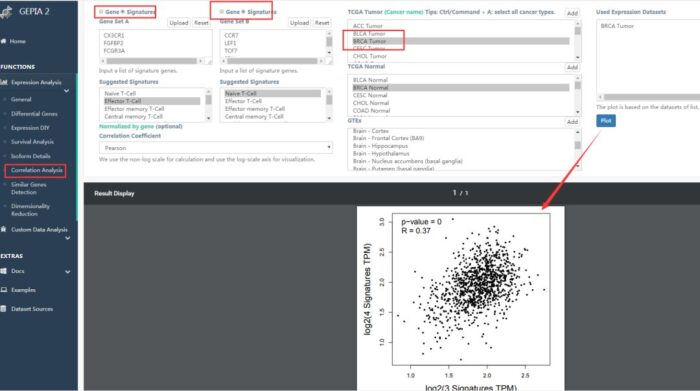
图20基因或基因集相关性分析
7. 相似基因检测
在识别相似基因时,往往采用Pearson相关性进行度量,该工具可以输出按照相似性系数排秩的基因列表(图21)。
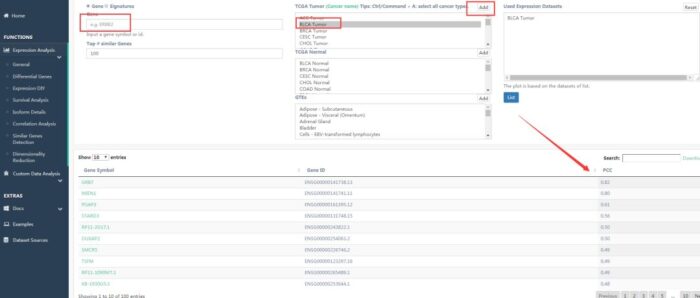
图21 相似基因列表
8. PCA降维
由于基因数目较多,维度较大,对肿瘤进行可视化比较困难。利用主成分分析(PCA)进行可视化,这里以乳腺癌的正常和肿瘤为例,选择基因进行降维(图22)。可以得到每个主成分的方差贡献图(图23)。以及样本的二维和三维可视化(图24,25)。
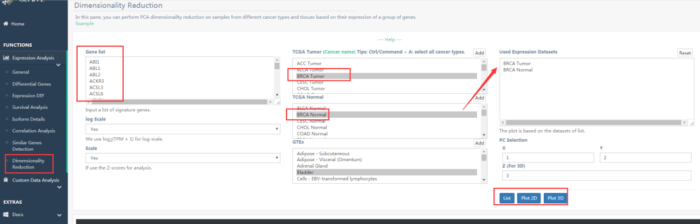
图22 PCA降维
图23 主成分的方差贡献
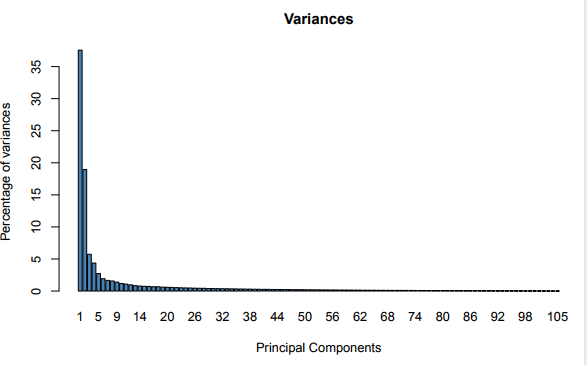
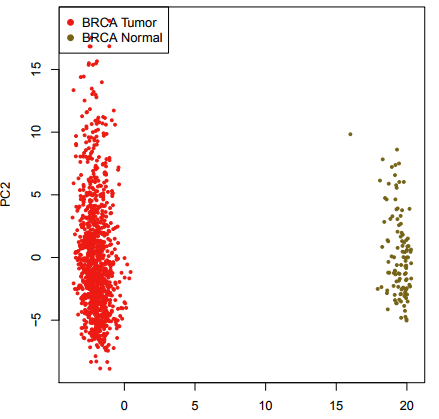
图24 二维可视化
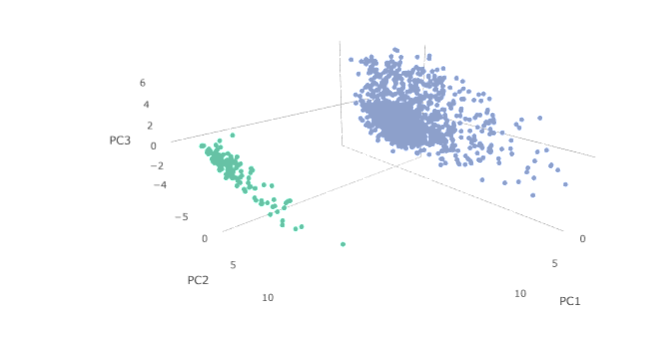
图25 三维可视化
好了今天的数据库介绍就到这里了,希望该数据库对大家的TCGA数据分析以及结果展示提供一定的帮助


