SignalP是目前应用最广泛的氨基酸序列信号肽在线预测工具。最新的版本是SignalP 4.1,预测方法基于多种人工神经网络算法,可预测细菌和真核生物氨基酸序列中的信号肽切割位点。

SignalP 4.1网址:http://www.cbs.dtu.dk/services/SignalP/。
下面就以EGFR_HUMAN 基因为例,预测其是否具有信号肽剪切位点。
首先是序列的上传,主要有两种方式(见下图),一种是点击 选择文件 直接上传FASTA文件;另一种是将氨基酸序列复制粘贴到“文本框”中。注意,每次最多可提交2,000个序列和200,000个氨基酸,每个序列不超过6000个氨基酸。
接下来是,进行简单的设置,由于是人的基因,物种这里选真核生物,D-cutoffvalues 保持默认,结果图片选包含EPS(一种常见矢量图格式)的选项。

由于仅对一个基因进行预测,结果的输出格式可选Standard和Long。在明确知道无跨膜区域后,方法选不包括跨膜区域(TM regions)预测会更精确;分析区域可自定义设置,这里保持默认的前70 个氨基酸。

点击Submit 按钮,就会得到下面的结果。下图给出了不同位点的3个分值的变化情况:
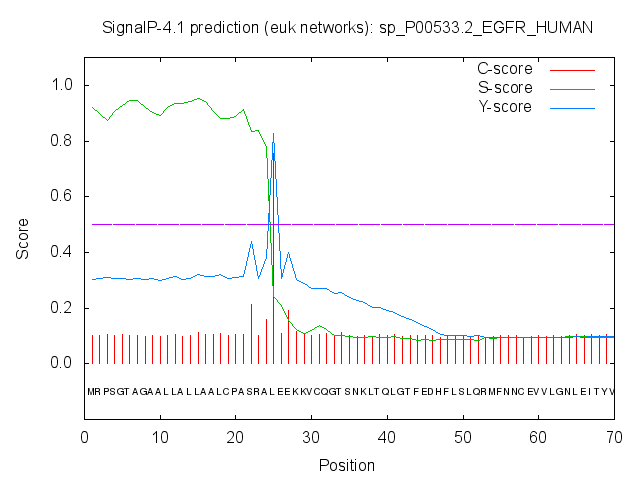
C-score (raw cleavage site score):用来区分是否为剪切位点,最高峰值为剪切位点后的第一个氨基酸(即成熟蛋白的第一个氨基酸残基);
S-score (signal peptide score):用来区分相应位置是否为信号肽区域;
Y-score (combined cleavage site score):C-score和S-score的几何平均数,用于避免多个高分C-score值对结果的影响。
此外,还有mean S和D-score(discrimination score)值(如下图):前者是从氨基酸N端到剪切位点处各氨基酸的S-score平均值,D-score值是meanS和Y-max的加权平均值。

从最终的结果可以看出,该基因具有有信号肽,且信号肽的剪切位点位于第24和25号氨基酸之间。


