Sinha, R., et al 近期在 bioRxiv上发布了一篇题为 ‘Index switching causes “spreading-of-signal” among multiplexed samples in Illumina HiSeq 4000 DNA sequencing’ 的尚未发表文章,这个题目可以简单地理解为:某个样品测序得到的序列有可能含有同一测序文库中其它样品的序列。本文作者称样品”交叉污染“的比例可达5-10%。小编看到后,想尽快让大家都知道这个信息,更新了这篇文章,Word 群体基因组下周会继续更新。
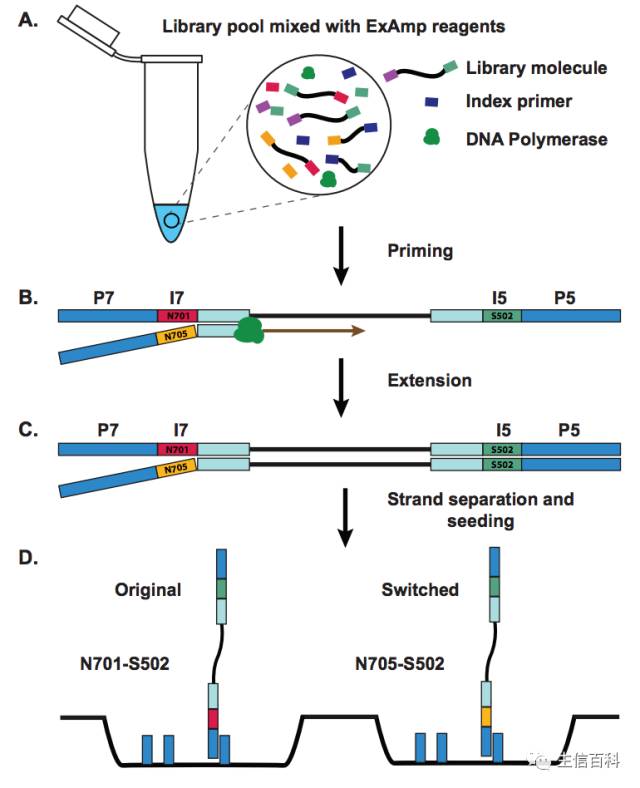
图1 ExAmp 过程中发生 Index switching 的机制 (Sinha et al. 2017)
混样测序 (多个样品混到一个文库进行测序) 时,需要使用 I7 index 和/或 I5 index 来区分样品 (但自建库时,使用位于插入片段前端或者后端的 barcode 来区分样品时,可不使用 index)。 把多个含有特定 index 的样品进行混合后,如果使用 Illumina 推荐的标准方法——AMPure XP beads 进行纯化时,得到的文库内会含有少量建库时残存的 free index primers。
这些残存的 free index primers 在 HiSeq 3000/4000/X Ten 之前的测序仪上不会出现任何问题,因为之前的测序仪在成簇前都有一个 bind-and-wash 的步骤,所有未结合到 flow cell 上的片段和 index primer 等都会被洗掉,随后才会进行扩增反应。然而,ExAmp 标准流程内已经没有 bind-and-wash 步骤,而在成簇反应时有一个必要的步骤: isothermal amplification。 因此文库经过变性后产生的单链需要先与 ExAmp 成簇反应液 (Illumina) 混合。混合后的混合液中含有的成分有: 单链的文库片段,index primer 以及 DNA 聚合酶 (图1 A)。
那么问题来了,自由的 index primer 可能会随机的加到一个已经存在的文库片段上 (图1 B),在 DNA 聚合酶的作用下扩增出一个带有不同 index 的文库片段。这样,原本属于同一样本的片段就带上了不同的 index (图1 C). 新合成链与模板分离后结合到 patterned flow cell 内纳米孔上的结合位点上(图1 D). 这些已结合的文库片段随后会扩增成簇,然后进行测序反应。这样就造成了测序得到序列的一部分被错误地分给不同的样品,也就是作者所说的 Index switching,作者还发现 Index switching 与文库中 free index prime 的含量成正比。在文中作者通过精细的实验来验证此报道的准确性。
【参考文献】
Sinha, R., et al. 2017. Index Switching Causes “Spreading-Of-Signal” Among Multiplexed Samples In Illumina HiSeq 4000 DNA Sequencing. bioRxiv. DOI: 10.1101/125724
可能产生的影响?
看到这个消息后,先不要急着砸键盘!并不是每个项目都会受到影响的。可以先想一下自己的实验目的是什么,是否使用了大量生物学重复,用什么方式建库、用什么平台测序以及后续的分析中是否能移除这种错误等。我大致总结了一下Index switching 可能产生的影响,如有遗漏,欢迎大家补充。
- 如果自建库未使用 Index, 而使用位于插入片段前端或者后端的 barcode 来识别样品的话,不受影响。
- 如果一个 lane 只测序一个样品,将不会受到任何影响。
- 混样测序时,如果在片段两端均使用了 uniquely dual-indexed adapters, 得到的结果很可能也不受影响。(可以通过双端的 Index 对数据进行过滤)
- 混样测序时,如果使用了标准的 dual-indexed adapters (一端的 index 是共享的),可能会受到影响,但受到的影响很小。
- 对于很多实验,尤其是有很多生物学重复的实验,我们在分析过程中仅使用经过严格过滤的数据进行分析,已经尽可能减少了问题序列的影响。但是对涉及使用低频率等位基因进行分析的实验仍会有影响,值得关注。
我们该如何做?
这是一篇尚未发表的文章,我们需要等待同行评议的结果,也需要确认是否有其他人可以重复此实验。不过我通过 google 搜索发现,确有其它实验室发现过类似的情况,甚至有人做 poster 介绍过。此实验结果一旦证实,Illumina 应该会给出相应的解决方案。
如果感觉自己的实验确实会受到影响,那么建议改变测序策略,比如:
- 一个 lane 仅测序一个样品
- 使用其它的方式来纯化文库以尽可能地去除多余的 free index primers
- 混样测序时不同样品间不要共享 Index (每个库能混的样品数大大减少)
- 等待厂商提供新的试剂盒 (unique-at-both-ends adapter sets)
【延伸阅读】
Illumina 测序:
- https://www.illumina.com/documents/products/techspotlights/techspotlight_sequencing.pdf
- https://support.illumina.com/content/dam/illumina-support/courses/examp-cluster-workflow/story.html?iframe
Index Switching 的相关报道:
- http://enseqlopedia.com/2017/04/index-swapping-illumina-examp-clustering/
- http://enseqlopedia.com/2016/12/index-mis-assignment-between-samples-on-hiseq-4000-and-x-ten/
- http://www.molecularecologist.com/2017/04/right-reads-wrong-index-concerns-with-data-from-illuminas-hiseq-4000/
- https://www.youtube.com/watch?v=ZR_xQFCxGWA