前面已经分享了一篇文章介绍《使用DnaSP计算核苷酸多样性和单倍型多样性》,最近刚好在用DnaSP v5,在写一篇文章与大家分享一下利用DnaSP 计算Ka、Ks,即dN,dS。
第一步:打开主界面之后,在File里面读取比对好的基因核苷酸序列(DnaSP支持fatsa,phylip等多种格式)
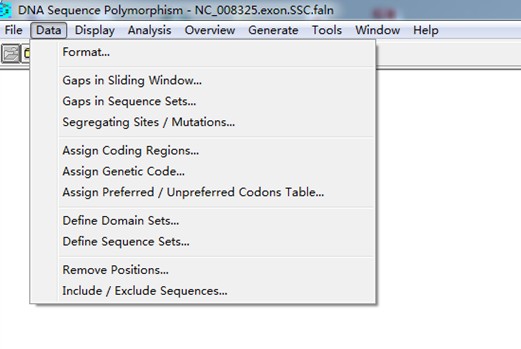
第二步:设置编码区域与密码子。
在data这一栏里面,有一个Assign Coding Regions的选项,在这里设定一下编码区域的其实位置,如果提交的序列全是DNA序列,可以设置为从序列的起始到终止。点击后提示是否需要设置其他编码区域,可以选择否。
另外还需要设置一下使用的密码子表,这一项需要在Assign Genetic Code这一栏来选择,这里面有目前所有的密码子表。
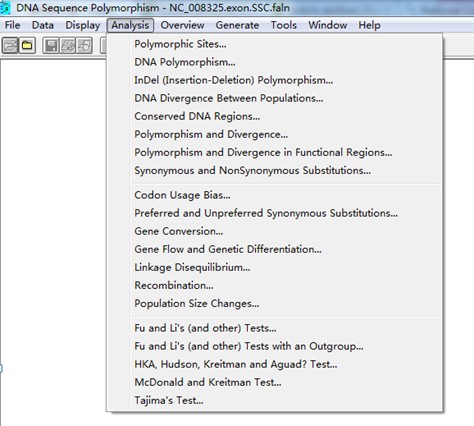
第三步:在Analysis里面有一个选项是Synonymous and NonSynomymous Substitutions,点击这项进行Ka,Ks(即dN,dS)分析。
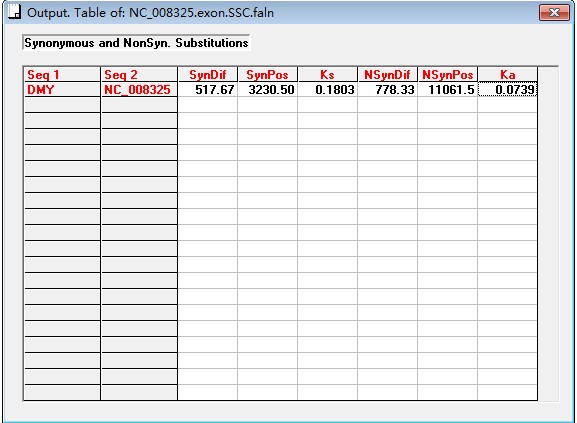
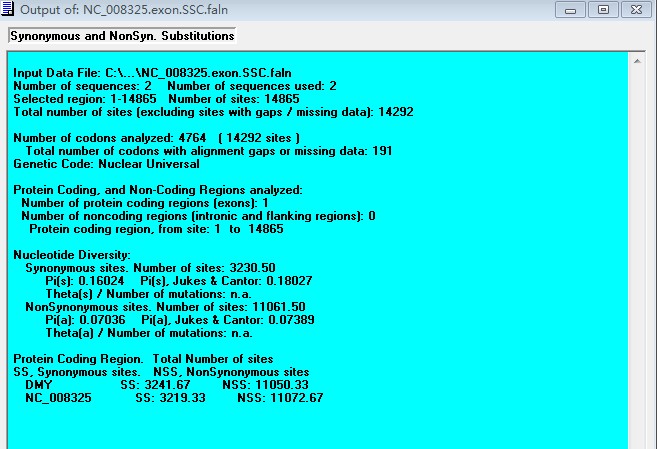
第四步:执行完第三步后,会出现两个结果页面,其一是pair-wise的基因Ka,Ks值得列表,例如:
另一个是以文本的形式展现的,额外还有其他的信息。



1F
您好,这篇文章,对我帮助很大,谢谢您,不过还有一点不太明白,就是那个密码子表如何设置,怎么选,根据啥选?能否在我邮箱里回复一下,谢谢
2F
您好!计算出来Ks或ka的值时na,是说明这两个序列同源性太低了么?http://