bismark 软件根据序列的比对情况就可以识别甲基化位点,首先需要对基因组建立索引,建好索引之后,就可以开始比对了。
我用的软件自带的单端测序的数据集进行测试, 命令如下
bismark hg19_bismark_db/ test_data.fastq -o test
第一个参数为bismark_genome_preparation命令构建的基因组索引所在的目录,第二个参数为需要比对的序列, -o参数指定输出的目录
bismark的参数很多,通常情况下,采用默认参数就好。其他参数全部默认的情况下:bismark 比对的过程分为以下几步:
1 . 将输入序列进行C->T的转换
软件在运行过程中的log信息如下:
Input file is in FastQ format Writing a C -> T converted version of the input file test_data.fastq to test_data.fastq_C_to_T.fastq Created C -> T converted version of the FastQ file test_data.fastq (10000 sequences in total) Input file is test_data.fastq_C_to_T.fastq (FastQ)
2 . 将C-> 转换好的序列分别与 C->T 转换的基因组和G->A 转换的基因组进行比对
Now starting the Bowtie 2 aligner for CTreadCTgenome Using Bowtie 2 index: hg19_bismark_db/CT_conversion/BS_CT Using Bowtie 2 index: hg19_bismark_db/GA_conversion/BS_GA
我用的是 1.9.0 版本的bismark, 现在绝大多数的BS-seq的文库构建都是采用illumina提供的的标准protocol, 构建出来的文库都是链特异性的文库,所以从0.7.0版本之后的bismark, 默认只做两次比对,但是这个默认情况只适合链特异性文库,如果你的文库不是链特异性的,那么就需要添加--non_directional选项。
何为链特异性文库,就是说链是由方向性的。对于普通的文库,测序的插入片段都是双链,但是链特异性文库是单链。通过在反向互补时添加特定的标记,在双链合成后,将第二条链去除,最后用于测序的就只有一条链了。如果一个甲基化位点发生在基因组的正链上,那么这段区域在测序时插入序列就只有正链上的序列,如果发生在负链上,则只有负链作为插入序列。
在bismark中,将基因组的正链定义为top strand , 简称OT, 负链定义为bottom strand, 简称OB; 亚硫酸氢盐处理后,正负链之间并不是完全的反向互补的,将OT链的反向互补链定义为CTOT, 将OB链的反向互补链定义为CTOB。
对于链特异性文库而言,由于插入序列为单链,只需要比对OT和OB两条链即可,大大减少了运算量,所以目前illumina的标准BS-seq protocol构建的文库都是链特异性文库,新版的bismark默认的运行方式也是针对链特异性文库的。
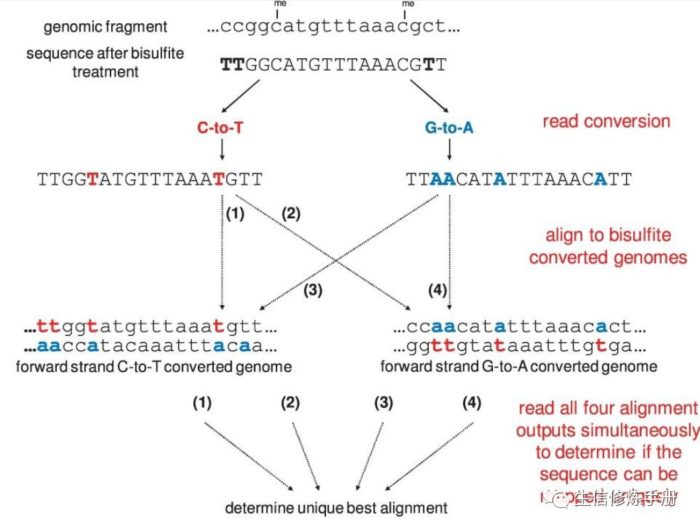
图中展示了bismark比对的过程, 包括了原始序列转换和比对两个过程:
原始序列转换包括两种方式:
- C->T 的转换
- G->A 的转换
比对也包括两种基因组:
- C->T 转换的基因组
- G->A 转换的基因组
所以每条reads 最多会有 2 X 2 = 4 种比对情况,对于链特异性的文库,只有C->T 转换,所以只有2种比对情况。