刘国庆 白音宝力高 邢永强
人类基因组中仅仅约 2%的 DNA 是用来编码蛋白质的,而剩余 98%的序列则一度 被认为是近中 性 进 化 过程中 产 生的 没有 功 能的 “垃圾 DNA (Junk DNA)”. 随着研究的深入,人们觉察到所谓的“垃圾 DNA”其实是人们尚未认识的宝藏[1]. 假 基因是最典型的“垃圾 DNA”. 假基因是功能基因的缺陷拷贝,它在序列结构上与功能基因非常相似,但已丧失了正常的蛋白质编码功能. 由于假基 因与功能基因的紧密相关性以及它在基因组进化过程中的重要性,在过去 10 年,假基因一直是“非 编码 DNA”领域的研究热点之一. 假基因在脊椎动物基因组中比较常见,尤其在哺乳动物基因组中比较“泛滥”. 据估计,人类基因组中有 2 万个左 右假基因及其片段.
假基因的重要性体现在以下几方面.a. 假基 因为基因组动态学研究(genome dynamics)和进化研究提供了非常宝贵的材料. 在长期的进化过程中,不同的生物如何适应选择压力获得生 存竞争的优 势,是基因组进化研究探讨的主要问题. 假基因的演化过程对于此类问题的研究非常重要,因为假基因精确地记录了基因组 DNA 在进化选择压力下怎 样改变的“痕迹”,保留了数百万年前祖先功能基因的分子记录. 从这种意义上来讲,假基因是基因 组“化石”. 通过同源假基因的比较 基因组学分析,可以绘制基因组突变谱(碱基置换,插入 / 缺 失突变),也可以判断假基因在各物种中出现的时间顺序及进化方向.有些假基因是有功能的,但多数假基因可能没有功能. 没有功能的假基因是基因 组中理想的中性进化标签,因此可通过假基因一级序列的比对分析获得分子的中性进化速率时标,并将其用于分子进化模型中的参数标准化. 假基因序 列中包含了很多信息,包括假基因产生的驱动力、机制及特点均可在假基因的序列结构特征中找到线索. 从假基因的一级序列变化(如非同义、同义突 变率的比值 Ka/Ks)中能够判断出假基因所受的选择 压力.如果说假基因的一级序列是从分子水平上记 录选择压力的“精细”标签,那么,假基因在基因组中的丰度和分布则是从“宏观”层面上反映与群 体动态(population dynamics)相联系的众多进化选择 压力的重要参数. 例如,由假基因的年代和数量分 析 , 可 获 得 DNA 重 复 (duplication) 和 返 座 (retrotransposition) 事件 (产 生假基因的 两 种主 要 途 径)的活跃度在进化时间轴上的分布情况. b. 研究 表明有些假基因是有功能的. 功能主要包含以下两 方面的内容:(1)有些假基因对基因的表达调控发 挥着重要作用. 层出不穷的假基因转录的证据表明 假基因可能在 RNA 水平上参与基因的表达调控.部分假基因在进化过程中的高度保守性也启示这些假基因可能具有人们尚未知晓的功能. (2)曾“死去”的假基因有时可重获新生,对新基因的产生及功能扩展有所贡献. c. 由于与功能基因之间的相 似性,假基因对基因注释和遗传疾病的诊断与治疗带来了很多麻烦.因此,全基因组范围的假基因识别对于基因的正确注释以及在临床医学上也是非常 重要的.
在过去几年时间里,有关假基因的研究取得了很大的进展,主要集中在假基因识别、假基因在染 色体上的分布及其分子进化规律,以及假基因功能揭示等几个方面.本文围绕这几方面对假基因的研 究现状进行较全面的概述.
1 假基因的来源
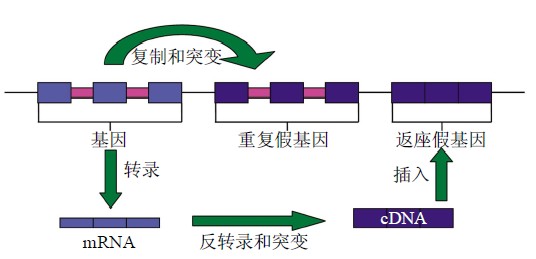
假基因是功能基因的缺陷拷贝,它源于蛋白质 编码基因、与起源基因非常相似,但是不能编码蛋 白质. 假基因的形成,即基因正常活性的丧失是由 对基因表达有阻断作用的突变导致的. 这些变化主 要包括消除起始转录的信号,阻止外显子 / 内含子 连接点的剪接或过早地终止翻译等. 产生假基因的 渠道主要有以下 两种[2-3] (图 1):一是 基因组 DNA 重复或染色体不均等交换过程中基因编码区或调控 区发生突变(如碱基置换、插入或缺失),导致复制 后的基因丧失正常功能而成为假基因,这种假基因 称 为重 复 假基因 (duplicated pseudogene); 二 是 mRNA 转录本反转录成 cDNA 后重新 整合到基因 组,由于插入位点不合适或序列发生突变而失去正 常功能,这样形成的假基因称为加工假基因或返座 假基因(processed pseudogene or retropseudogene)
Fig. 1 Pseudogene origin
图 1 假基因来源
重复假基因具有与功能基因非常相似的结构, 在相应的位置上还有相当于外显子和内含子的序列,而且倾向于出现在其祖先基因的侧翼. 这些特征均归因于重复假基因的产生机制—DNA 水平上的片段重复(segmental duplication). 值得一提的 是片段重 复是 新基因( 即重 复基因) 产生的主 要 途径.在这种重复过程中基因的启动子很可能被一起复制,因此重复假基因的上游可能有调控序列,这一 点 与 加 工假基因 不同 . 由于 加 工 假 基因是 mRNA(没有内含子,很少有启动子)反转录后随机 插入到基因组形成的,所以加工假基因一般没有启 动子,更没有内含子. 加工假基因以序列中终止密码子的提前出现 (premature stop codon)、移码突变(frameshift)和没有启动子为主要的缺陷特征[2-3]. 有证据表明加工假基因是在活性长散置元 LINE-1(L1)编码的逆转录酶的辅助下进行转座的非自主性反转录转座子[4-5]. 由于返座机制 ,年轻加工假基因两侧一般存在正向重复序列,3忆端有 poly (A)尾[2-3]. 对于无功能的假基因来说,随着序列的中性进化[6], 这些特征将逐渐消失,随之出现的是在随机突变的累积过程中产生的移码突变、终止密码子的提前出现和散置重复序列的插入[7-8]. 真核基因组中假基 因通过 DNA 重复和返座两种方式产生,而原核基因组却没有 L1 元件,所以在原核基因组中还没有 以返座的方式产生假基因的报道. 研究表明,基因的横向转移、DNA 水平上的重复以及单拷贝基因 的退化是原核基因组中产生假基因的主要途径[9].
核基因组中还发现从线粒体 DNA 转移过来的假基因[10],这些假基因的祖先基因是线粒体 DNA中的基因,这使得基因注释等工作更加复杂化. 由 于线粒体和核基因组基因的蛋白质编码方式(即遗传密码)有所不同,有些线粒体基因转移到核基因组后便丧失蛋白质编码功能,从而成为假基因. 线粒体基因向核基因组转移时 有两种可能的转移方式:DNA 水平上的转移[11]和 RNA- 介导的转移[12],实验数据倾向于支持 DNA 水平上转移的假说[13].研究 发现, 假 基因 存 在 于 大 肠 杆菌 、 酵母 、 拟南芥、水稻、线虫、果蝇、小鼠、人等多种基因组 中[7-8,14-21],在哺乳动物基因组中分布较多,尤其在 人类基因组中存在上万条假基因 [7-8],其中多数为 加工假基因[21].
2 假基因研究概况
2.1 假基因的识别
假基因的鉴别是假基因相关研究的基础,而且它对基因注释的精确度也有很大影响. 尤其是基因与重复假基因之间的区分是很有挑战性的问题. 假基因的识别工作主要是由耶鲁大学 Gerstein 实验室研究 小 组 完 成 的 [7,10,16-19]. 他 们 从 果 蝇 、 线 虫 、 小 鼠、人等很多物种基因组中系统地搜 索识别假基因, 并创 建了 专门 的假基 因数 据 库 [21] (http://www.pseudogene.org), 可供 研究 人员 免费 下 载 使 用 .表 1 中列举了目前国际上几个通用的 假基因数据库[21-23]和假基因识别程序[24-26].
表 1假基因数据库和假基因识别程序
Table 1 Pseudogene databases and pseudogene identification programs
在全基因组范围内识别人类假基因的代表性工作 还 包 括 Torrent( 称 为 Bork 假基因数 据 集 ) 和Khelifi(称为 Hoppsigen 假基因数据集)等的工作[8, 23].这些工作采取的策略基本相同,那就是基于与已知功能基因的序列相似性搜索方法识别假基因,其中 查询 序列 (query sequences) 可 以 是 基 因的 DNA 序 列,也可以是对应的蛋白质序列. 当然,相似性搜索方法中参数标准(如 E 值)和外加条件(如序列结 构特征或演化特征信息)以及所使用的基准基因库的不同都会造成所识别出的假基因在数量和结构上有较大差别. 例如,Gerstein 小组识别出的人类加工假基因大约有 8 000 条[7],而 Bork 中人类加工假 基因却多达 17 000 余条[8],Hoppsigen 中人类加工假 基因 却只 有 5 000 条左右 [23]. Hoppsigen 假基因 库是人和小鼠基因组的加工假基因数 据库,其中不 含 重 复 假 基因 . 由于 使 用 的 方 法 比 较 严格 [23],Hoppsigen 库中的假基因多数是可信的,但数量上最少. 相比之下,Bork 的假基因识别程序是最不严格 的,它对 序列 相似 性( 或保 守性 )的要求 并 不高,导致识别出的假基因在数量上最多,但可信度 却值得怀疑. Bork 的假基因识别中还使用同义 / 非 同义突变率的比值作为“无功能”的判别标准,这一点与其他组的做法不同. 不同数据集间的比较结果显示,50%(以小样本数据集为标准)以上的加工 假基因是不同数据集间所共有的[23].
Gerstein 小组[27]于 2006 年改进了其假基因识别程序,主要是对查询序列进行了细化. 先前的程序 是以完整的 CDS 序列或蛋白质一级序列作为查询序列后用 Blast 软件在基因组上扫描,筛选出相似序列作为假基因候选序列. 若候选序列中有较长的插入片段( > 60 bp),则这个插入片段就被认为是内含子,从而这条序列被归类 为重复假基因. 事实上,这条序列有可能是转座子插入其中的加工假基 因. 为克服这一缺陷,Gerstein 小组 Zheng 等[27]改用以左右两侧各延长 50 bp 的延长外显子作为查询序列后在基因组序列上扫描. 这一程序的优点在于 以基因的内含子—外显子结构作为基因重复的标记,从而能够精确地鉴别出重复假基因. 用这种方法,他们从 ENCODE 计划的 44 个序列片段中共找出了 164 个假基因,其中 16 个为重复假基因. 不 同的假基因识别程序识别出来的假基因数量和结构上都有很大差异 .Zheng 等[28] 于 2007 年 又发 展了 一种融合各种假基因识别方法为主要优点的假基因注释方法,并应用于 ENCODE 区域的假基因注释, 发现了 201 个假基因,其中 2/3 的假基因是加工假基因,约 80%的加工假基因是灵长类特异假基因,表明灵长类细胞中返座活性的持续增强趋势. 保守性和进化分析发现, ENCODE 区域的假基因多数经历 着中 性进化. 转录 活性分析 发现,约 1/5 的ENCODE 区域假基因是转录的.
由 非 长 末 端 重 复返 座机 制 (non-long terminal repeat retrotransposition)产生的返座序列有两种显著的特征:序列末端有 poly (A)尾巴,序列两侧有相同的靶位点重复片段(TSD). 返座假基因就是由这 种机制所产生[4-5]. 基于这种特征,Terai 等[29] 编写了鉴别返座假基因的程序 TSDscan,扫描人类基因 组后发现了 654 个长度较短( < 300 bp)的假基因.分析这些假基因及其对应的 mRNA 的长度后发现,与短 mRNA 相比,较长的 mRNA 能够产生更短的假基因. 为解释这种现象,他们提出假设:多数长mRNA 在反转录之前被切断,切断的 mRNA 片段在反转录过程中迅速被降解,这样,已经反转录并插入到基因组的小片段就成为了短假基因.假基因遍布于低等细菌到高等生物的多个基因 组[7-8, 14-21]. 据估计大肠杆菌基因组可能有上百个假基因[14-15],人和小鼠基因组各有约 2 万个假基因[7-8, 19].
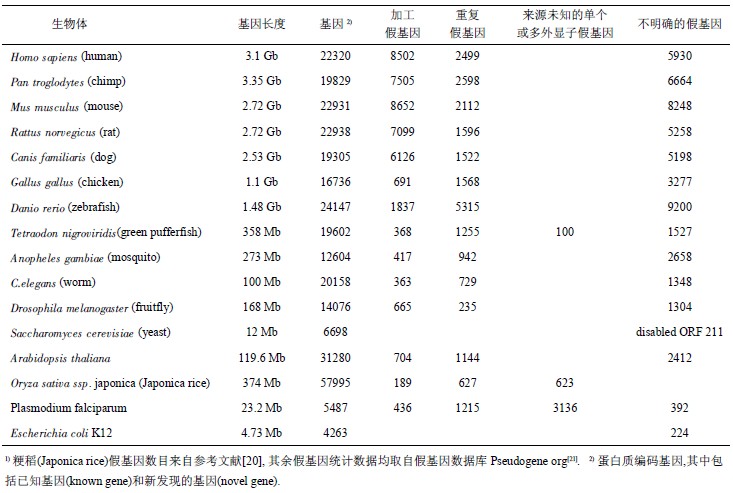
目前,除了人类基因组以外,对果蝇 、线虫、小鼠、大鼠、酵母和水稻等基 因组的全基因组范围的假基因识别工作也已完成,假基因数量信息列于表 2. 值得一提的是,我们所看到的只是在目前种群中幸存下来的假基因,而不少假基因在过去可能早已被淘汰[7-8]. 淘汰是指假 基因在选择压力或突 变的作用下退出基因组舞台或变得不可识别.
2.2 假基因进化研究
假基因是丧失蛋白质编码能力的基因拷贝,是基因组 DNA 进化的“遗迹”,它从分子水平上记 录了基因组序列数百万年的进化路线,为基因组进化研究提供了理想的材料. 假基因的进化研究包括假基因进化年代(或距离)的估计、进化特征、进化 过程中所受选择压力分析等几方面[7, 30-31]. 从假基因与其祖先功能基因的比较中可以获得和判断假基因产生的时间[7]、突变(碱基置换、插入 / 缺失突变)谱[30]、演化方向[31]等. 研究表明,目前所识别出的绝 大 部 分 人类 加 工假 基 因是 哺乳动 物 辐射 (mammalian radiation)之后(即约 7500 万年前啮齿类和 灵长类分歧之后)产生的[7]. 随着进化 进程的推进,假 基 因在组 分 ( 包 括 GC 含量 、 假 想 密 码 子和 氨 基 酸 组 分 ) 上 呈 现 出向 基因 间 序列 漂 移 的 趋 势[31]. 通过人类加工假基因的研究,人们还发现假 基因序列中发生的碱基置换突变对其紧邻碱基存在依赖性[30].
加工假基因在基因组中的总数量受到三种因素的约束:转座活动(transposition activity)的频率、假 基因受到的选择压力和基因组背景突变速率. 加工假基因是在相关酶的作用下反转录转座而产生的,因此转座活动越频繁, 加工假基因数量越多 . 然而,并不是所有产生的假基因都能在基因组中长期存留下来,这取决于基因组背景突变速率和根据假 基因的利弊对其施加的选择压力. 基因组背景突变速率影响假基因丰度的典型例子是果蝇基因组[18],果蝇基因组较频繁发生的点突变和缺失突变[32],导 致假基因迅速退化并不可辨认.选择压力是影响假基因丰度的重要因素. 例如,若一个假基因在染色体上的插入是个选择有害的突变,则进化过程中这个假基因受到较大的负选择,从而很快地退出基因 组舞台. 在进化过程中,假基因所受的选择压力可从其同义突变率和非同义突变率的比值中判断. 如果假基因一产生就失去了活性,那么其同义位点和 非同义位点应该具有相同的趋异度,因为只有翻译对基因置换位点产生选择压力时,这两种位点上的趋异度才会不同.但实际上,非同义置换率要比同 义置换率小,这有可能是方法上的缺陷造成的[7],也 有 可 能是 反映 淘汰 非 同 义 置 换 突变 的 选择 作 用[2]. 假基因即使受到这种选择压力,与真基因相比这种选择压力还是比较微弱的[7].
不同的基因对应的加工假基因数量也有很大差 异,这种不均匀性有以下几方面的原因:首先,种系细胞、受精卵或早期胚胎细胞是产生加工假基因的决定性阶段,因为只有在那些早期发育阶段产生 的假基因才可能遗传,并在后代基因 组中固定下来 [7]. 因 此 ,在 早 期 发 育阶 段 具 有 较高 转 录 活 性(mRNA 丰度较高)的基因更易发生反转录转座并产生加工假基因. 由人类核糖体蛋白基因产生的高丰度的加工假基因 验证了 这种 假说 [7, 33]. 无独 有偶 , 在人和小鼠基因组中不仅富含核糖体蛋白返座假基因,而且还富含核糖体蛋白返座基因[34]. 除了转录活性以外,基因的 GC 含量、mRNA 的长度和稳定性也与加工假基因的丰度有关[7, 35]. GC 含量较低、长度较短的基因,对应的加工假基因丰度也高,前 者可能与加工假基因的退化速率有关,而后者可能 涉及到反转录转座效率与 mRNA 转录本长度间的关系[7, 33].
2.3 假基因分布
揭示加工假基因分布中所隐藏的进化压力对加工假基因群体动态研究和基因组进化研究具有重要意义. 在长期的进化过程中,不同的生物如何适应 选择压力获得生存竞争的优势,是基因组进化研究探讨的主要问题.假基因的演化过程对于此类问题的研究非常重要,因为它们提供了基因组 DNA 在进化选择压力下怎样改变的“痕迹”. “痕迹”不仅 在 DNA 分子 水平 上 反映 出 来 , 在假 基 因的数 量、分布中也有所体现.研究表明,加工假基因在染色体上的分布与染色体局域 GC 含量[7]、DNA 更换 率 (turnover rate) [18]、 转 座机 制 的 插 入 偏好 性 [5]、 染色体结构稳定性[36] 、卵子发生时间长短[37]以及功能元件的分布和染色体重组频率[38-39]都有联系.
从加工假基因的产生机制来看,它在染色体上 分布应该与转座机制的插入偏好性紧密相关. 加工 假基因和 Alu 重复序列是非自主转座子,灵长类基因组中这两种元件被认为是由 L1 重复序列编码酶的作用下进行转座的[4-5]. 研究表明 L1 重复序列转 座时通常以富含 AT 的区域作为靶位点[5]. 据此可以推测三者在染色体上应该 具有相似的、在富含AT 区分布的偏好性. 然而事实表明三者在染色体上的分布偏好性各不相同:L1 偏好分布在 AT 富含区,Alu 偏好分布在 GC 富含区,加工假基因则偏好分布在 GC 含量中等区[7]. 这种差别可能是在转座后的进化过程中出现的. 一种可能性是 Alu、L1 和加工假基因在染色体上的分布可能与序列组分有关的负选择机制有关[7, 36]. 具体来说,这些序 列在染色体上与自身核苷酸 组分相似区域更加稳定,否则在自然选择压力下发生快速突变,突变的方向是序列组分在进化过程中逐步趋向基因间序列或其侧翼序列(flanking sequences), 从而 变得不能用相似性搜索方法识别这些 序列. 这可能就是高GC 含量的 Alu 序列偏好分布在高 GC 含量区,高AT 含量的 L1 序列偏好分布在高 AT 含量区,而加工假基因则偏好分布在中等 GC 含量区的原因. 另一种可能性是 L1、Alu 和加 工假基因在染色体上 的分布可能会随时间的推移向功能密集区(如基因密集区,同样是高 GC 含量区)偏移,而这种偏移是异位重组事件在基因分布较少的区域相对频繁地发生并导致序列删除的被动结果[40]. 加工假基因密 度 与基因 密度 正相 关 [38-39], 这可 能是 由较 容 易 发 生在基因稀少区的异位重组事件对假 基因的删除所致.
卵子发生时间的长短(length of oogenesis)可能 与加工假基因的丰度有关[37]. 卵母细胞减数分裂过程中形成的灯刷染色体的侧环是转录活性较高的区 域,而且 mRNA 的丰度又与加工假基因的产生数量成正比,因此加工假基因在基因组中的丰度应与灯刷染色体的持续时间成正比.因为灯刷染色体是对卵母细胞而言的,所以加工假基因在某一染色体上的丰度与该染色体的宿主性别有关[37]. 从生命的延续过程来看,X 染色体、常染色体和 Y 染色体在女性和男性细胞中存在的时间比值分别是 2/1、1/1 和 0/1[41]. 可见,X 染色体在女性细胞中存在的时间较长,这很可能是导致加工假基因较多地分布在 X 染色体上的原因[37]. 加工假基因在人 Y 染色 体上非常少,其中一些可能是通过假常染色体区的同源重组过程从 X 染色体转移过来的[37]. 研究还发现,人类基因组中保守的、转录的、加工假基因富含于 X 染色体上[42],这可能与哺乳动物 X 染色体上基因的双向返座运输(trafficking)机制有关[43],即X 染色体向其他染色体输送返座基因和从其他染色 体接受返座基因的频率均高于常染色体. 染色体重组频率的高低也会影响加工假基因在染色体上的分 布[38-39]. 从转座元件(包括 DNA 转座子和反转录转座子)分布与重组率的相关性[44-45]得到启发,我们发 现人类加工假基因密度与重组率负相关[38]. 通过进一步研究,我们证实加工假基因在低重组区的分布偏好性是由高重组区同源加工假基因之间异位重组事件的负选择作用造成的[39]. 研究还表明较长的加工假基因更加偏好分布于低重组区[39].
2.4 假基因功能
在假基因研究中,假基因功能始终是人们关注的一大焦点. 假基因本身并没有明显的选择优势, 因此曾被认为是一种“分子寄生物”. 然而随着研究的深入,人们发现在某些情况下,假基因在基因表达调控、基因组进化等方面发挥着一些重要作 用[2, 46-47]. 发现假基因功能的典型例子 来自小鼠基因组:Hirotsune 等[46]在研究随机插入到小鼠基因组 中的 果 蝇 基因表达 时 , 偶 然 发现 可 转 录 的Makorin1-p1 假基因对其同源编码基因的表达具有调节作用.这可能是因为转录的假基因利用链互补性与其同源编码基因的 mRNA 相结合,通过这种反 义 干扰 (antisense-interference) 作用 影响 基 因表 达. 但也有研究表明,Makorin1-p1 假基因是否具 有上述功能还是有争议的问题[48]. 研究还发现,在小鼠卵母细胞中转录的假基因能够进一步产生小干扰 RNA(siRNA),从而影响其同源基因的表达[49-50].转 录 的一 氧化 氮 合酶 (NOS)假 基因 同样 是 以 反 义RNA 的形式影响神经元型一氧化氮合酶的表达[51].最近,Guo 等[52]发现水稻基因组中转录的假基因产生长度约为 24 bp 的小 RNA,这些小 RNA 可能会 与其起源基因的 RNA 转录本或与邻近的同源假基因的 RNA 转录本相结合,从而影响基因的表达.这些都表明,转录的假基因可能是在 RNA 水平上发挥其基因表达调控作用.
可转录的假基因还可以作为小分子核仁 RNA (snoRNA)的载体而起到保护 snoRNA 的作用[53-54].真核 细 胞 中, 无 义 介 导 的 mRNA 降解 机 制(nonsense-mediated mRNA decay, NMD) 会 使 提前 出现终止密码子的 mRNA 降解掉,以免表达出有害的不完整蛋白.Mitrovich 等[53]在线虫基因组中发现,含有终止密码子的假基因转录本受到 NMD 的作用. 他们还发现有一个 snoRNA 是从假基因内含子转录而来的. 由于 NMD 只作用于成熟的 mRNA上,包含在内含子里的 snoRNA 会躲过 NMD 的降 解. 从 这个 意义 上来 讲,假 基因是 snoRNA 的载体,且起到了保护 snoRNA 的作用.
寻找假基因转录证据的研究潮流证实了很多基 因组(如人、小鼠、植物和酵母等基因组)中有些假基因确实是转录的[55-59],而且有些转录假基因在不同基因组中具有同源性和保守性[42, 47, 60]. 转录不局 限 于 重 复 假 基 因, 加 工 假基因 也可 以 转 录 [55-56].ENCODE 计划研究表明,人类基因组可能在整个基因组范围内广泛转录[56],其中功能未知的转录事 件偏好发生于包括假基因在内的传统“垃圾 DNA”区[61]. 转录并不意味着肯定有功能. 序列保守性是推 断 基 因 组序列 有 无 功 能的重要 指 标 . Svensson等[47]通过比较基因组学方法发现,有 30 个可转录的假基因在人、黑猩猩和小鼠基因组之间是保守的. 假基因在进化过程中的高保守性 及高同义突 变率均意味着这些假基因可能承受着一定的功能约束[2]. Khachane 等[42]发现:剔除同一数据库和不同数据库中相互重叠的冗余数据后,人类基因组约含有 15 000 个假基因,其中转录 假基因约 占 11.5% (1750 个);只有极少部分(约 3%~ 6%)的假基因在反义 方向 (antisense direction)上 转录, 这可 能是 阻 止假基因转录本与其同源基因转录本之间互补杂交的选择压力所致;相比人类基因组,小鼠假基因中可转录假基因所占的比例较少( < 2%). 通过同源性搜索结合共线性(synteny)分析发现,一些人类转录假基因在进化过程中保持保守,一半以上的人类转录假基因在人和猕猴之间保 持共线保守,而只有3%的转录假基因在人和小鼠之间保持保守,表明多数人类转录假基因是灵长类特异的,在不同物种之间,转录假基因的保守性显著强于其侧翼序列,意 味 着 转 录 假 基因 经 受 着 显 著 的 选择 压力 [42].Balasubramanian 等[60]通过对人、黑猩猩、小鼠和大鼠基因组的分析发现,核糖体蛋白加工假基因的数量与其对应的 mRNA 的丰度并无关联,这一点与之前的发现[33]相矛盾,共线区的假基因在人和黑猩 猩之间表现出较高的保守性,但灵长类和啮齿类之间没有保守性,共线区假基因的序列突变率低于侧 翼基因间序列,与表达序列标签 (EST) 进行 比对 ,发现有两个在人和小鼠之间保持保守的假基因可以转录,并且其中之一 RPS27 可能会表达成蛋白质, 这一点与人类基因组包含 80 个(而非 79 个)核糖体 蛋白基因的假说相吻合.
除了与基因表达调控有关以外,假基因在基因组进化过程中可能扮演着重要角色. 假基因是不是 大自然事先在基因组中储备好的用于发展新功能的素材呢?现在普遍认为产生大量加工假基因的返座作用作为一种进化的动力,通过促进 序列连续性复制、散置和重组,保持着真核生物 基因组流动性[5, 62]. 假基因、转座子、微卫星等序列中发生的看起来无用的遗传变异可能为物种进化的正选择、负选择及中性突变提供了丰富的材料[5, 62]. 有时假 基因可以重获新生,丰富生物细胞的功能多样性.从这种意义上讲,假基因并不是进化的死末端,而是基因组创建更好的遗传信息表达方 式的备用素材,是产生新基因、扩增新功能的又一源泉. 研究 还指出,假基因介导 的基因转 换 (gene conversion) 可能有助于提高生物体的免疫力,促进适应性进化[63-64]. 除了积极的一面以外,假基因还有一些负面影响. 例如,假基因也有可能通过基因转换把自身的缺陷部位传递给功能基因或与非等位位点上的同源基因发生重组,从而导致疾病的发生[22, 65]. 另外,由于同源重组依赖序列同源性,假基因的插入突变可能会导致重组频率的改变[2],其自然选择层面上的利弊关系还不是很清楚.
随着“基因”的重新定义[66-67]和假基因潜在功 能的发现,有人指出重新定义“假基因”已迫在眉睫.据新的定义,假基因是源于功能基因,但不能够表达成与原有 基因相同类型的产物( 如蛋 白质 、tRNA,rRNA 等)的基因组序列[68].
纵观假基因功能的文献报道,我们认为少数假 基因是有功能的,而大多数假基因可能没有功能.
3 总结与展望
本文从假基因的起源与序列特征、假基因的识 别、假基因在染色体上的分布及其分子进化规律, 以及假基因功能等几个方面较为全面地介绍了该领域最新研究进展.假基因的识别是基础性工作. 在假基因的识别中已不再把“无功能”作为假基因的必要条件.这是合乎情理的假设,因为你可以证明一条序列有功能,却很难证明一条序列肯定没有功 能.基于目前的假基因识别程序,我们可以非常便捷地从基因组中搜索出自己所需的、不同特征的假基因. 假基因在基因组进化研究中的意义是不言而喻的,它是迄今为止人们发现的最令人振奋的记录 基因组进化脚印的分子化石 . 当前,“假基因功 能”在该研究领域中备受瞩目.学术界已经从实验上发现了 有 些 假 基 因 在 基 因 表 达 调控 网络 中 以RNA 干扰的方式起作用,然而这种调控作用是凤 毛麟角还是司空见惯还有待进一步的实验和理论工作去检验. 显然,建立“假 基因对基因的表达调 控”理论还有一段路要走.
参考文献
[1] Wickelgren I. Spinning junk into gold. Science, 2003, 300 (5626): 1646-1649
[2] Balakirev E S, Ayala F J. Pseudogenes: are they “ junk” or functional DNA?. Annu Rev Genet, 2003, 37: 123-151
[3] Vanin E F. Processed pseudogenes: characteristics and evolution. Annu Rev Genet, 1985, 19: 253-272
[4] Esnault C, Maestre J, Heidmann T. Human LINE retrotransposons generate processed pseudogenes. Nat Genet, 2000, 24(4): 363-367
[5] Kazazian H H. Mobile elements: drivers of genome evolution. Science, 2004, 303(5664): 1626-1632
[6] Li W H, Gojobori T, Nei M. Pseudogenes as a paradigm of neutral evolution. Nature, 1981, 292(5820): 237-239
[7] Zhang Z, Harrison P M, Liu Y, et al. Millions of years of evolution preserved: a comprehensive catalog of the processed pseudogenes in the human genome. Genome Res, 2003, 13(12): 2541-2558
[8] Torrents D, Suyama M, Zdobnov E, et al. A genome-wide survey of human pseudogenes. Genome Res, 2003, 13(12): 2559-2567
[9] Liu Y, Harrison P M, Kunin V, e t al. Comprehensive analysis of pseudogenes in prokaryotes: widespread gene decay and failure of putative horizontally transferred genes. Genome Biol, 2004, 5 (9): R64
[10] Zhang Z, Gerstein M. Identification and characterization of over 100 mitochondrial ribosomal protein pseudogenes in the human genome. Genomics, 2003, 81(5): 468-480
[11] Lopez J V, Yuhki N, Masuda R, e t al. Numt, a recent transfer and tandem amplification of mitochondrial DNA to the nuclear genome of the domestic cat. J Mol Evol, 1994, 39(5): 174-190
[12] Nugent J M, Palmer J D. RNA-mediated transfer of the gene COXII from the mitochondrion to the nucleus during flowering plant evolution. Cell, 1991, 66(3): 473-481
[13] Woischinik M, Moraes C T. Pattern of organization of human mitochondrial pseudogenes in the nuclear genome. Genome Res,2002, 12(6): 885-893
[14] Lerat E, Ochman H. Recognizing the pseudogenes in bacterial genomes. Nucleic Acids Res, 2005, 33(10): 3125-3132
[15] Ochman H, Davalos L M. The nature and dynamics of bacterial genomes. Science, 2006, 311(5768): 1730-1733
[16] Harrison P M, Kumar A, Lan N, e t al. A small reservoir of disabled ORFs in the yeast genome and its implications for the dynamics of proteome evolution. J Mol Biol, 2002, 316(3): 409-419
[17] Harrison P M, Echols N, Gerstein M. Digging for dead genes: an analysis of the characteristics of the pseudogene population in the Cae norhabditis e le gans genome. Nucleic Acids Res, 2001, 29 (3): 818-830
[18] Harrison P M, Milburn D, Zhang Z, e t al. Identification of pseudogenes in the Drosop hila me lanogaster genome. Nucleic Acids Res, 2003, 31(3): 1033-1037
[19] Zhang Z, Carriero N, Gerstein M. Comparative analysis of processed pseudogenes in the mouse and human genomes. Trends Genet, 2004, 20(2): 62-67
[20] Thibaud-Nissen F, Ouyang S, Buell C R. Identification and characterization of pseudogenes in the rice gene complement. BMC Genomics, 2009, 10: 317
[21] Karro J E, Yan Y, Zheng D, e t al. Pseudogene.org: a comprehensive database and comparison platform for pseudogene annotation. Nucleic Acids Res, 2007, 35(Database Issue): D55-D60
[22] Bischof J M, Chiang A P, Scheetz T E, e t al. Genome-wide identification of pseudogenes capable of disease-causing gene conversion. Human Mutation, 2006, 27(6): 545-552
[23] Khelifi A, Duret L, Mouchiroud D. HOPPSIGEN: a database of human and mouse processed pseudogenes. Nucleic Acids Res, 2005, 33(Database Issue): D59-D66
[24] Zhang Z, Carriero N, Zheng D, e t al. PseudoPipe: an automated pseudogene identification pipeline. Bioinformatics, 2006, 22 (12): 1437-1439
[25] Ortutay C, Vihinen M. PseudoGeneQuest-Service for identification of different pseudogene types in the human genome. BMC Bioinformatics, 2008, 9: 299
[26] van Baren M J, Brent M R. Iterative gene prediction and pseudogene removal improves genome annotation. Genome Res, 2006, 16(5): 678-685
[27] Zheng D, Gerstein M. A computational approach for identifying pseudogenes in the ENCODE regions. Genome Biol, 2006, 7 (Suppl 1): S13
[28] Zheng D, Frankish A, Baertsch R, e t al. Pseudogenes in the ENCODE regions: Consensus annotation, analysis of transcription, and evolution. Genome Res, 2007, 17(6): 839-851
[29] Terai G, Yoshizawa A, Okida H, et al. Discovery of short pseudogenes derived from messenger RNAs. Nucleic Acids Res, 2010, 38(4): 1163-1171
[30] Zhang Z, Gerstein M. Patterns of nucleotide substitution, insertion and deletion in the human genome inferred from pseudogenes. Nucleic Acids Res, 2003, 31(18): 5338-5348
[31] Echols N, Harrison P M, Balasubramanian S, et al. Comprehensive analysis of amino acid and nucleotide composition in eukaryotic genomes comparing genes and pseudogenes. Nucleic Acids Res, 2002, 30(11): 2515-2523
[32] Petrov D A, Hartl D L. High rate of DNA loss in the Drosophila me lanogaster and Drosophila virilis species groups. Mol Biol Evol, 1998, 15(3): 293-302
[33] Zhang Z, Harrison P M, Gerstein M. Identification and analysis of over 2000 ribosomal protein pseudogenes in the human genome. Genome Res, 2002, 12(10): 1466-1482
[34] Yu Z, Morais D, Ivanga M, e t al. Analysis of the role of retrotransposition in gene evolution in Vertebrates. BMC Bioinformatics, 2007, 8: 308
[35] Pavlicek A, Gentles A J, Paces J, e t al. Retroposition of processed pseudogenes: the impact of RNA stability and translational control. Trends Genet, 2006, 22(2): 69-73
[36] Pavlicek A, Jabbari K, Paces J, et al. Similar integration but different stability of Alus and LINEs in the human genome. Gene, 2001, 276(1-2): 39-45
[37] Drouin G. Processed pseudogenes are more abundant in human and mouse X chromosomes than in autosomes. Mol Biol Evol, 2006, 23(9): 1652-1655
[38] 刘国庆, 李 宏. 人类基因组中加工假基因分布与重组率和基因 密度的关系. 生物物理学报, 2008, 24(5): 371-378 Liu G Q, Li H. Acta Biophys Sin, 2008, 24(5): 371-378
[39] Liu G, Li H, Cai L. Processed pseudogenes are located preferentially in regions of low recombination rates in the human genome. J Evol Biol, 2010, 23(5): 1107-1115
[40] Abrusan G, Krambeck H J. The distribution of L1 and Alu retroelements in relation to GC content on human sex chromosomes is consistent with the ectopic recombination model. J Mol Evol, 2006, 63(4): 484-492
[41] Miyata T, Hayashida H, Kuma K, et al. Male-driven molecular evolution: a model and nucleotide sequence analysis. Cold Spring Harbor Symp Quant Biol, 1987, 52: 863-867
[42] Khachane A N, Harrison P M. Assessing the genomic evidence for conserved transcribed pseudogenes under selection. BMC Genomics, 2009, 10: 435
[43] Emerson J J, Kaessmann H, Betran E, e t al. Extensive gene traffic on the mammalian X chromosome. Science, 2004, 303(5657): 537- 540
[44] Jensen-Seaman M I, Furey T S, Payseur B A, e t al. Comparative recombination rates in the rat, mouse, and human genomes. Genome Res, 2004, 14(4): 528-538
[45] Hua-Van A, Rouzic A L, Maisonhaute C, et al. Abundance, distribution and dynamics of retrotransposable elements and transposons: similarities and differences. Cytogenet Genome Res, 2005, 110(1-4): 426-440
[46] Hirotsune S, Yoshida N, Chen A, et al. An expressed pseudogene regulates the messenger-RNA stability of its homologous coding gene. Nature, 2003, 423(6935): 91-96
[47] Svensson O, Arvestad L, Lagergren J. Genome-wide survey for biologically functional pseudogenes. PLOS Comput Biol, 2006, 2(5): 358-369
[48] Gray T A, Wilson A, Fortin P J, e t al. The putatively functional Mkrn1-p1 pseudogene is neither expressed nor imprinted, nor does it regulate its source gene in trans. Proc Natl Acad Sci USA, 2006, 103(32): 12039-12044
[49] Tam O H, Aravin A A, Stein P, e t al. Pseudogene-derived small interfering RNAs regulate gene expression in mouse oocytes. Nature, 2008, 453(7194): 534-538
[50] Watanabe T, Totoki Y, Toyoda A, e t al. Endogenous siRNAs from naturally formed dsRNAs regulate transcripts in mouse oocytes. Nature, 2008, 453(7194): 539-543
[51] Korneev S A, Park J H, O'Shea M. Neuronal expression of neural nitric oxide synthase (nNOS) protein is suppressed by an antisense RNA transcribed from an NOS pseudogene. J Neurosci, 1999, 19(18): 7711-7720
[52] Guo X, Zhang Z, Gerstein M, e t al. Small RNAs Originated from pseudogenes: cis- or trans-acting?. PLoS Comput Biol, 2009, 5 (7): e1000449
[53] Mitrovich Q M, Anderson P. mRNA Surveillance of expressed pseudogenes in C. e le gans . Curr Biol, 2005, 15(10): 963-967
[54] Weischenfeldt J, Lykke-Andersen J, Porse B. Messenger RNA surveillance: Neutralizing natural nonsense. Curr Biol, 2005, 15(14): R559-R562
[55] Harrison P M, Zheng D, Zhang Z, e t al. Transcribed processed pseudogenes in the human genome: an intermediate form of expressed retrosequence lacking protein-coding ability. Nucleic Acids Res, 2005, 33(8): 2374-2383
[56] The ENCODE Project Consortium. Identification and analysis of functional elements in 1% of the human genome by the ENCODE pilot project. Nature, 2007, 447(7146): 799-816
[57] Yano Y. Saito R, Yoshida N, et al. A new role for expressed pseudogenes as ncRNA: regulation of mRNA stability of its homologous coding gene. J Mol Med, 2004, 82(7): 414-422
[58] Yamada K, Lim J, Dale J M, et al. Empirical analysis of transcriptional activity in the A rab idop sis genome. Science, 2003, 302(5646): 842-846
[59] Harrison P M, Kumar A, Lan N, et al. A small reservoir of disabled ORFs in the yeast genome and its implications for the dynamics of proteome evolution. J Mol Biol, 2002, 316(3): 409-419
[60] Balasubramanian S, Zheng D, Liu Y J, e t al. Comparative analysis of processed ribosomal protein pseudogenes in four mammalian genomes. Genome Biol, 2009, 10(1): R2
[61] Willingham A T, Gingeras T R. TUF love for‘junk’ DNA. Cell, 2006, 125(7): 1215-1220
[62] Weiner A M, Deininger P L, Efstratiadis A. Nonviral retroposons: genes, pseudogenes, and transposable elements generated by the reverse flow of genetic information. Annu Rev Biochem, 1986, 55: 631-661
[63] Hansen T F, Carter A J, Chiu C H. Gene conversion may aid adaptive peak shifts. J Theor Biol, 2000, 207(4): 495-511
[64] Hayakawa T, Angata T, Lewis A L, et al. human-specific gene in microglia. Science, 2005, 309(5741): 1693
[65] Roesler J, Curnutte J T, Rae J, e t al. Recombination events between the p47-p hox gene and its highly homologous pseudogenes are the main cause of autosomal recessive chronic granulomatous disease. Blood, 2000, 95(6): 2150-2156
[66] Pearson H. Genetics: what is a gene?. Nature, 2006, 441 (7092): 398-401
[67] Gerstein M, Bruce C, Rozowsky J S, et al. What is a gene, post-ENCODE? History and updated definition. Genome Res, 2007, 17(6): 669-681
[68] Zheng D, Gerstein M. The ambiguous boundary between genes and pseudogenes: the dead rise up, or do they?. Trends Genet, 2007, 23(5): 219-224




1F
很好
2F
请教一下怎么下载这里面的文章,谢谢