miRNA研究现在是如火中天了,不少同学现在也开始从事这方面研究。有些同学常常会迷惑,因为不知道到底需要做哪些研究。下面简单和大家分享一下miRNA测序后分析的主要内容有哪些。
一、基因注释(推荐)
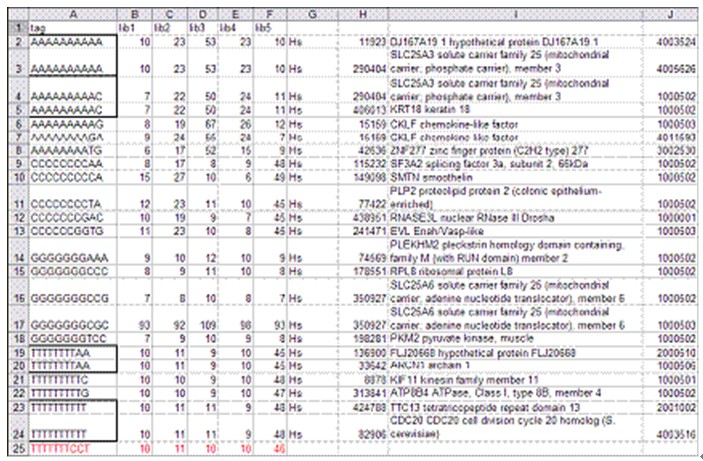
我们将测序结果进行比对和分析,确定tag代表的基因,用于后续分析。
二、 差异基因筛选(推荐)
对样品之间进行差异基因筛选。筛选到表达有差异变化的基因。然后对变化基因的趋势进行归类,以方便分析和描述。实例如下图:
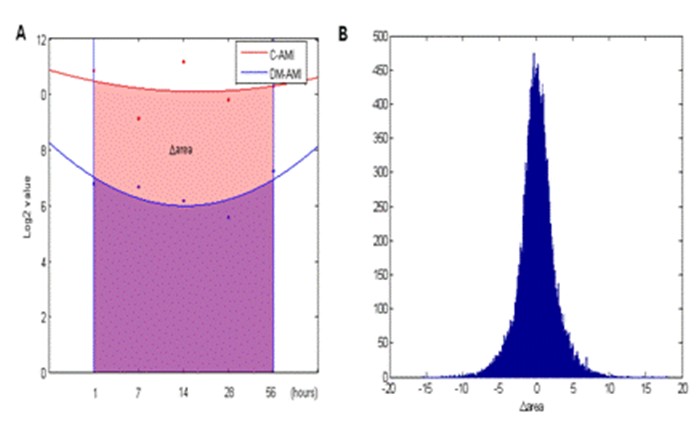
三、样品之间的比较分析
我们将通过曲线拟合的方法,寻找样品之间趋势差异最大的一些基因:
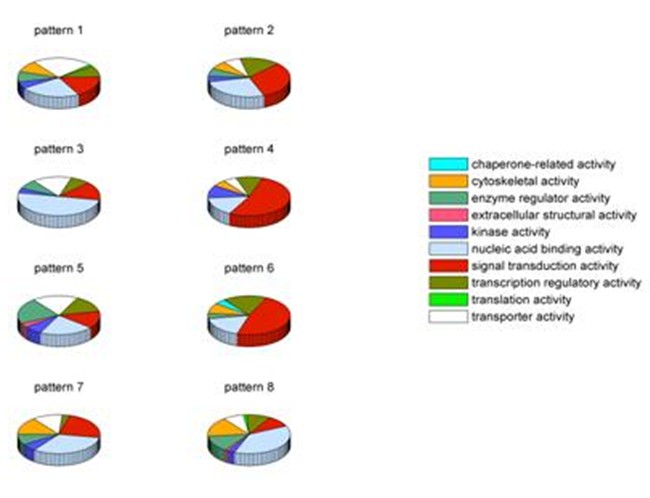
四、GO(gene ontology)分析(推荐)
对于每一种表达趋势的基因,选择性的进gene ontology:功能分析. 对差异表达的所有基因向gene ontology数据库的各节点映射。计算每个节点的基因数目,并结合整个数据库的基因作为背景分部,对于每个节点,得到一个2x2的表格,使用超几何分布检验基因在每个GO节点的富集或贫乏程度。
五、pathway分析(推荐)
与GO分析类似,我们同样将差异基因使用GenMAPP v2.1向KEGG pathway和BioCarta数据库映射。通过统计方案,找到统计上最有意义的pathway。
六、Knowledge-driven Network分析(推荐)
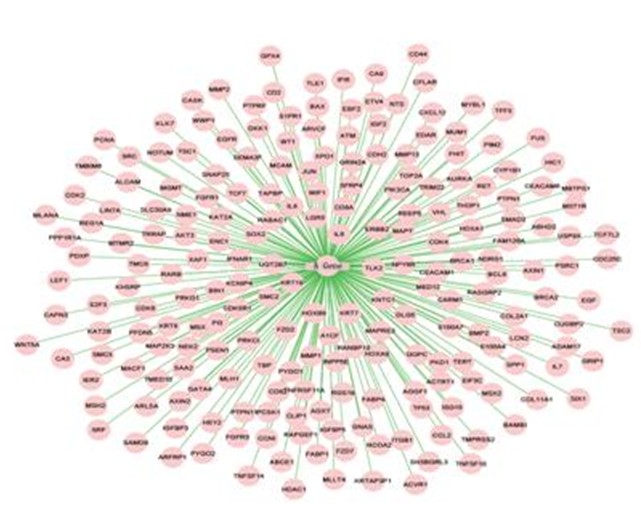
通过整合PubMed text mining,同源预测,基因neighbor,蛋白-蛋白相互作用,基因融合等数据,建立一个all differentially expressed genes in a single plot的调控网络。这是一种已有知识驱动的网络构建.。
七、转录因子分析(推荐)
利用相关的转录因子(TF)数据库,将差异基因的启动子(基因第一个外显子上游1000bp)提取出来。我们使用HsPD 提供的启动子序列。转录因子数据库使用TRANSFAC 7.0 public。转录因子结合位点预测使用pwmatch 程序,对每个转录因子分析其在上调基因和下调基因的分布情况,利用chi-square test 寻找有差异的转录因子。
八、反义转录本的寻找
基本流程:
九、新转录本的寻找:
基本方法:同八
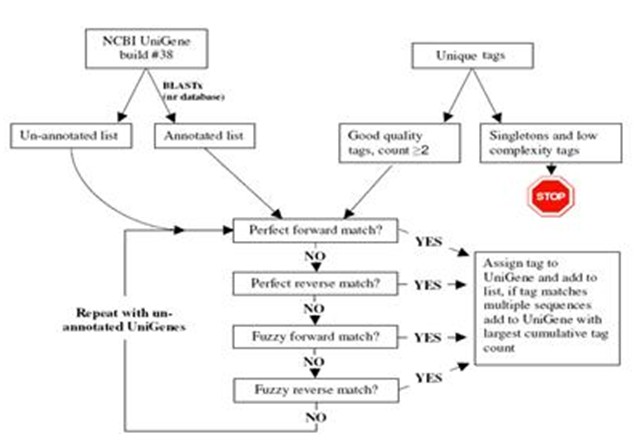
我们对无法注释的tag(un-annotated list)进行基因组定位,并提取上下文序列。利用同源预测或者从头预测程序进行基因预测。一般来讲,同源预测的可靠性较高。而从头预测算法假阳性较高。
资料来源:http://wenku.baidu.com/view/2554a1252f60ddccda38a009.html (上海敏芯信息科技有限公司 mRNA测序结果分析方案书)




1F
明明是mRNA分析,抄百度文库还抄错
来自外部的引用